21.1: Redes Regulatorias- Inferencia, Análisis, Aplicación
- Page ID
- 54989
Los sistemas vivos están compuestos por múltiples capas que codifican información sobre el sistema. Las capas primarias son:
1. Epigenoma: Definido por la configuración de la cromatina. La estructura de la cromatina se basa en la forma en que las histonas organizan el ADN. El ADN se divide en regiones libres de nucleosomas y nucleosomas, formando su forma final e influyendo en la expresión génica.
- Genoma: Incluye ADN codificante y no codificante. Los genes definidos por ADN codificante se utilizan para construir ARN, y los elementos reguladores de la CIS regulan la expresión de estos genes.
- Los ARN del transcriptoma (por ejemplo, ARNm, miARN, ARNc, ARNpi) se transcriben a partir del ADN. Tienen funciones reguladoras y fabrican proteínas.
- Proteoma Compuesto por proteínas. Esto incluye factores de transcripción, proteínas de señalización y enzimas metabólicas.
Las interacciones entre estos componentes son todas diferentes, pero entenderlas puede poner partes particulares del sistema en el contexto del todo. Para descubrir relaciones e interacciones dentro y entre capas, podemos usar redes.
Presentamos Redes Biológicas
Las redes biológicas están compuestas de la siguiente manera: Red
Reguladora — conjunto de interacciones reguladoras en un organismo.
- Los nodos son reguladores (por ejemplo, factores de transcripción) y dianas asociadas.
- Los bordes corresponden a la interacción regulatoria, dirigida desde el factor regulatorio hasta su objetivo. Se firman según el eect positivo o negativo y se ponderan según la fuerza de la reacción.
Metabolic Net — conecta los procesos metabólicos. Hay cierta flexibilidad en la representación, pero un ejemplo es una gráfica que muestra productos metabólicos compartidos entre enzimas.
- Los nodos son enzimas.
- Los bordes corresponden a reacciones reguladoras, y se ponderan de acuerdo con la fuerza de la reacción.
Red de Señalización — representa rutas de señales biológicas.
- Los nodos son proteínas llamadas receptores de señalización.
- Los bordes se transmiten y reciben señales biológicas, dirigidas del transmisor al receptor.
Protein Net: muestra interacciones físicas entre proteínas.
• Los nodos son proteínas individuales.
• Los bordes son interacciones físicas entre proteínas.
Red de coexpresión: describe las funciones de coexpresión entre genes. Muy general; representa redes de interacción funcionales más que físicas, a diferencia de los otros tipos de redes. Potente herramienta en el análisis computacional de datos biológicos.
• Los nodos son genes individuales.
• Los bordes son relaciones de coexpresión.
Hoy, nos centraremos exclusivamente en las redes regulatorias. Las redes reguladoras controlan la expresión génica específica del contexto y, por lo tanto, tienen un gran control sobre el desarrollo. Vale la pena estudiarlos porque son propensos al mal funcionamiento y a causar enfermedades.
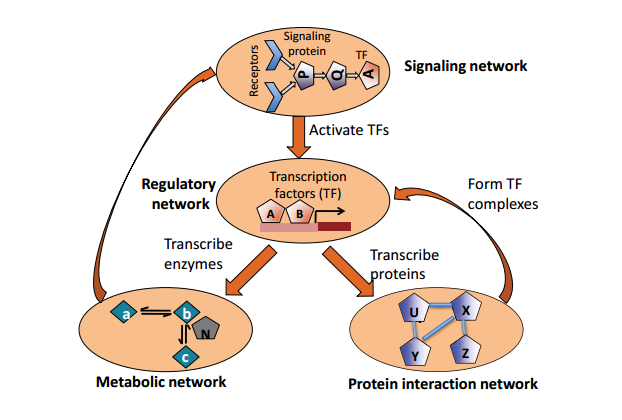
Licencia Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
a) Interacciones entre redes biológicas.
Interacciones entre redes biológicas
Las redes biológicas individuales (es decir, capas) pueden considerarse nodos en una red más grande que representa todo el sistema biológico. Podemos, por ejemplo, tener una red de señalización detectando el entorno que gobierna la expresión de los factores de transcripción. En este ejemplo, la red mostraría que los TFs gobiernan la expresión de proteínas, las proteínas pueden desempeñar papeles como enzimas en las vías metabólicas, y así sucesivamente.
Las rutas generales de intercambio de información entre estas redes se muestran en la figura 21.4.
Estudiando Redes Regulatorias
En general, las redes se utilizan para representar dependencias entre variables. Las dependencias estructurales se pueden representar por la presencia de un borde entre nodos; como tal, los nodos no conectados son condicionalmente independientes. Probabilísticamente, a los bordes se les puede asignar un “peso” que represente la fuerza o la probabilidad de la interacción. Las redes también pueden verse como matrices, permitiendo operaciones matemáticas. Estos marcos proporcionan una manera efectiva de representar y estudiar sistemas biológicos.
Estas redes son particularmente interesantes de estudiar porque el mal funcionamiento puede tener un gran efecto. Muchas enfermedades son causadas por las reconexiones de las redes regulatorias. Controlan la expresión específica del contexto en el desarrollo. Debido a esto, se pueden usar en biología de sistemas para predecir el desarrollo, el estado celular, el estado del sistema y más. Además, encapsulan gran parte de la diferencia evolutiva entre organismos que son genéticamente similares.
Para describir las redes regulatorias, hay varias preguntas desafiantes que responder.
Identificación de elementos ¿Cuáles son los elementos de una red? En la última conferencia se identificaron elementos constitutivos de redes regulatorias Estos incluyen motivos aguas arriba y sus factores asociados.
Análisis de estructura de red ¿Cómo se conectan los elementos de una red? Dada una red, el análisis de estructura consiste en el examen y caracterización de propiedades importantes. Se puede hacer redes biológicas pero no se restringe a ellas.
Inferencia de red ¿Cómo interactúan los reguladores y activan los genes? Esta es la tarea de identificar los bordes de los genes y caracterizar sus acciones.
Aplicaciones de red ¿Qué podemos hacer con las redes una vez que las tenemos? Las aplicaciones incluyen la función de predicción de genes reguladores y la predicción de los niveles de expresión de genes regulados.
1 Más en la conferencia de epigenética.


