28.4: Flujo de genes en el subcontinente indio
- Page ID
- 54764
La evidencia genética sugiere que las poblaciones modernas del subcontinente indio descendieron de dos poblaciones ancestrales diferentes que se mezclaron hace 4,000 años. Los datos de la matriz SNP se recolectaron de alrededor de 500 personas diferentes de 73 grupos indios con diferentes familias lingüísticas [? ]. Una gráfica de análisis de componentes principales revela que los grupos lingüísticos dravidianos/indoeuropeos y los grupos lingüísticos austroasiáticos se encuentran en dos grupos diferentes, lo que sugiere que tienen diferentes linajes. Dentro de los grupos lingüísticos dravidianos/indoeuropeos, existe un gradiente de relación con los grupos de Eurasia Occidental.
Se utilizó la misma técnica de mosaico utilizada en el estudio de mezcla africano/europeo para estimar la fecha de la mezcla. La población india es una mezcla de un grupo centroasiático-europeo y otro grupo más estrechamente relacionado con la gente de las Islas Andamán. El tamaño del trozo del ADN que pertenece a cada grupo sugiere una mezcla de unas 100 generaciones de antigüedad, o hace 2,000 a 4,000 años. Muchos grupos tienen este patrimonio mixto, pero la mezcla se detiene después de la creación del sistema de castas.
El conocimiento del patrimonio de los genes puede predecir enfermedades. Por ejemplo, una mutación del sur de Asia en la proteína C de unión a miosina provoca un aumento de siete veces en la insuficiencia cardíaca. Muchos grupos étnicos son endogámicos y tienen una diversidad genética baja, lo que resulta en una mayor prevelancia de enfermedades recesivas.
Encuestas anteriores en la India han estudiado aspectos como la variación antropométrica, el ADNmt y el cromosoma Y. El estudio antropométrico analizó diferencias significativas en las características físicas entre grupos separados por geografía y etnia. Los resultados mostraron una variación muy superior a la de Europa. El estudio de ADNmt fue una encuesta de linaje materno y los resultados sugirieron que había un solo árbol indio de tal manera que la edad del linaje podría inferirse por el número de mutaciones. Los datos también mostraron que las poblaciones indias estaban separadas de las poblaciones no indias al menos hace 40 mil años. Finalmente, el estudio del cromosoma Y analizó el linaje paterno y mostró una similitud más reciente con los hombres de Oriente Medio y dependencias de geografía y casta. Estos datos entran en conflicto con los resultados de ADNmt. Una posible ex- planación es que hubo una migración masculina más reciente. De cualquier manera, los estudios genéticos realizados en la India han servido para mostrar su complejidad genética. La alta variación genética, la disimilitud con otras muestras y la diculdad de obtener más muestras llevan a que la India quede fuera de HapMap, el Proyecto 1000 Genomas y el HGDP.
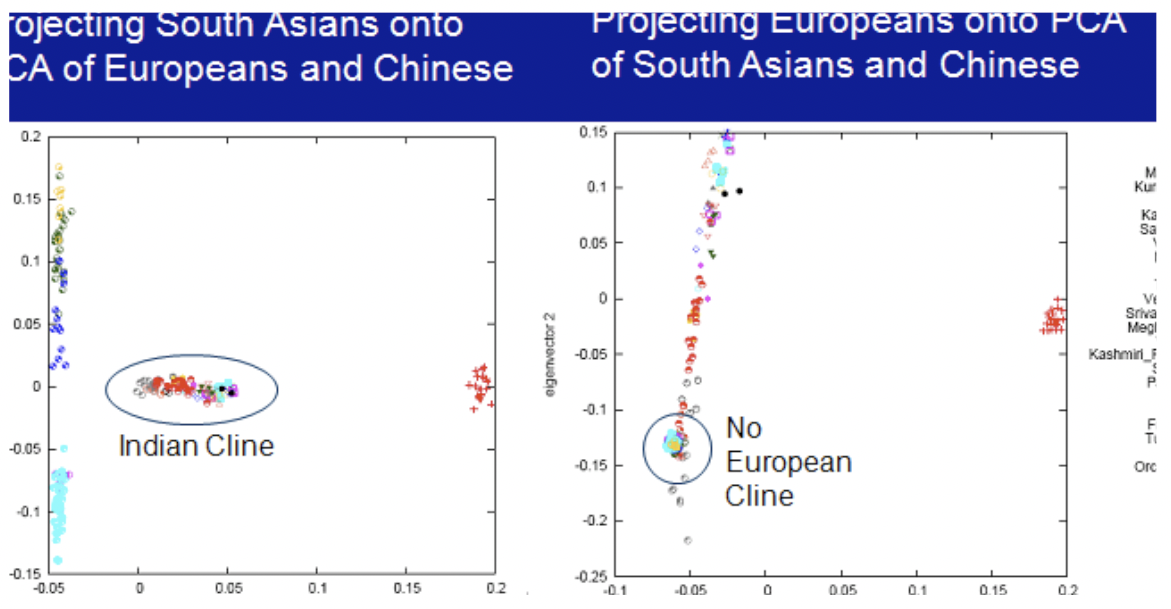
En el estudio de David Reich y colaboradores de la India, se eligieron 25 grupos indios para representar diversas geografías, raíces lingüísticas y etnias. Los datos brutos incluyeron cinco muestras para cada uno de los veinticinco grupos. A pesar de que este número parece pequeño, el número de SNP de cada muestra tiene mucha información. Aproximadamente quinientos mil marcadores fueron genotipados por individuo. Al observar los datos que emergen del estudio, si se usa Análisis de Componentes Principales en datos de Eurasianos Occidentales y Asiáticos, y si se comparan las poblaciones indias usando los mismos componentes, emerge el Cline de la India. Esto muestra un gradiente de similitud que podría indicar una divergencia escalonada de poblaciones indias y europeas.
Casi todos los grupos de indios continentales son mixtos
Un análisis posterior del fenómeno India Cline produce resultados interesantes. Por ejemplo, algunas subpoblaciones paquistaníes tienen ascendencia que también cae a lo largo del clino indio. Las poblaciones pueden proyectarse sobre los componentes principales de otras poblaciones: los asiáticos del sur proyectados sobre los componentes principales chinos y europeos producen un efecto lineal (el clino de la India), mientras que los europeos proyectados sobre los componentes principales del sur de Asia y China no. Una interpretación es que la ascendencia india muestra más variabilidad que los otros grupos. Una evaluación de variabilidad similar aparece al comparar poblaciones africanas con no africanas. De este análisis surgen dos hipótesis de árbol:
1. hubo eventos de fundadores en serie en la historia de la India o
2. hubo flujo de genes entre poblaciones ancestrales.
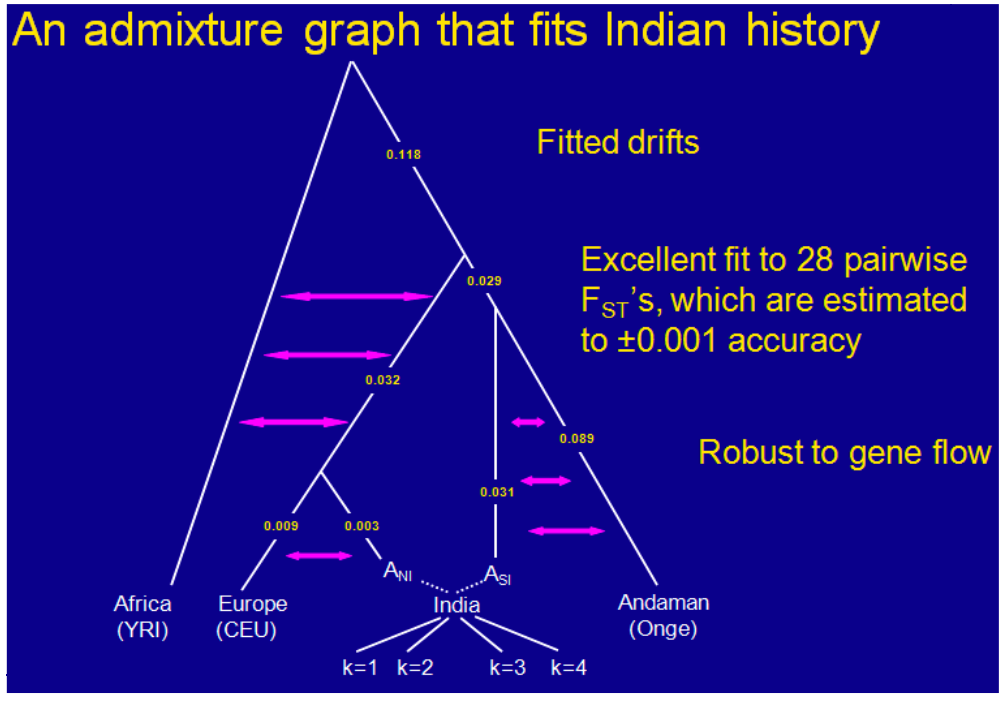
Los autores desarrollaron una prueba formal de cuatro poblaciones para probar hipótesis de ascendencia en presencia de mezcla u otros efectos de confusión. La prueba toma una topología de árbol propuesta y suma sobre todos los SNP de (Ppi Pp2) (Pp3 Pp4), donde los valores de P son frecuencias para las cuatro poblaciones. Si el árbol propuesto es correcto, la correlación será 0 y las poblaciones en cuestión forman un clado. Este método es resistente a varios problemas que limitan otros modelos. Se puede construir un modelo completo para adaptarse a la historia. La información topológica de las gráficas de mezcla se puede aumentar con valores de Fst a través de un procedimiento de ajuste. Este método no hace suposiciones sobre los tiempos de división de la población, la expansión y las contracciones, y la duración del flujo génico, lo que resulta en un procedimiento de estimación más robusto.
Además, al estimar las proporciones de la mezcla utilizando el estadístico de 4 poblaciones, se obtienen estimaciones de error para cada uno de los grupos del árbol. La historia complicada no tiene en cuenta en este cálculo, siempre y cuando la topología determinada por la prueba de 4 poblaciones sea válida.
Estas pruebas y el análisis clino permitieron a los autores determinar la fuerza relativa de la ascendencia ancestral del norte de la India y Ancestral del Sur de la India en cada muestra poblacional representativa. Encontraron
fuente desconocida. Todos los derechos reservados. Este contenido está excluido de nuestro Creativo
Licencia Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
que la alta ascendencia ancestral del norte de la India se correlaciona con la casta tradicionalmente superior y ciertas agrupaciones lingüísticas. Además, la ascendencia ancestral de la India Norte (ANI) y del sur de la India (ASI) es tan diferente de la china como la europea.
La estructura poblacional en la India es diferente a la de Europa
La estructura poblacional en la India está mucho menos correlacionada con la geografía que en Europa. Incluso corrigiendo las poblaciones por diferencias lingüísticas, geográficas y de estatus social, el primer valor es 0.007, aproximadamente 7 veces el de las poblaciones más divergentes de Europa. Una pregunta abierta es si esto podría deberse a la falta de SNP (en gran parte específicos de la India) en las matrices de genotipado. Esto se debe a que el conjunto de SNP dirigidos se identificaron principalmente a partir del proyecto HapMap, que no incluyó fuentes indias.
La mayor parte de la variación genética india no surge de eventos fuera de la India. Adicionalmente, los matrimonios consanguíneos no pueden explicar la señal. Muchos eventos de fundadores en serie, quizás vinculados a las castas o grupos precursores, podrían contribuir. Analizando un solo grupo a la vez, se hace evidente que las castas y subcastas tienen mucha endogamia. La autocorrelación del reparto de alelos entre pares de muestras dentro de un grupo se utiliza para determinar si ocurrió un evento fundador y su edad relativa. Hay segmentos de ADN de un fundador, muchos indicando eventos de más de 1000 años de antigüedad. En la mayoría de los grupos hay evidencia de un fuerte evento fundador antiguo y posterior endogamia. Esto contrasta con la estructura poblacional en la mayor parte de Europa o África, donde se produce más mezcla poblacional (menos endogamia).
Estos eventos de fundadores en serie y su estructura resultante tienen importantes implicaciones médicas. Los fuertes eventos fundadores seguidos de la endogamia y algunas mezclas han llevado a grupos que tienen fuertes propensiones a diversas enfermedades recesivas. Esta estructura significa que los grupos indios tienen una colección de enfermedades prevalentes, similares a las ya conocidas en otros grupos, como los judíos asquenazíes o los finlandeses. La variación única dentro de la India significa que los vínculos con los alelos de enfermedades prevalentes en la India podrían no ser detectables usando solo fuentes de datos no indias. Se necesita un pequeño número de muestras de cada grupo, y más grupos, para mapear mejor estas enfermedades recesivas. Estos mapas se pueden utilizar para predecir mejor los patrones de enfermedades en la India.
28.4.3 Discusión
En general, los fuertes eventos de los fundadores seguidos de la endogamia han dado a la India más subestructura que a Europa. Todos los grupos tribales y castas encuestados muestran una fuerte mezcla de ascendencia ANI y ASI, variando entre 35% y 75% de identidad ANI. Estimar el tiempo y el mecanismo de la mezcla ANI-ASI es actualmente una prioridad alta. Adicionalmente, futuros estudios determinarán si se pueden aplicar nuevas técnicas como la prueba de 4 poblaciones y los gráficos de mezcla a otras poblaciones y cómo.

