3.3: El Núcleo
- Page ID
- 56592
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
El núcleo es el sello distintivo de las células eucariotas; el término mismo eucariota significa tener un “núcleo verdadero”.
La envolvente nuclear
El núcleo está envuelto por un par de membranas que encierran un lumen que es continuo con el del retículo endoplásmico. La membrana interna se estabiliza mediante una malla de proteínas de filamentos intermedios llamadas laminas. La envoltura nuclear está perforada por miles de complejos de poros nucleares (NPC) que controlan el paso de moléculas dentro y fuera del núcleo.
Cromatina
 El núcleo contiene los cromosomas de la célula. Cada cromosoma consiste en una sola molécula de ADN complejada con una masa igual de proteínas. Colectivamente, el ADN del núcleo con sus proteínas asociadas se llama cromatina.
El núcleo contiene los cromosomas de la célula. Cada cromosoma consiste en una sola molécula de ADN complejada con una masa igual de proteínas. Colectivamente, el ADN del núcleo con sus proteínas asociadas se llama cromatina.
La mayor parte de la proteína consiste en múltiples copias de 5 tipos de histonas. Estas son proteínas básicas, erizadas con residuos de arginina y lisina cargados positivamente. (Tanto Arg como Lys tienen un grupo amino libre en su grupo R, que atrae protones (H +) dándoles una carga positiva). Solo la elección de los aminoácidos que harías para unirse firmemente a los grupos fosfato cargados negativamente del ADN.
La cromatina también contiene pequeñas cantidades de una amplia variedad de proteínas no histonas. La mayoría de estos son factores de transcripción (por ejemplo, los receptores de esteroides) y su asociación con el ADN es más transitoria.
La imagen de la derecha muestra las 5 histonas separadas por electroforesis. Estas 5 proteínas varían poco de un tipo celular a otro o incluso de una especie a otra. Sin embargo, las muchas proteínas no histonas en la cromatina (también mostradas a la derecha) varían de un tipo de célula a otro y de una especie a otra. (Cortesía de Gary S. Stein y Janet Swinehart Stein, Universidad de Florida.)
Dos copias de cada uno de los cuatro tipos de histonas H2A, H2B, H3, H4 forman un núcleo de proteína, el núcleo del nucleosoma. Alrededor de esto se envuelven alrededor de 147 pares de bases de ADN. De 20 a 60 pb de ADN enlazan un nucleosoma al siguiente. Cada región enlazadora está ocupada por una sola molécula de histona 1 (H1). Esta región es más larga (50-150 pb) adyacente a los promotores de genes, lo que presumiblemente hace más espacio para la unión de factores de transcripción.
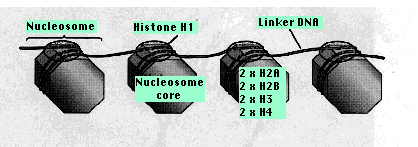
Nucleosomas Nucleosomas Esquemas
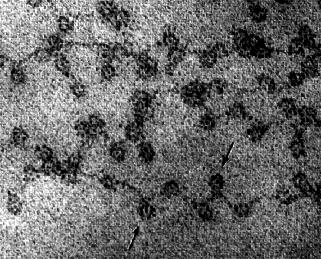
La unión de las histonas al ADN no depende de secuencias de nucleótidos particulares en el ADN, sino que depende críticamente de la secuencia de aminoácidos de la histona. Las histonas son algunas de las moléculas más conservadas durante el transcurso de la evolución. La histona H4 en el ternero difiere de H4 en la planta de guisante en solo 2 residuos de aminoácidos en la cadena de 102. La micrografía electrónica anterior (cortesía de David E. Olins y Ada L. Olins) para nucleosomas muestra cromatina del núcleo de un glóbulo rojo de pollo (las aves, a diferencia de la mayoría de los mamíferos, conservan el núcleo en sus glóbulos rojos maduros). Las flechas apuntan a los nucleosomas. Se puede ver por qué la disposición de los nucleosomas se ha comparado con “cuentas en una cuerda”.
La formación de nucleosomas ayuda algo, pero no lo suficiente, a hacer que el ADN sea lo suficientemente compacto como para encajar en el núcleo. Para encajar 46 moléculas de ADN (en humanos), con un total de más de 2 metros de longitud, en un núcleo que puede tener solo 10 µm de ancho requiere un plegamiento y compactación más extensos. Las interacciones entre las “colas” expuestas de las histonas centrales hacen que los nucleosomas se asocien en una fibra compacta de 30 nm de diámetro. Estas fibras se pliegan luego en estructuras más complejas cuya configuración precisa es incierta y que probablemente cambia con el nivel de actividad de los genes en la región.
Modificaciones de histonas
Aunque su secuencia de aminoácidos (estructura primaria) es invariable, las moléculas de histonas individuales varían en estructura como resultado de modificaciones químicas que ocurren más tarde en aminoácidos individuales. Estos incluyen agregar:
- grupos acetilo (CH 3 CO-) a lisinas
- grupos fosfato a serinas y treoninas
- grupos metilo a lisinas y argininas
Aunque el 75— 80% de la molécula de histona se incorpora en el núcleo, el resto —en el extremo N-terminal— cuelga del núcleo como una “cola” (no se muestra en la figura). La mayoría de las modificaciones químicas ocurren en estas colas, especialmente de H3 y H4. La mayoría de estos cambios son reversibles. Por ejemplo, los grupos acetilo son añadidos por enzimas llamadas histona acetiltransferasas (HAT s) (que no deben confundirse con el medio “HAT” utilizado para producir anticuerpos monoclonales) y también son removidos por histona desacetilasas (HDAC). La mayoría de las veces, la acetilación de las histonas ocurre en regiones de la cromatina que se vuelven activas en la transcripción génica. Esto tiene un sentido intuitivo ya que la adición de grupos acetilo neutraliza las cargas positivas en Lys, reduciendo así la fuerza de la asociación entre el ADN altamente negativo y las histonas altamente positivas.
No obstante, seguramente hay más en la historia. La acetilación de Lys-16 en H4 (“H4K16ac”) evita la interacción de sus “colas” necesarias para formar la estructura compacta de 30 nm de la cromatina inactiva y por lo tanto se asocia con genes activos (nótese que este caso implica interrumpir las interacciones proteína-proteína no proteína-ADN). La metilación, que también neutraliza la carga en las lisinas (y argininas), puede estimular o inhibir la transcripción génica en esa región.
- La adición de 3 grupos metilo a lisina-4 y/o lisina-36 en H3 (H3K4me3 y H3K36me3 respectivamente) se asocia con la transcripción génica activa, mientras que
- trimetilación de lisina-9 y/o lisina-27 en H3 (H3K9me3 y H3K27me3 respectivamente) se asocia con genes inactivos. (Estos incluyen aquellos genes impresos que han sido inactivados permanentemente en células somáticas).
- Y agregar fosfatos hace que los cromosomas se vuelvan más —no menos— compactos a medida que se preparan para la mitosis y la meiosis.
En cualquier caso, ahora está claro que las histonas son un componente dinámico de la cromatina y no simplemente un material inerte para empaquetar el ADN.
Variantes de histonas
Tenemos genes para 8 variedades diferentes de histona 1 (H1). La variedad que se encuentra en un enlazador particular depende de factores tales como el tipo de célula, dónde se encuentra en el ciclo celular, y su etapa de diferenciación. En algunos casos, al menos, una variante particular de H1 se asocia con ciertos factores de transcripción para unirse al potenciador de genes específicos desactivando la expresión de esos genes.
Algunos otros ejemplos de variantes de histonas:
- H3 es reemplazado por CENP-A (“proteína centromérica A”) en los nucleosomas cerca de los centromeros. El hecho de no sustituir el CENP-A por H3 en estas regiones bloquea la estructura y función del centrómero.
- H2A se reemplaza por la variante H2A.Z en promotores y potenciadores génicos.
- Todas las histonas “estándar” son reemplazadas por variantes a medida que se desarrollan los espermatozoides.
En general, las histonas “estándar” se incorporan a los nucleosomas a medida que se sintetiza nuevo ADN durante la fase S del ciclo celular. Posteriormente, algunas son reemplazadas por histonas variantes según lo dictan las condiciones en la célula.
Territorios cromosómicos
Durante la interfase, poco se puede observar de la estructura de la cromatina (excepto en casos especiales como los cromosomas politenos de Drosophila y algunas otras moscas). Aunque cada cromosoma es muy alargado, tiende a ocupar una región discreta dentro del núcleo llamada su territorio. Esto se puede demostrar mediante:
- dirigir un pequeño rayo láser a una pequeña porción del núcleo. Si todos los cromosomas estuvieran entrelazados, uno esperaría que todos recibieran algún daño. Eso no ocurre — sólo uno o dos cromosomas están dañados.
- Las manchas fluorescentes específicas para un cromosoma particular tiñen solo dos regiones en el núcleo, revelando el territorio de los dos homólogas.
Cromosomas “besando”
Porciones de un cromosoma pueden salir de su territorio e interactuar con parte de un cromosoma diferente que sale de su territorio. Se trata de cromosomas “besando”. Los ejemplos que se han encontrado hasta el momento indican que estas interacciones son otra forma de coordinar la actividad de genes que residen en diferentes cromosomas.
El genoma humano contiene muchos genes —dispersos a lo largo de diferentes cromosomas— que se encienden por la llegada de una sola señal. Entre los muchos genes activados por el estrógeno, se encuentran TIFP1 en el cromosoma 21 y GREB1 en el cromosoma 2. Mediante el análisis FISH, investigadores de la Universidad de California en San Diego demostraron que en poco como 2 minutos después de exponer las células a los estrógenos, los loci TIFF1 y GREB1 se mueven de sus respectivos territorios cromosómicos y se “besan”.
En el ratón, las células T colaboradoras ingenuas, a la espera de una señal para dirigirlas a convertirse en células Th1 o células Th2 tienen
- la parte del cromosoma 10 que porta el gen para el interferón gamma (una citocina Th1) besándose
- la parte del cromosoma 11 que porta los genes de IL-4 e IL-5 (citocinas Th2).
Cuando la célula recibe las señales comprometiéndola a una ruta u otra, las dos regiones se separan, la apropiada va a una región de transcripción activa; la otra a una región de heterocromatina.
Y otro ejemplo más (en este caso, dos loci muy separados en el mismo cromosoma “beso”):
En la región de cabeza de la larva de Drosophila, se cierra la expresión de los genes de homeobox (HOX) Antp y Abd-B. El análisis FISH muestra que estos dos loci —separados por 10.000.000 pares de bases en el cromosoma III— se unen en el núcleo unidos por proteínas que impiden su transcripción.
Eucromatina versus Heterocromatina
La densidad de la cromatina que compone cada cromosoma (es decir, qué tan apretada está empaquetada) varía a lo largo de la longitud del cromosoma. Las regiones densas se llaman heterocromatina y las regiones menos densas se llaman eucromatina. La heterocromatina se encuentra en partes del cromosoma donde hay pocos o ningún gen, como
- centromeres y
- telómeros. Esta heterocromatina se encuentra en todo tipo de células del organismo.
- también se encuentra en regiones ricas en genes del cromosoma pero donde los genes están inactivos; es decir, no transcritos. La ubicación de esta heterocromatina varía de un tipo de célula diferenciada a otra (como esperaríamos —una célula hepática, por ejemplo, debería cerrar la expresión de genes que no son necesarios para sus funciones.
- está densamenteembalado.
- está muy enriquecido con transposones y otros tipos de ADN que no contribuyen al proteoma.
- se replica tarde en la fase S del ciclo celular.
- ha reducido el cruce en la meiosis.
- se localiza cerca de la superficie interna de la envoltura nuclear, en la mayoría de las células animales.
- Las histonas en los nucleosomas de la heterocromatina muestran modificaciones características:
- disminución de la acetilación;
- aumento de la metilación de lisina-9 en la histona H3 (H3K9), que ahora proporciona un sitio de unión para la proteína heterocromatina 1 (HP1), que bloquea el acceso por los factores de transcripción necesarios para la transcripción génica
- aumento de la metilación de lisina-27 en histona H3 (H3K27)
Eucromatina
- se encuentra en partes del cromosoma que contienen muchos genes activos.
- está flojamente empaquetado en bucles de fibras de 30 nm.
- separado de hetero cromatina adyacente por aislantes.
- En las células animales, la eucromatina y por lo tanto la transcripción génica activa se produce cerca del centro del núcleo.
- Los genes en eucromatina muestran
- disminución de la metilación de las citosinas en sitios CpG del promotor (es) del gen
- aumento de la acetilación de histonas cercanas
- disminución de la metilación de lisina-9 y lisina-27 en histona H3
Nucleosomas y Transcripción
Los factores de transcripción no pueden unirse a su promotor si el promotor está bloqueado por un nucleosoma. Una de las primeras funciones de los factores de transcripción ensamblados es expulsar el nucleosoma del sitio donde comienza la transcripción o al menos deslizar los nucleosomas a lo largo de la molécula de ADN. Cualquiera de las dos acciones expone el promotor del gen para que los factores de transcripción puedan entonces unirse a él.
La transcripción real de los genes que codifican proteínas se realiza mediante la ARN polimerasa II (Pol II o RNAP II). Para que viaje a lo largo del ADN a transcribir, un complejo de proteínas elimina los nucleosomas frente a él y luego los reemplaza después de que Pol II haya transcrito esa porción de ADN y haya seguido adelante.
Nucleosomas y Replicación de ADN
Como es el caso en la transcripción, la hélice del ADN debe abrirse para permitir que continúe la replicación del ADN. Esto, también, requiere que los nucleosomas que preceden a la horquilla de replicación se retiren y luego se vuelvan a ensamblar rápidamente a medida que se sintetizan las hebras delantera y rezagada.
El Nucleolo
Durante el periodo entre divisiones celulares, cuando los cromosomas están en su estado extendido, uno o más de ellos (10 en células humanas) tienen bucles que se extienden en una masa esférica llamada nucleolo. Aquí se sintetizan tres (de los cuatro) tipos de moléculas de ARN (28S, 18S, 5.8S) utilizadas en el ensamblaje de las subunidades grandes y pequeñas de los ribosomas.
El ARN ribosómico 28S, 18S y 5.8S se transcribe (mediante la ARN polimerasa I) de cientos a miles de genes de ADNr dispuestos en tándely distribuidos (en humanos) en 10 cromosomas diferentes. Las regiones que contienen ADNr de estos 10 cromosomas se agrupan en el nucleolo.
(En la levadura, las moléculas de ARNr 5S —así como las moléculas de ARN de transferencia— también se sintetizan (por la ARN polimerasa III) en el nucleolo.)
Una vez formadas, las moléculas de ARNr se asocian con las docenas de diferentes proteínas ribosómicas utilizadas en el ensamblaje de las subunidades grandes y pequeñas del ribosoma.
Pero las proteínas se sintetizan en el citosol —y todos los ribosomas son necesarios en el citosol para hacer su trabajo—, por lo que debe haber un mecanismo para el transporte de estas grandes estructuras dentro y fuera del núcleo. Esta es una de las funciones de los complejos de poros nucleares.
Complejos Nucleares de Poros (PNJ)
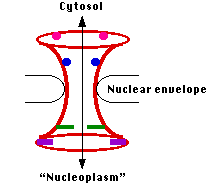 La envoltura nuclear está perforada con miles de poros. Cada uno está construido a partir de múltiples copias de varias docenas de proteínas diferentes llamadas nucleoporinas.
La envoltura nuclear está perforada con miles de poros. Cada uno está construido a partir de múltiples copias de varias docenas de proteínas diferentes llamadas nucleoporinas.
Todo el ensamblaje forma un canal acuoso que conecta el citosol con el interior del núcleo (“nucleoplasma”). Cuando los materiales van a ser transportados a través del poro, se abre para formar un canal de unos 27—41 nm de ancho, lo suficientemente grande como para obtener ensamblajes tan grandes como subunidades ribosómicas.
El transporte a través de los complejos de poros nucleares está activo; es decir, requiere
- energía
- muchas moléculas portadoras diferentes, cada una especializada para transportar una carga en particular
- moléculas de acoplamiento en el NPC (representadas aquí como varillas y discos de colores)
Importar en el núcleo
Las proteínas se sintetizan en el citosol y las que necesita el núcleo deben ser importadas en él a través de los NPC. Incluyen:
- todas las histonas necesarias para hacer los nucleosomas
- todas las proteínas ribosómicas necesarias para el ensamblaje de ribosomas
- todos los factores de transcripción (por ejemplo, los receptores de esteroides) necesarios para activar (y desactivar) los genes
- todos los factores de corte y empalme necesarios para procesar pre-ARNm en moléculas de ARNm maduras; es decir, para cortar regiones de intrones y empalmar las regiones de exón.
Probablemente todas estas proteínas tienen una secuencia característica de aminoácidos —llamada secuencia de localización nuclear (NLS )— que las dirigen para su entrada.
Exportación desde el núcleo
Las moléculas y ensamblajes macromoleculares exportados desde el núcleo incluyen:
- las subunidades ribosómicas que contienen tanto ARNr como proteínas
- moléculas de ARN mensajero (ARNm) (acompañadas de proteínas)
- moléculas de ARN de transferencia (ARNt) (también acompañadas de proteínas)
- factores de transcripción que se devuelven al citosol para esperar su reutilización
Tanto las moléculas de ARN como de proteína contienen una secuencia de exportación nuclear característica (NES) necesaria para asegurar su asociación con las moléculas portadoras adecuadas para llevarlas al citosol.


