15.4F: Diversidad de Receptor de Antígeno
- Page ID
- 56783
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Actualmente se estima que el genoma humano contiene entre 20 y 25 mil genes. El número de receptores de células T para antígeno (TCR) que elaboramos se estima en 2.5 x 10 7; el número de diferentes tipos de moléculas de anticuerpos (BCR) es probablemente aproximadamente el mismo.
¿Cómo podrían 2.5 x 10 4 genes codificar 2.5 x 10 7 TCR diferentes y el mismo número de diferentes BCR?
La respuesta: cada cadena receptora
- cadenas pesadas (H) más kappa (κ) o lambda (λ) para BCR;
- cadenas alfa (α) y beta (β) o gamma (γ) y delta (δ) para TCR)
está codificado por varios segmentos génicos diferentes. El genoma contiene un conjunto de segmentos génicos para cada tipo de cadena. El surtido aleatorio de estos segmentos hace la mayor contribución a la diversidad de receptores.
Células B
Uso del segmento génico para BCR
Para las cadenas pesadas (H) de los BCR (anticuerpos), los segmentos génicos son:
- Segmentos de 51 V H. Cada uno de estos codifica la mayor parte del N-terminal del anticuerpo, incluyendo las dos primeras (pero no la tercera) región hipervariable o determinante de complementariedad (CDR).
- 27 segmentos génicos D H (="diversity”). Éstos codifican parte de la tercera CDR (“CDR3").
- 6 segmentos génicos J H (="joining”). Éstos codifican el resto de la región V de la BCR (incluyendo el resto de CDR3).
- 9 segmentos del gen CH. Éstos codifican la región C de la BCR (y el anticuerpo derivado de ella). Los segmentos del gen C son
- 1 mu (µ); codifica la región C de IgM
- 1 delta (δ) para IgD
- 4 segmentos génicos gamma (γ) para los cuatro tipos de IgG
- 1 épsilon (ε) para IgE
- 2 segmentos génicos alfa (α) para los dos tipos de IgA
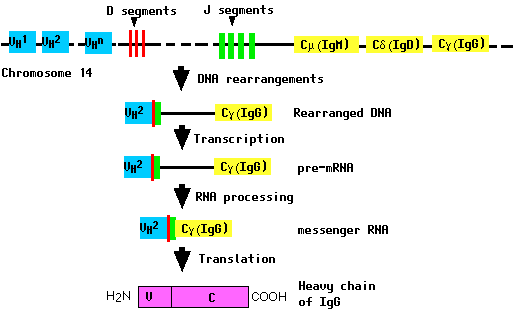
Todos estos segmentos génicos están agrupados en un locus complejo en el cromosoma 14. Durante la diferenciación de la célula B (y mucho antes de cualquier encuentro con un antígeno), el ADN en este locus se corta y se recombina para hacer un gen intacto para la cadena pesada. Este gen puede entonces transcribirse en pre-ARNm, que luego se procesa para formar el ARNm que se traducirá en la cadena polipeptídica pesada (H).
V (D) J Unión

- Cada segmento génico (V, D y J) tiene una secuencia de señal de recombinación (RSS) adyacente
- en el extremo 3' de cada segmento V
- en ambos extremos de cada segmento D
- en el extremo 3' de cada segmento J
- Éstas son reconocidas por dos proteínas codificadas por dos genes G que estimulan la combinación R
- RAG-1
- RAG-2
- Las proteínas RAG-1 y RAG-2 cortan ambas cadenas de ADN en la RSS formando roturas bicatenarias (DSB).
- Luego, la maquinaria regular para reparar DSB (por unión de extremos no homólogos) se balancea en acción.
- Los extremos cortados se cosen juntos (ligados) para formar:
- una junta de codificación (D-J o V-DJ para cadenas pesadas; V-J para cadenas ligeras)
- una articulación de señal (generalmente un bucle de ADN que elimina todo el ADN intermedio inicialmente presente entre los 2 segmentos génicos elegidos).
- La unión D-J ocurre primero; luego el segmento DJ combinado (aún unido al grupo de segmentos génicos de la región constante) se une a un segmento V (como se muestra en la figura).
- El segmento génico V elegido puede estar a miles de pares de bases lejos del segmento D-J, por lo que el cromosoma debe dibujarse en un bucle para unir los dos. El bucle se estabiliza mediante
- una proteína denominada CTCF (“factor de unión a CCCTC”; denominada así por la secuencia de nucleótidos a la que se une). El CTCF en el sitio D-J en el ADN forma un dímero con el CTCF en el sitio V en el ADN que une las dos regiones juntas.
- cohesin: el mismo complejo proteico que mantiene unidas a las cromátidas hermanas durante la mitosis y la meiosis.
- En el proceso, el grupo de segmentos génicos se mueve desde la periferia del núcleo (una región de genes inactivos) hasta el centro del núcleo (una región de transcripción génica activa).
Algunos casos de inmunodeficiencia combinada severa en humanos (IDCG) son causados por defectos en la unión V (D) J.
- Una versión es causada por mutaciones en ambas copias de RAG1 o RAG2.
- Otra es causada por mutaciones en un gen necesario para la unión de extremos no homólogos. (No se forma ninguna junta de codificación aunque una junta de señal se forme normalmente).
Si los segmentos génicos 51 V H, 27 D H y 6 J H se ensamblaran aleatoriamente (probablemente no lo son), eso proporcionaría un mínimo de 8.3 x 10 3 diferentes posibles combinaciones. Pero las posibilidades de diversidad de la región V de anticuerpos resultan ser mayores que eso. El proceso de recombinación no es preciso.
- Los puntos exactos de corte y empalme entre V H y D H y entre D H y J H pueden variar a lo largo de varios nucleótidos
- También se pueden insertar nucleótidos adicionales, llamados regiones N, en estas articulaciones.
- Todos estos se suman en gran medida a la diversidad de CDC3.
Cadenas ligeras
Una vez que el gen de la cadena H se ensambla, transcribe y traduce, la cadena H resultante puede emparejarse con una cadena L que es en sí misma el producto de un proceso de recombinación similar que ocurre
- en el cromosoma 2 para los segmentos del gen kappa
- en el cromosoma 22 para los segmentos del gen lambda
| Anticuerpos (BCR) | Segmentos génicos | Combinaciones |
|---|---|---|
| Vκ | 40 | |
| Jκ | 5 | 200 cadenas de κ |
| Vλ | 31 | |
| Jλ | 4 | 124 cadenas λ |
| V H | 51 | |
| D H | 25 | |
| J H | 6 | 7,650 cadenas H |
| Cualquier cadena H con cualquier cadena L (324) | 2.5 x 10 6 | |
Como muestra la tabla, esto sienta las bases para un repertorio potencial de células B de 2.5 x 10 6 regiones V de anticuerpos diferentes. Pero el número verdadero es probablemente prácticamente ilimitado debido a la variación en el punto de corte y empalme exacto y la introducción de N nucleótidos ambos de los cuales aumentan la diversidad de CDR3.
La diversidad tiene un precio
La combinación de los segmentos génicos V, D y J junto con la incorporación aleatoria de nucleótidos adicionales (regiones N) en las articulaciones, crea una enorme variabilidad de codificación. También crea un alto riesgo (dos de cada tres veces) de introducir un desplazamiento de marco para que los codones para el resto de la región V codifiquen tonterías. Aunque muchas células B se desperdician, las probabilidades no son tan malas como parecen. Si la célula B no logra producir un producto funcional a partir del grupo de segmentos génicos en uno de sus cromosomas, puede recurrir a los segmentos génicos en su homólogo y volver a intentarlo. Si falla ambas veces para hacer una cadena L kappa funcional, todavía tiene dos intentos de hacer una cadena L lambda funcional.
Hipermutación somática (SHM) y diversidad de anticuerpos
Los mecanismos de diversificación descritos anteriormente tienen lugar antes de que la célula B se encuentre con el antígeno. Después de que una célula B se encuentre con antígeno, puede comenzar la mitosis, creciendo en un clon de células sintetizando el mismo BCR (y, eventualmente, secretando anticuerpos con el mismo sitio de unión). Las mutaciones puntuales pueden ocurrir mientras esto sucede. Algunos de estos pueden generar un sitio de unión con mayor afinidad por su epítopo. Se trata de mutaciones favorables, y el “subclón” en el que ocurren tiende a ser favorecido y puede sustituir al clon ancestral. El resultado es la maduración por afinidad, la producción de anticuerpos de afinidad cada vez mayor por el antígeno.
Recombinación de conmutación de clase (CSR)

A medida que las células B crecen en un clon en respuesta al antígeno, pueden reordenar su ADN una vez más. Por ejemplo, una célula B que ha ensamblado un gen completo para la cadena H de IgM (µ), puede cortar el gen en el lado 3' de los segmentos de la región V ensamblados y mover el ensamblaje al lado 5' de otro de sus segmentos génicos CH. Ahora la célula comienza a producir una clase diferente de anticuerpos, como IgG o IgA. Pero la especificidad antigénica del anticuerpo sigue siendo la misma porque el N-terminal de la cadena H permanece inalterado (como lo hace toda la cadena L).
La recombinación de cambio de clase permite al cuerpo producir anticuerpos con diferentes funciones efectoras; es decir, diferentes medios para tratar con el mismo antígeno. La capacidad de una célula B para cambiar los segmentos génicos C H depende de que reciba ayuda de las células T colaboradoras.
Células T
Células T alfa/beta (αβ)
Las células T más abundantes en la sangre expresan un receptor de antígeno (TCR) que es un heterodímero de dos cadenas designadas alfa (α) y beta (β). Cada uno de estos está codificado por un gen ensamblado a partir de los segmentos génicos V, D, J y C. Al igual que los BCR, existen múltiples variantes de estos segmentos génicos dispuestos en grupos:
- segmentos génicos de la cadena alfa en el cromosoma 14
- segmentos génicos de la cadena beta en el cromosoma 7
| Receptores de células T (TCR) | Segmentos génicos | Combinaciones |
|---|---|---|
| Vα | 50 | |
| Jα | 50 | 2.5 x 10 3 cadenas alfa |
| Vβ | 20 | |
| Jβ | 13 | |
| Dβ | 2 | 520 cadenas beta |
| Cualquier alfa con cualquier cadena beta | 1.3 x 10 6 | |
Y al igual que las células B, la mayor diversidad en los receptores de las células T αβ ocurre en la tercera región determinante de la complementariedad (CDR3) de las cadenas alfa y beta debido a la diversidad de unión entre los segmentos V, J y D y la adición de nucleótidos de la región N. Sin embargo, las células T no parecen usar mutación somática para aumentar la diversidad de receptores. Las mediciones reales del repertorio en humanos revelan una cifra de aproximadamente 2.5 x 10 7.
Células T gamma/delta (γδ)
El repertorio TCR de las células T γδ parece mucho más pequeño que el de sus primos αβ. Los segmentos génicos de la cadena gamma se agrupan en el cromosoma 7. Los segmentos génicos de la cadena delta están agrupados en el cromosoma 14 (dentro del grupo de cadenas alfa).


