9.4: Traducción
- Page ID
- 54069
La síntesis de proteínas es uno de los procesos metabólicos que más consumen energía en una célula. A su vez, las proteínas representan más masa que cualquier otro componente de los organismos vivos (con la excepción del agua), y las proteínas realizan una amplia variedad de las funciones de una célula. El proceso de traducción, o síntesis de proteínas, implica decodificar un mensaje de ARNm en un producto polipeptídico. Los aminoácidos se encadenan covalentemente en longitudes que van desde aproximadamente 50 aminoácidos hasta más de 1,000.
La maquinaria de síntesis de proteínas
Además del molde de ARNm, muchas otras moléculas contribuyen al proceso de traducción. La composición de cada componente puede variar entre especies; por ejemplo, los ribosomas pueden consistir en diferentes números de ARN ribosómicos (ARNr) y polipéptidos dependiendo del organismo. Sin embargo, las estructuras y funciones generales de la maquinaria de síntesis de proteínas son comparables de bacterias a células humanas. La traducción requiere la entrada de un molde de ARNm, ribosomas, ARNt y diversos factores enzimáticos (Figura\(\PageIndex{1}\)).
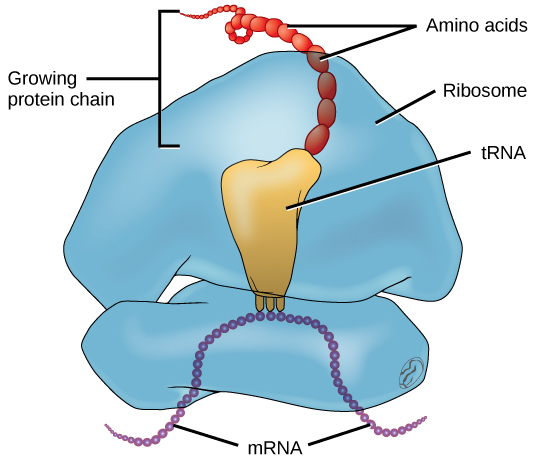
En E. coli, hay 200 mil ribosomas presentes en cada célula en un momento dado. Un ribosoma es una macromolécula compleja compuesta por ARNr estructurales y catalíticos, y muchos polipéptidos distintos. En eucariotas, el nucleolo está completamente especializado para la síntesis y ensamblaje de ARNr.
Los ribosomas se localizan en el citoplasma en procariotas y en el citoplasma y retículo endoplásmico de eucariotas. Los ribosomas están formados por una subunidad grande y una pequeña que se unen para su traducción. La subunidad pequeña es responsable de unirse al molde de ARNm, mientras que la subunidad grande se une secuencialmente a ARNt, un tipo de molécula de ARN que lleva aminoácidos a la cadena en crecimiento del polipéptido. Cada molécula de ARNm es traducida simultáneamente por muchos ribosomas, todos sintetizando proteínas en la misma dirección.
Dependiendo de la especie, existen de 40 a 60 tipos de ARNt en el citoplasma. Sirviendo como adaptadores, los ARNt específicos se unen a secuencias en el molde de ARNm y agregan el aminoácido correspondiente a la cadena polipeptídica. Por lo tanto, los ARNt son las moléculas que realmente “traducen” el lenguaje del ARN al lenguaje de las proteínas. Para que cada ARNt funcione, debe tener su aminoácido específico unido a él. En el proceso de “carga” de ARNt, cada molécula de ARNt está unida a su aminoácido correcto.
El Código Genético
Para resumir lo que conocemos hasta este punto, el proceso celular de transcripción genera ARN mensajero (ARNm), una copia molecular móvil de uno o más genes con un alfabeto de A, C, G y uracilo (U). La traducción del molde de ARNm convierte la información genética basada en nucleótidos en un producto proteico. Las secuencias proteicas constan de 20 aminoácidos que ocurren comúnmente; por lo tanto, se puede decir que el alfabeto proteico consta de 20 letras. Cada aminoácido se define por una secuencia de tres nucleótidos llamada codón triplete. La relación entre un codón nucleotídico y su aminoácido correspondiente se denomina código genético.
Dados los diferentes números de “letras” en los “alfabetos” de ARNm y proteína, las combinaciones de nucleótidos correspondieron a aminoácidos individuales. El uso de un código de tres nucleótidos significa que hay un total de 64 (4 × 4 × 4) combinaciones posibles; por lo tanto, un aminoácido dado es codificado por más de un triplete de nucleótidos (Figura\(\PageIndex{2}\)).
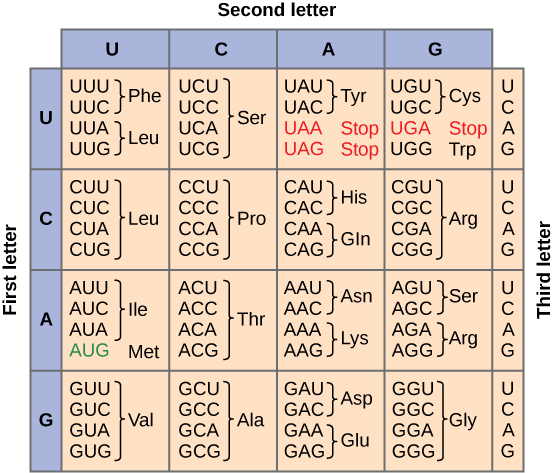
Tres de los 64 codones terminan la síntesis de proteínas y liberan el polipéptido de la maquinaria de traducción. A estos trillizos se les llama codones de parada. Otro codón, AUG, también tiene una función especial. Además de especificar el aminoácido metionina, también sirve como codón de inicio para iniciar la traducción. El marco de lectura para la traducción se establece mediante el codón de inicio AUG cerca del extremo 5' del ARNm. El código genético es universal. Con algunas excepciones, prácticamente todas las especies utilizan el mismo código genético para la síntesis de proteínas, lo que es una poderosa evidencia de que toda la vida en la Tierra comparte un origen común.
El mecanismo de síntesis de proteínas
Al igual que con la síntesis de ARNm, la síntesis de proteínas se puede dividir en tres fases: iniciación, elongación y terminación. El proceso de traducción es similar en procariotas y eucariotas. Aquí exploraremos cómo se produce la traducción en E. coli, un procariota representativo, y especificaremos cualquier diferencia entre la traducción procariota y eucariota.
La síntesis de proteínas comienza con la formación de un complejo de iniciación. En E. coli, este complejo involucra la subunidad ribosómica pequeña, el molde de ARNm, tres factores de iniciación y un ARNt iniciador especial. El ARNt iniciador interactúa con el codón de inicio AUG y se une a una forma especial del aminoácido metionina que normalmente se elimina del polipéptido después de completar la traducción.
En procariotas y eucariotas, los fundamentos del alargamiento de polipéptidos son los mismos, por lo que revisaremos la elongación desde la perspectiva de E. coli. La subunidad ribosómica grande de E. coli consta de tres compartimentos: el sitio A se une a los ARNt cargados entrantes (ARNt con sus aminoácidos específicos unidos). El sitio P se une a ARNt cargados que portan aminoácidos que han formado enlaces con la cadena polipeptídica en crecimiento pero que aún no se han disociado de su ARNt correspondiente. El sitio E libera ARNt disociados para que puedan recargarse con aminoácidos libres. El ribosoma desplaza un codón a la vez, catalizando cada proceso que ocurre en los tres sitios. Con cada etapa, un ARNt cargado ingresa al complejo, el polipéptido se vuelve un aminoácido más largo y se aparta un ARNt sin carga. La energía para cada enlace entre aminoácidos se deriva de GTP, una molécula similar al ATP (Figura\(\PageIndex{3}\)). Sorprendentemente, el aparato de traducción de E. coli tarda solo 0.05 segundos en agregar cada aminoácido, lo que significa que un polipéptido de 200 aminoácidos podría traducirse en solo 10 segundos.
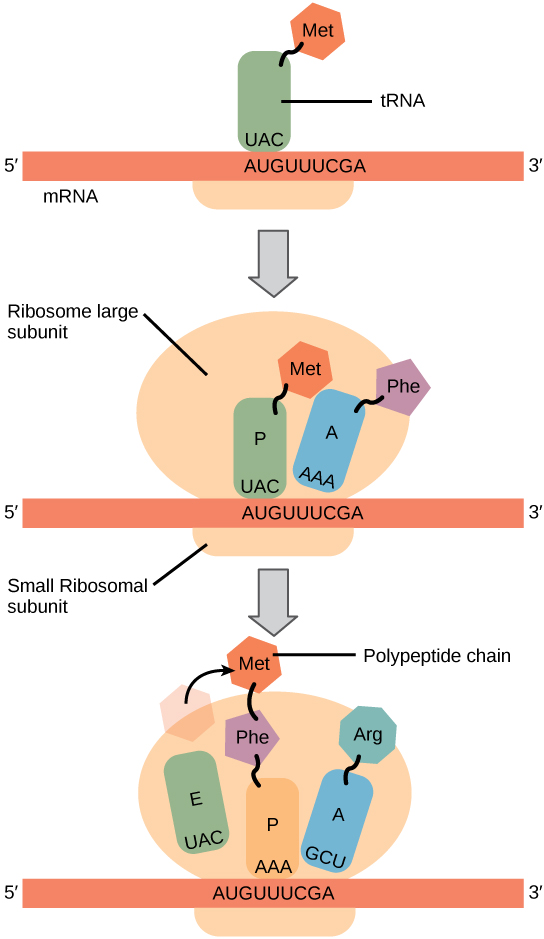
La terminación de la traducción ocurre cuando se encuentra un codón de parada (UAA, UAG o UGA). Cuando el ribosoma se encuentra con el codón de parada, el polipéptido en crecimiento se libera y las subunidades del ribosoma se disocian y dejan el ARNm. Después de que muchos ribosomas hayan completado la traducción, el ARNm se degrada por lo que los nucleótidos se pueden reutilizar en otra reacción de transcripción.
CONCEPT EN ACCIÓN
Transcribir un gen y traducirlo a proteína usando apareamiento complementario y el código genético en este sitio.
Resumen
El dogma central describe el flujo de información genética en la célula desde los genes hasta el ARNm y las proteínas. Los genes se utilizan para producir ARNm por el proceso de transcripción; el ARNm se usa para sintetizar proteínas por el proceso de traducción. El código genético es la correspondencia entre el codón de ARNm de tres nucleótidos y un aminoácido. El código genético es “traducido” por las moléculas de ARNt, las cuales asocian un codón específico con un aminoácido específico. El código genético es degenerado porque 64 codones tripletes en ARNm especifican solo 20 aminoácidos y tres codones de terminación. Esto significa que más de un codón corresponde a un aminoácido. Casi todas las especies del planeta utilizan el mismo código genético.
Los actores en la traducción incluyen el molde de ARNm, ribosomas, ARNt y varios factores enzimáticos. La subunidad ribosómica pequeña se une al molde de ARNm. La traducción comienza en el AUG de inicio en el ARNm. La formación de enlaces ocurre entre aminoácidos secuenciales especificados por el molde de ARNm de acuerdo con el código genético. El ribosoma acepta ARNt cargados, y a medida que avanza a lo largo del ARNm, cataliza la unión entre el nuevo aminoácido y el extremo del polipéptido en crecimiento. El ARNm completo se traduce en “etapas” de tres nucleótidos del ribosoma. Cuando se encuentra un codón de parada, un factor de liberación se une y disocia los componentes y libera la nueva proteína.
Glosario
- codón
- tres nucleótidos consecutivos en ARNm que especifican la adición de un aminoácido específico o la liberación de una cadena polipeptídica durante la traducción
- código genético
- los aminoácidos que corresponden a codones de tres nucleótidos de ARNm
- ARNr
- ARN ribosómico; moléculas de ARN que se combinan para formar parte del ribosoma
- codón de parada
- uno de los tres codones de ARNm que especifica la terminación de la traducción
- codón de inicio
- el AUG (o, raramente GUG) en un ARNm a partir del cual comienza la traducción; siempre especifica metionina
- ARNt
- transferir ARN; una molécula de ARN que contiene una secuencia específica de anticodón de tres nucleótidos para emparejarse con el codón de ARNm y también se une a un aminoácido específico


