10.4: Transposones - “genes saltadores”
- Page ID
- 57167
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Los transposones son segmentos de ADN que pueden moverse a diferentes posiciones en el genoma de una sola célula. En el proceso, pueden provocar mutaciones e incrementar (o disminuir) la cantidad de ADN en el genoma de la célula, y si la célula es precursora de un gameto, en los genomas de cualquier descendiente. Estos segmentos móviles de ADN a veces se llaman “genes saltadores” y hay dos tipos distintos. Los transposones Clase II consisten en ADN que se mueve directamente de un lugar a otro. Los transposones de clase I son retrotransposones que primero transcriben el ADN en ARN y luego usan transcriptasa inversa para hacer una copia de ADN del ARN para insertarlo en una nueva ubicación.
Transposones Clase II
Los transposones de Clase II se mueven por un proceso de "cortar y pegar": el transposón se corta de su ubicación (como Comando/Control-X en su computadora) y se inserta en una nueva ubicación (Comando/Control-V). Este proceso requiere una enzima —una transposasa — que esté codificada dentro de algunos de estos transposones.

Fig.10.4.1 Transposones
La transposasa se une a ambos extremos del transposón, que consisten en repeticiones invertidas; es decir, secuencias idénticas que leen en direcciones opuestas. También se unen a una secuencia de ADN que conforma el sitio diana. Algunas transposasas requieren una secuencia específica como su sitio diana; otras pueden insertar el transposón en cualquier parte del genoma.
El ADN en el sitio diana se corta de manera desplazada (como los “extremos pegajosos” producidos por algunas enzimas de restricción). Después de ligar el transposón al ADN del huésped, los huecos se rellenan mediante el apareamiento de bases Watson-Crick. Esto crea repeticiones directas idénticas en cada extremo del transposón. A menudo, los transposones pierden su gen para la transposasa. Sin embargo, siempre y cuando en algún lugar de la célula haya un transposón que pueda sintetizar la enzima, sus repeticiones invertidas son reconocidas y ellas, también, pueden trasladarse a una nueva ubicación.
Elementos transponibles de repetición invertida en miniatura (ácaros)
La reciente finalización de la secuencia genómica de arroz y C. elegans ha revelado que sus genomas contienen miles de copias de un motivo recurrente que consiste en secuencias casi idénticas de aproximadamente 400 pares de bases flanqueadas por repeticiones invertidas características de aproximadamente 15 pares de bases tales como
5' GGCCAGTCACAATGG.. ~400 nt.. CCATTGTGACTGGCC 3'
3' CCGGTCAGTGTTACC.. ~400 nt.. GGTAACACTGACCGG 5'
Los ácaros son demasiado pequeños para codificar cualquier proteína. Aún no se sabe cómo se copian y se trasladan a nuevas ubicaciones. Probablemente los transposones más grandes que sí codifican la enzima necesaria y reconocen las mismas repeticiones invertidas son los responsables. Hay más de 100 mil ácaros en el genoma del arroz (lo que representa alrededor del 6% del genoma total). Algunas de las mutaciones que se encuentran en ciertas cepas de arroz son causadas por la inserción de un MITE en el gen. También se han encontrado ácaros en los genomas de humanos, Xenopus y manzanas.
Transposones en Maíz
Los primeros transposones fueron descubiertos en la década de 1940 por Barbara McClintock quien trabajaba con maíz (Zea mays, llamado “maíz” en Estados Unidos). Encontró que eran responsables de una variedad de tipos de mutaciones genéticas, generalmente inserciones y deleciones (indels) y translocaciones. Algunas de las mutaciones (c, bz) utilizadas como ejemplos de cómo se mapean los loci génicos en el cromosoma fueron causadas por transposones. En el desarrollo de tejidos somáticos como los granos de maíz, una mutación (e.g., c) que altera el color se transmitirá a todas las células descendientes. Esto produce el patrón abigarrado que es tan apreciado en el “maíz indio”. (Foto cortesía de Whals Farms.) Otros científicos tardaron unos 40 años en apreciar plenamente la importancia de los descubrimientos de Barbara McClintock. Finalmente fue galardonada con el Premio Nobel en 1983.

Transposones en Drosophila
Los elementos P son transposones Clase II encontrados en Drosophila. Hacen poco daño porque la expresión de su gen transposasa suele ser reprimida. Sin embargo, cuando las moscas macho con elementos P se aparean con moscas hembra que carecen de ellos, la transposasa se activa en la línea germinal produciendo tantas mutaciones que sus crías son estériles. En la naturaleza esto ya no es un problema. Los elementos P parecen haber aparecido por primera vez en Drosophila melanogaster hace unos 50 años. Desde entonces, se han extendido por todas las poblaciones de la especie. Hoy en día, las moscas que carecen de elementos P solo se pueden encontrar en cepas viejas mantenidas en el laboratorio. Los elementos P han proporcionado valiosas herramientas para los genetistas de Drosophila. Las moscas transgénicas que contienen cualquier gen deseado se pueden producir inyectando al embrión temprano un elemento P manipulado que contiene ese gen. Otros transposones están siendo estudiados por su capacidad para crear insectos transgénicos de importancia agrícola y de salud pública.
Transposones en bacterias
Algunos transposones en bacterias llevan —además del gen de la transposasa— genes para una o más (generalmente más) proteínas que imparten resistencia a los antibióticos. Cuando dicho transposón se incorpora en un plásmido, puede abandonar la célula huésped y trasladarse a otra. Esta es la manera en que el alarmante fenómeno de la resistencia a los antibióticos multidrogas se propaga tan rápidamente. La transposición en estos casos ocurre por un mecanismo de "copiar y pegar"”. Esto requiere una enzima adicional —una resolvasa — que también esté codificada en el propio transposón. El transposón original permanece en el sitio original mientras su copia se inserta en un nuevo sitio.
Retrotransposones
Los retrotransposones también se mueven por un mecanismo de "copiar y pegar" pero a diferencia de los transposones descritos anteriormente, la copia está hecha de ARN, no de ADN. Luego, las copias de ARN se transcriben de nuevo en ADN —usando una transcriptasa inversa— y éstas se insertan en nuevas ubicaciones en el genoma. Muchos retrotransposones tienen repeticiones terminales largas (LTR) en sus extremos que pueden contener más de 1000 pares de bases en cada uno. Al igual que los transposones de ADN, los retrotransposones generan repeticiones directas en sus nuevos sitios de inserción. De hecho, es la presencia de estas repeticiones directas lo que muchas veces es la pista de que el tramo intermedio de ADN llegó allí por retrotransposición. Alrededor del 50% de todo el genoma humano consiste en retrotransposones.
LINES (Elementos intercalados largos)
El genoma humano contiene más de un millón de LINES (que representan el 19% del genoma). Los más abundantes de estos pertenecen a una familia llamada LINE-1 (L1). Estos elementos L1 son secuencias de ADN que varían en longitud desde unos pocos cientos hasta hasta 9,000 pares de bases. Sólo alrededor de 50 elementos L1 son “genes” funcionales; es decir, pueden ser transcritos y traducidos. Los elementos L1 funcionales tienen aproximadamente 6,500 pb de longitud y codifican tres proteínas, incluyendo una endonucleasa que corta ADN y una transcriptasa inversa que hace una copia de ADN de un transcrito de ARN.
La actividad L1 procede de la siguiente manera:
- La ARN polimerasa II transcribe el ADN de L1 en ARN.
- El ARN es traducido por ribosomas en el citoplasma a las proteínas.
- Las proteínas y el ARN se unen y vuelven a entrar en el núcleo.
- La endonucleasa corta una cadena de ADN “diana”, a menudo en el intrón de un gen.
- La transcriptasa inversa copia el ARN L1 en ADN L1 que se inserta en el ADN diana formando allí un nuevo elemento L1.
A través de este mecanismo de copia-pasta, el número de líneas puede aumentar en el genoma. La diversidad de líneas entre genomas humanos individuales los convierte en marcadores útiles para la “huella digital” de ADN. La variación ocurre en la longitud de los elementos L1: La transcripción de un elemento L1 activo a veces continúa aguas abajo en ADN adicional produciendo un elemento transpuesto más largo. La transcripción inversa del ARN L1 a menudo concluye prematuramente y produce un elemento transpuesto acortado.
Si bien los elementos L1 no son funcionales, pueden desempeñar un papel en la regulación de la eficiencia de la transcripción del gen en el que residen. Ocasionalmente, la actividad L1 produce e inserta una copia de un ARNm celular (por lo tanto, un ADNc natural). Al carecer de intrones así como de los elementos de control necesarios como promotores, estos genes no se expresan. Representan una categoría de pseudogén.
SINEs (Elementos intercalados cortos)
Los SINE son secuencias cortas de ADN (100-400 pares de bases) que representan moléculas de ARN transcritas inversamente transcritas originalmente por la ARN polimerasa III; es decir, moléculas de ARNt, ARNr 5S y algunos otros ARN nucleares pequeños. Los SINE más abundantes son los elementos Alu. Hay más de un millón de copias en el genoma humano (lo que representa 9% de nuestro ADN total). Los elementos Alu consisten en una secuencia con un promedio de 260 pares de bases que contiene un sitio que es reconocido por la enzima de restricción AluI. Parecen ser transcripciones inversas de ARN 7S, parte de la partícula de reconocimiento de señal. La mayoría de los SINE no codifican ninguna molécula funcional y dependen de la maquinaria de los elementos L1 activos para ser transpuestos; es decir, copiados y pegados en nuevas ubicaciones.
VIH-1
El VIH-1 —la causa del SIDA— y otros retrovirus humanos (por ejemplo, HTLV-1, el virus de la leucemia/linfoma de células T humanas) se comportan como retrotransposones. El genoma de ARN del VIH-1 contiene un gen para la transcriptasa inversa y uno para la integrasa. La integrasa cumple la misma función que las transposasas de los transposones de ADN. Las copias de ADN se pueden insertar en cualquier parte del genoma. Las moléculas de ambas enzimas se incorporan en la partícula del virus.
Transposones y Mutaciones
Los transposones son mutágenos y pueden causar mutaciones de varias maneras. Si un transposón se inserta en un gen funcional, probablemente lo dañará. La inserción en exones, intrones e incluso en ADN que flanquea los genes (que pueden contener promotores y potenciadores) puede destruir o alterar la actividad del gen. La reparación defectuosa de la brecha que queda en el sitio antiguo (en transposición de corte y pasta) puede llevar a la mutación allí. La presencia de una cadena de secuencias repetidas idénticas presenta un problema para el emparejamiento preciso durante la meiosis. ¿Cómo va a asegurar la tercera, digamos, de una cadena de cinco secuencias de Alu en la “hebra invasora” de una cromátida que se empareja con la tercera secuencia en la otra hebra? Si accidentalmente se empareja con una de las otras secuencias de Alu, el resultado será un cruce desigual —una de las causas más comunes de duplicaciones.
Se cree que la inserción de un retrotransposón en el ADN flanqueando un gen para la síntesis de pigmentos produjo uvas blancas a partir de un antepasado de piel negra. Posteriormente, la pérdida de ese retrotransposón produjo las variedades de uva de piel roja que se cultivan hoy en día.
Los SINE (en su mayoría secuencias de Alu) y LINES causan solo un pequeño porcentaje de mutaciones humanas. (Incluso puede haber un mecanismo por el cual eviten insertarse en genes funcionales). Sin embargo, se ha encontrado que son la causa de las mutaciones responsables de algunos casos de enfermedades genéticas humanas, entre ellas:
- Hemofilia A (gen del Factor VIII) y Hemofilia B [Gen del Factor IX]
- Inmunodeficiencia Combinada Severa Ligada al X (SCID) [gen para parte del receptor IL-2]
- porfiria
- predisposición a pólipos de colon y cáncer [gen APC]
- Distrofia muscular de Duchenne [gen de la distrofina]
¿De qué sirven los transposones?
Los transposones se han llamado ADN “basura” y ADN “egoísta”. Son “egoístas” porque su única función parece hacer más copias de sí mismos y “basura” porque no hay beneficio obvio para su anfitrión. Debido a las similitudes de secuencia de todas las LINES y SINE, también constituyen una gran parte del “ADN repetitivo” de la célula. Los retrotransposones no pueden ser tan egoístas que reduzcan la supervivencia de su huésped. Y ahora parece que muchos, al menos, confieren algún beneficio. El proyecto ENCODE encontró que alrededor del 75% de nuestro ADN repetitivo ocurre dentro de secuencias, como potenciadores, que regulan la expresión génica o se solapa con ellas.
Algunas otras posibilidades:
- Los retrotransposones a menudo llevan algunas secuencias adicionales en su extremo 3' a medida que se insertan en una nueva ubicación. Quizás estos ocasionalmente crean nuevas combinaciones de exones, promotores y potenciadores que benefician al huésped.
Ejemplo:
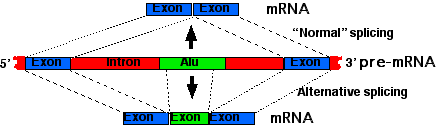
- Miles de nuestros elementos Alu ocurren en los intrones de los genes.
- Algunos de estos contienen secuencias que cuando se transcriben en el transcrito primario son reconocidas por el spliceosoma.
- Estos pueden luego ser empalmados en el ARNm maduro creando un nuevo exón, que se transcribirá en un nuevo producto proteico.
- El empalme alternativo puede proporcionar no solo el nuevo ARNm (y por lo tanto la proteína) sino también el antiguo.
- De esta manera, la naturaleza puede probar nuevas proteínas sin el riesgo de abandonar la probada y verdadera vieja.
- Los elementos L1 insertados en los intrones de genes funcionales reducen la transcripción de esos genes sin dañar el producto génico; cuanto más largo sea el elemento L1, menor es el nivel de expresión génica. Alrededor del 79% de nuestros genes contienen elementos L1, y tal vez sean un mecanismo para establecer el nivel basal de actividad génica.
- La telomerasa, la enzima esencial para mantener la longitud de los cromosomas, está estrechamente relacionada con la transcriptasa inversa de LINES y puede haber evolucionado a partir de ella.
- RAG-1 y RAG-2. Las proteínas codificadas por estos genes son necesarias para ensamblar el repertorio de anticuerpos y receptores de células T (TCR) utilizados por el sistema inmune adaptativo. El mecanismo se asemeja al método de corte y pasta de los transposones Clase II, y los genes RAG pueden haber evolucionado a partir de ellos. Si es así, el suceso ocurrió hace unos 450 millones de años cuando los vertebrados mandíbulas evolucionaron a partir de ancestros sin mandíbula. Solo los vertebrados mandíbulas tienen los genes RAG-1 y RAG-2.
- En Drosophila, la inserción de transposones en genes se ha relacionado con el desarrollo de resistencia al DDT y a los insecticidas organofosforados.
Los transposones y la paradoja del valor C
El genoma de Arabidopsis thaliana contiene ~1.2 x 10 8 pares de bases (pb) de ADN. Alrededor del 14% de esto consiste en transposones; el resto genes funcionales (25,498 de ellos). El genoma del maíz (maíz) contiene 20 veces más ADN (2.4 x 10 9 pb) pero seguramente no necesita 20 veces más genes. De hecho, 60% del genoma del maíz está compuesto por transposones (la cifra para los humanos es 42%). Entonces parece probable que la falta de asociación entre el tamaño del genoma y el número de genes funcionales —la paradoja del valor C— sea causada por la cantidad de ADN del transposón acumulado en el genoma.


