2.4: Dogma Central y el Código Genético
- Page ID
- 57341
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)El “Dogma Central”
Hemos visto cómo el ADN, con la ayuda de polimerasas específicas y proteínas accesorias, es capaz de replicarse. También hemos visto cómo podemos usar esta información para crear elementos extracromosómicos de replicación autónoma (es decir, plásmidos). Sin embargo, la utilidad real de tales sistemas surge cuando los usamos para crear proteínas de interés. Para llegar a las proteínas tenemos que pasar primero por el ARN.
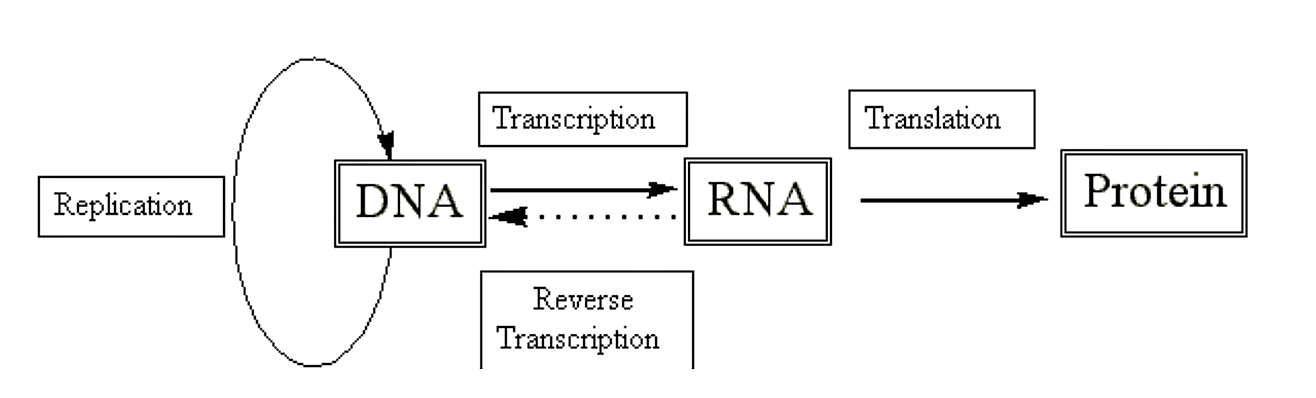.png)
Figura 2.4.1: Dogma central
Características estructurales del ARN:
- Similar al ADN excepto que contiene un grupo 2' hidoxilo (hace que el enlace fosfodiéster sea más lábil que el ADN).
- La timina en el ADN es reemplazada por Uracilo en el ARN
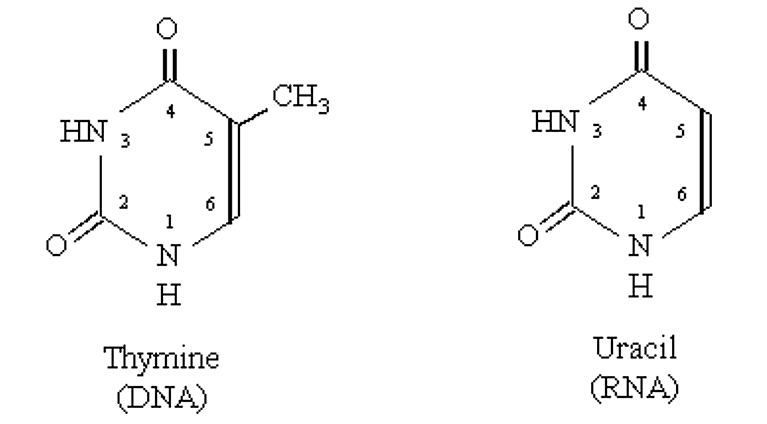.png)
Figura 2.4.2: Timina vs. Uracilo
3. Los ARN pueden adoptar estructuras tridimensionales regulares que les permiten funcionar en el proceso de expresión genética (es decir, la producción de proteínas).
- Esta capacidad de adoptar estructuras tridimensionales definidas que imparten funcionalidad coloca al ARN en una clase única, algo similar a las proteínas y diferente del ADN.
- Por ejemplo, ciertas moléculas de ARN, cuando están plegadas, exhiben capacidades catalíticas (por ejemplo, la escisión de moléculas de ARN).
- La mayoría del ARN en las células se encuentra en complejos con proteínas. El ejemplo más común son los ribosomas (involucrados en la síntesis de proteínas).
Transcripción: la copia de ADN por una ARN polimerasa para hacer ARN.
ARN polimerasa:
- Puede iniciar una nueva cadena de ácido nucleico dada una plantilla.
- Las ADN polimerasas no pueden; requieren un cebador (o más típicamente, una ARN polimerasa para proporcionar el cebador).
Síntesis de Proteínas
- Tres tipos de moléculas de ARN realizan diferentes funciones en el aparato de síntesis de proteínas:
- El ARN mensajero (ARNm) codifica la información genética copiada del ADN en forma de una secuencia de bases que especifica una secuencia de aminoácidos
- El ARN de transferencia (ARNt) es parte de la maquinaria estructural que descifra el código de ARNm. Portan aminoácidos específicos que son transferidos a un polipéptido nacente según las instrucciones contenidas en el ARNm.
- El ARN ribosómico (ARNr) forma un complejo con proteínas específicas para formar el ribosoma, que es el componente traduccional clave
-
- el ribosoma forma complejos con ARNm y dirige los ARNt apropiados y la síntesis del enlace polipeptídico.
Traducción:
Proceso mediante el cual se utiliza la información contenida dentro de un ARNm para dirigir la síntesis del polipéptido correspondiente.
El Código Genético
¿Cómo se almacena la información de una secuencia polipeptídica dentro de una molécula de ARNm? Hay veinte aminoácidos comunes diferentes, pero solo cuatro bases diferentes en el ARN (A, C, G y U).
|
Arreglo de Base |
Combinaciones Posibles |
|---|---|
|
1 |
4 1 =4 |
|
2 |
4 2 =16 |
|
3 |
4 3 =64 |
|
4 |
4 4 =256 |
Un arreglo de tripletes parecería ser la mínima combinación posible necesaria para codificar los 20 aminoácidos diferentes. Aunque, obviamente van a quedar muchos codones “sobrantes”. La mayoría de los aminoácidos están codificados por más de un solo triplete único y, por lo tanto, se dice que el código genético es degenerado.
Experimentos que condujeron a la solución del código genético:
Nirenberg y Matthei (1961): Nirenberg y Matthei trabajaron con extractos bacterianos que contenían todo lo necesario para la traducción, con excepción del ARNm. A esto le agregaron ARN poli A, poli U o poli C. Se determinaron las proteínas producidas por la traducción de estos ARN (el poli G no funcionó, probablemente debido a problemas conformacionales):
|
Poli U |
Poli A |
Poli C |
|---|---|---|
|
Phe |
Lys |
Pro |
Así, el triplete UUU = Phe, AAA = Lys, y CCC = Pro.
Korana (1963): En un sistema de extracto libre de células, Korana añadió ARNm con secuencias de nucleótidos repetidas. La secuencia... ACACACAC... resultó en un polipéptido con restos alternos de treonina e histidina. Pero, ¿la treonina estaba codificada por ACA y la histidina por CAC? ¿O vise versa? Para determinar la respuesta a esto, se intentó la secuencia de ARNm... AACAACAACAAC... Hubo tres marcos de lectura diferentes posibles para la traducción de este ARNm:
- AAC AAC
- ACA ACA ACA
- CAA CAA CAA
Pero el CAC no era un posible triplete. Se encontró que esta secuencia codificaba tres cadenas polipeptídicas diferentes: poli Asn, poli Thr y poli Gln. Dado que no se encontró histidina, la histidina fue codificada por el triplete CAC.
Nirenberg y Leder (1964): Nirenberg y Leder utilizaron un filtro que permitiría el paso de tripletes de ARN y ARNt cargados, pero evitaría el paso de ribosomas más grandes. Secuencias específicas de ARN triplete se unirían a ribosomas y causarían la unión de las moléculas de ARNt cargadas asociadas (codificadas por el triplete específico). En un experimento dado, si un ARNt cargado único se radiomarcaba (en el aminoácido), entonces se podría determinar si ese ARNt cargado particular estaba asociado por el triplete único. De esta manera, se determinaron los 61 codones para aminoácidos.
.png)
Figura 2.4.3: Experimento de Nirenberg y Leder
El código genético
|
5' Fin (Inicio) |
Segunda Posición |
Extremo 3' |
|||
|---|---|---|---|---|---|
|
|
U |
C |
A |
G |
|
|
U |
Phe |
Ser |
Tyr |
Cys |
U |
|
|
Phe |
Ser |
Tyr |
Cys |
C |
|
|
Leu |
Ser |
Detener |
Detener |
A |
|
|
Leu |
Ser |
Detener |
Trp |
G |
|
C |
Leu |
Pro |
Su |
Arg |
U |
|
|
Leu |
Pro |
Su |
Arg |
C |
|
|
Leu |
Pro |
Gln |
Arg |
A |
|
|
Leu |
Pro |
Gln |
Arg |
G |
|
A |
Ile |
Thr |
Asn |
Ser |
U |
|
|
Ile |
Thr |
Asn |
Ser |
C |
|
|
Ile |
Thr |
Lys |
Arg |
A |
|
|
Met (inicio) |
Thr |
Lys |
Arg |
G |
|
G |
Val |
Ala |
Asp |
Gly |
U |
|
|
Val |
Ala |
Asp |
Gly |
C |
|
|
Val |
Ala |
Glu |
Gly |
A |
|
|
Val |
Ala |
Glu |
Gly |
G |
Nota
Se indican las preferencias de codones de E. coli.
Todas las proteínas en procariotas y eucariotas comienzan la traducción con el codón iniciador AUG (metionina). Los tres codones, UAA, UGA y UAG son codones de terminación (no codifican ningún aminoácido sino señalan el final de la cadena proteica).
Obsérvese la aparente importancia relativa de la base media en el triplete de codones
|
U |
C |
A |
G |
|---|---|---|---|
|
Phe Leu Ile Met Val |
Ser Pro Thr Ala |
Tyr Su Gln Asn Lys Asp Glu Detener |
Cys Trp Arg Ser Gly Detener |
|
hidrofóbico |
Pequeño/Polar |
Cargado/Polar |
Polar |
¿Las arquitecturas comunes de proteínas pueden ser estampadas por un patrón cuaternario simple de residuos?
Los veinte aminoácidos comunes y sus siglas de tres letras y de una sola letra:
|
Aminoácido |
Acrónimos de tres letras |
Acrónico de una letra |
|---|---|---|
|
Alanina |
Ala |
A |
|
Cisteina |
Cys |
C |
|
Ácido aspártico |
Asp |
D |
|
Ácido glutámico |
Glu |
E |
|
Fenilalanina |
Phe |
F |
|
Glicina |
Gly |
G |
|
Histidina |
Su |
H |
|
Isoleucina |
Ile |
I |
|
Lisina |
Lys |
K |
|
Leucina |
Leu |
L |
|
Metionina |
Met |
M |
|
Asparagina |
Asn |
N |
|
Prolina |
Pro |
P |
|
Glutamina |
Gln |
Q |
|
Arginina |
Arg |
R |
|
Serina |
Ser |
S |
|
Treonina |
Thr |
T |
|
Valina |
Val |
V |
|
Triptófano |
Trp |
W |
|
Tirosina |
Tyr |
Y |


