11.1: Introducción
- Page ID
- 56174
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)El Dogma Central
El ADN fue descrito como una molécula compuesta por 2 hebras antiparalelas en una doble hélice por Francis Crick y James Watson. El elegante modelo ilustró la redundancia intrínseca que convirtió al ADN en un recipiente de almacenamiento de datos adecuado para la información genética. Francis Crick posteriormente planteó una noción de cómo esta información pasó del almacenamiento a un programa real que ejecuta celdas. Crick lo postuló primero como una “hipótesis de secuencia”. Esta idea de flujo de información se llama el Dogma Central de la Biología Molecular. El ADN almacena la información que se expresa como un mensaje intermedio de ARN. Este ARN se traduce luego en aminoácidos para producir proteínas.
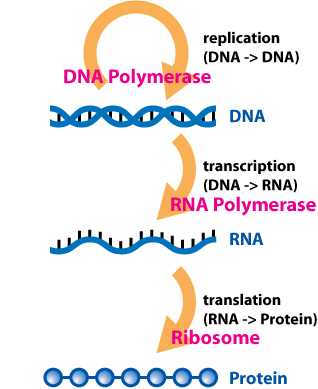
El flujo de información en las celdas. El ADN sirve como molde para copiarse a sí mismo (replicación). El ADN también puede servir como molde para el ARN (transcripción). El ARN se decodifica en aminoácidos para generar proteínas (traducción). Crédito Daniel Horspool (CC-BY-SA 3.0)
Transcripción
El ADN es simplemente un recipiente de almacenamiento de información genética. Se asienta en el núcleo y debe ser llamado a través de un proceso de transcripción donde una enzima llamada ARN polimerasa “lee en voz alta” la información almacenada en una molécula llamada ARN mensajero (ARNm). Dado que el ADN es bicatenario de manera antiparalela, automáticamente conocemos la secuencia de la segunda cadena al conocer la primera. El ARNm se elabora a través de apareamiento de bases complementarias a la cadena molde, que es el complemento inverso de la cadena codificante. La cadena codificante es la cadena que se lee de manera idéntica en secuencia al ARNm con las excepciones de que las T son reemplazadas por U.


Traducción

Esta cadena codificante es descodificada posteriormente por los ribosomas con la ayuda de ARNs de transferencia de ARNs) que actúan como decodificador del ensamblador de información y proteínas en un proceso llamado traducción. El ribosoma escanea a lo largo del ARNm y reconoce nucleótidos en lotes de 3. Estos lotes de 3 pueden traducirse en un aminoácido y se conocen como codones. Dado que hay 4 tipos de bases y se leen como grupos de 3, hay 4 3 (o 64) combinaciones de estos codones. Sin embargo, solo hay 20 aminoácidos que se utilizan para construir proteínas. Esto indica que hay espacio para la redundancia. Tres de estos codones le dicen al ribosoma que se detenga, como un periodo en una oración. A estos se les llama codones de parada. Hay un codón especial que realiza doble función: ATG. El codón (ATG) que codifica el aminoácido Metionina también actúa como codón de inicio que le dice al ribosoma de dónde empezar a leer. Al igual que los ácidos nucleicos, las proteínas tienen una polaridad y se sintetizan en una dirección amino a carboxilo. Abreviamos esto denominando el inicio de la secuencia de la proteína, N-terminal, y el final de la secuencia como el C-terminal.
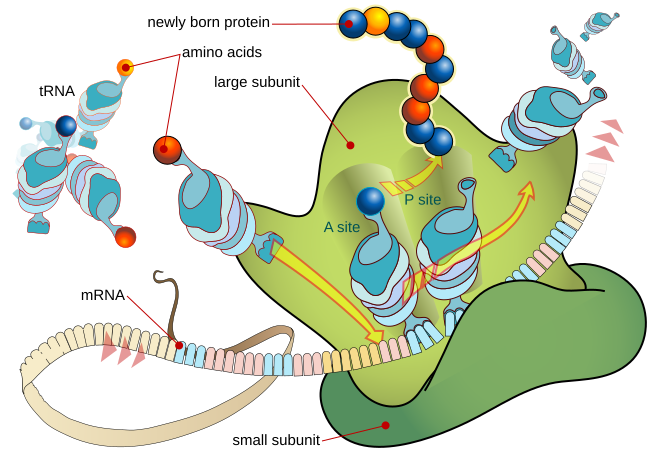
Los ribosomas son grandes complejos de enzimas que coordinan la decodificación del ARNm en aminoácidos para generar proteínas.

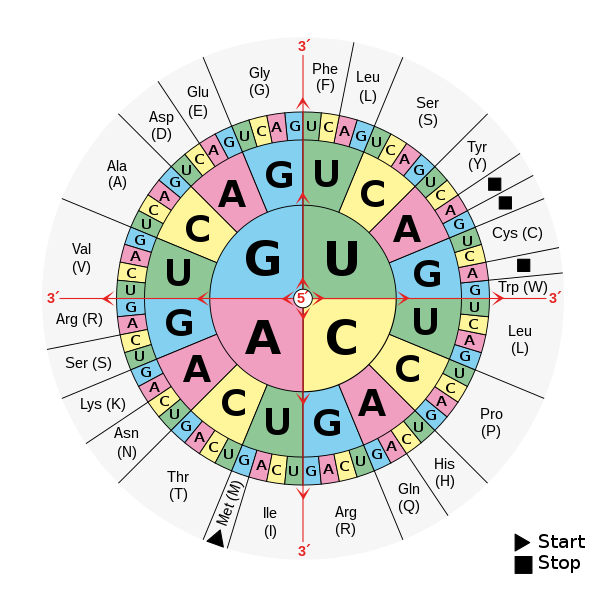
El código genético estándar.
Video Avanzado de Traducción
Decisiones... decisiones...
¿Qué tipo de decisiones se toman para que las células madre se diferencien en diferentes tipos de células? ¿Qué tipos de regulación ocurren durante este proceso?

Un grupo de células progenitoras neuronales (neuroesfera) se disocia y se diferencia en neuronas.
Reseña de video
- Simulación de Transcripción (Licencia MIT)
- Transcripción (CC-BY-NC-ND Cold Spring Harbor Lab — Centro de Aprendizaje de ADN)
https://www.dnalc.org/content/c15/15510/transcription_basic.mp4 - Traducción (CC-BY-NC-ND Cold Spring Harbor Lab — Centro de Aprendizaje de ADN)
https://www.dnalc.org/content/c15/15501/translation_basic.mp4



