13.1: Introducción
- Page ID
- 56308
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Criptozoología
La criptozoología es una pseudociencia centrada en la descripción de animales que tienen poca o ninguna evidencia de existencia. Estas bestias míticas incluyen Bigfoot, Yeti, Sasquatch, jackalope, Loch Ness Monster y chupacabras. Existen pocas pruebas que ilustren su existencia aparte del folclore.
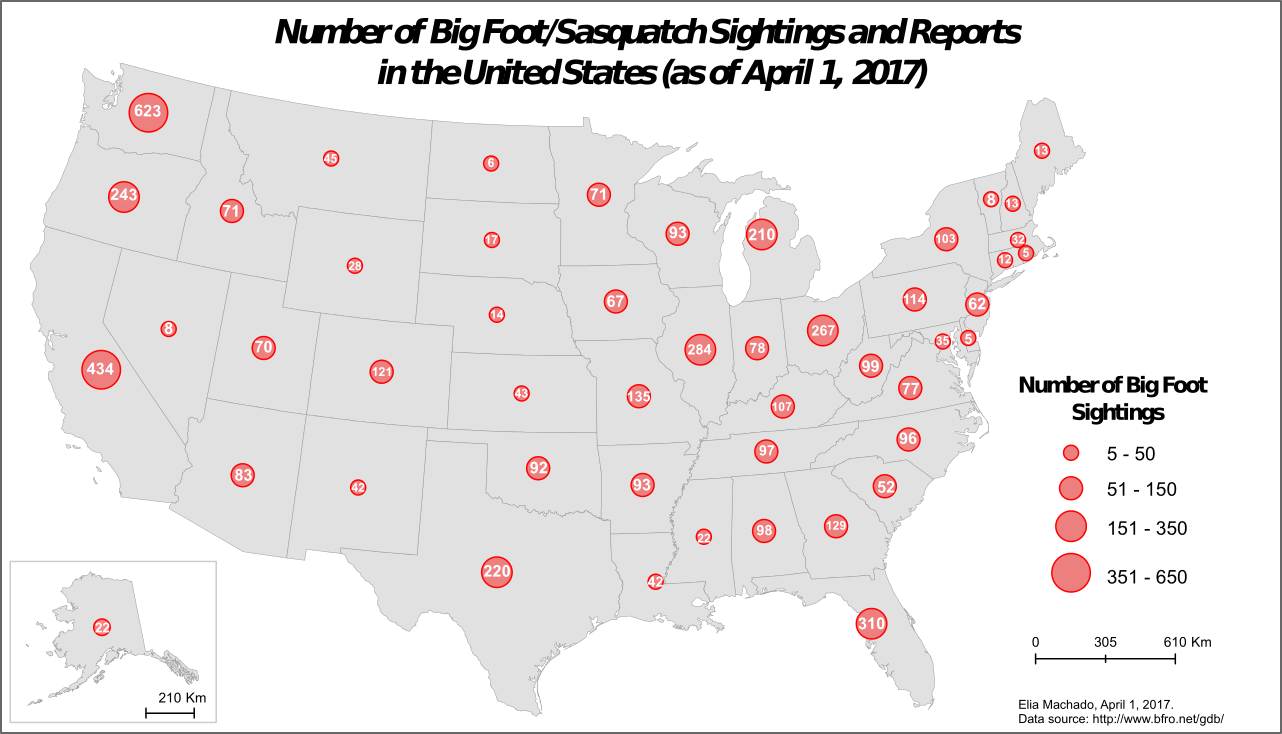
Avistamientos de Bigfoot en América del Norte. Crédito: Elia Machado (CC-BY-NC-SA)
En ocasiones, la evidencia física se deja atrás como el pelo o las heces. Con evidencia de ADN, podemos ayudar a confirmar la existencia de estas criaturas desconocidas. A continuación se presenta una tabla de Sykes et al. mostrando resultados sobre supuestos simios crípticos (Bigfoot/Yeti) y qué evidencia de ADN ha revelado que son.
Muestras criptozoológicas de pelo que se cree que surgen de animales legendarios como Bigfoot, Sasquatch, Yeti, etc.
Tabla tomada de http://rspb.royalsocietypublishing.org/content/281/1789/20140161 CC-BY
La necesidad de códigos de barras
La taxonomía de los seres vivos fue creada por Carl von Linné, quien la formalizó mediante el uso de un sistema de clasificación binomial para diferenciar organismos. Se utilizó la nomenclatura binomial para describir un género y un nombre de especie a cada organismo para proporcionar una identidad. En estos días, las clasificaciones de organismos son cada vez más importantes como medida de la diversidad ante la destrucción del hábitat y el cambio climático global. No hay consenso sobre cuántas formas de vida existen en este planeta, pero la estimación de las tasas de extinción es de aproximadamente 1 especie por cada 100-1000 millones de especies. La clasificación en el día de Linné se realizó principalmente por diferencias morfológicas. Esto se llevó a cabo en fósiles. Sin embargo, la morfología tiene muchos inconvenientes, especialmente en especies sexualmente dimórficas o especies con múltiples morfologías de desarrollo.

Larva (arriba) del Encajado Verde y el adulto (abajo)
La biología molecular y las tecnologías de ADN han revolucionado el sistema de clasificación de los seres vivos especialmente al proporcionar la capacidad de igualar la relación de estas especies. El código de barras de ADN, como su nombre lo indica, busca utilizar marcadores de ADN para identificar diferencialmente organismos. Pero, ¿qué marcadores de ADN se deben usar? ¿Qué criterios utilizamos para desarrollar códigos de barras? Discriminación, Universalidad y Robustez son los criterios utilizados para definir la utilidad de los códigos de barras.

Dado que el objetivo del código de barras es definir organismos específicos, la discriminación es el objetivo principal. La discriminación se refiere a la diferencia de secuencias que ocurren entre especies. Sin embargo, la ciencia es más fácil cuando hay cierta universalidad en el locus utilizado para la discriminación. Como suena, la universalidad es un intento de usar el mismo locus en genomas dispares. Si bien la discriminación se refiere a la singularidad de las secuencias, la universalidad busca utilizar un solo conjunto de cebadores de PCR que puedan amplificar esa misma región distinta con similitud de secuencia variable. Si alguna región del ADN no tiene absolutamente ninguna desviación de secuencia entre especies, ésta tiene gran universalidad pero escasa discriminación. Pero si una secuencia tiene una similitud de secuencia muy baja, esto es genial para la discriminación pero no tiene absolutamente ninguna universalidad y no puede amplificarse con el mismo conjunto de cebadores. La robustez se refiere a la fiabilidad de la amplificación por PCR de una región. Algunas regiones del ADN simplemente no se amplifican bien o es demasiado difícil diseñar cebadores apropiados y únicos para ese locus.

Un caso donde hay universalidad para diseñar imprimaciones, pero no un área donde pueda darse discriminación.
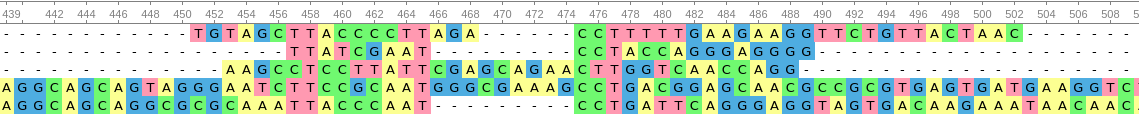
Si bien la discriminación de diferentes organismos puede ocurrir en esta situación, la falta de similitud en la secuencia dificultaría el diseño de cebadores. Es decir, la falta de universalidad en secuencia también haría que esta PCR no fuera robusta.

La suficiente variabilidad en estas secuencias nos da la capacidad de discriminar entre especies. La alta similitud nos proporciona la universalidad requerida para diseñar cebadores que puedan ser lo suficientemente robustos como para amplificarse por PCR.

En ocasiones, las especies son tan similares para una secuencia que se requiere un segundo marcador. Así como el código de barras estándar UPC tiene una serie de líneas verticales de diferente espaciado y ancho, un código de barras bidimensional agrega esa segunda dimensión de información en un cuadrado de puntos como en un código QR (código de respuesta rápida). También podemos utilizar el segundo, el tercero o el cuarto conjunto de loci que ayudarán a aumentar la discriminación, así como CoDIS utiliza múltiples sitios STR para definir a personas individuales. En los animales, el código de barras más utilizado es el gen mitocondrial, Citocromo Oxidasa I (COI). Dado que todos los animales tienen mitocondrias y tienen este gen mitocondrial, ofrece una alta universalidad. Es un locus robusto que es fácil de amplificar y tiene un alto número de copias con suficiente desviación de secuencia entre especies para discriminar entre ellas.
Los genomas mitocondriales animales varían de 16kb-22kb. Sin embargo, las plantas, los hongos y los protistas tienen genomas mitocondriales muy diferentes y más grandes. Para las plantas, utilizamos un gen cloroplasto, subunidad grande de ribulosa-bisfosfato carboxilasa (rbCl) o maturasa K (mATK) (Hollingsworth et al. 2011). Los procariotas a menudo son discriminados por su gen 16s rRNA mientras que los eucariotas pueden ser identificados por 18s rRNA. El COI (un gen de transmisión maternal) no creará una imagen clara de la identidad de las especies en el caso de animales híbridos (mulas, ligers, coydogs, etc.). En ocasiones, las especies estrechamente relacionadas también son indistinguibles por un solo código de barras, por lo que la inclusión de 18s con COI puede ser necesaria para definir la identidad de la especie. Dado que es muy difícil cumplir con los tres criterios (robustez, universalidad y discriminación) para todas las especies, es importante contar con estos múltiples códigos de barras. Los hongos demuestran ser difíciles en la identificación por COI, por lo que se utiliza otro marcador llamado espaciador interno transcrito (ITS) para ayudar en su identificación. También hay que recordar que no todo lo que tiene cloroplastos son plantas y, por lo tanto, se utilizan marcadores adicionales para identificar a los protistas.
Mezclas de Organismos

Los líquenes son organismos compuestos compuestos por cianobacterias u otras algas con hongos. En este caso, un solo código de barras identificaría incorrectamente a la especie.

Los gránulos de kéfir representan colonias de microbios mixtos que se utilizan para generar kéfir. Crédito: A. Kniesel (CC-BY-SA 3.0)

Se utiliza una colonia simbiótica de bacterias y levaduras para fermentar la kombucha. Como su nombre lo indica, se trata de una compleja colonia compuesta de múltiples especies que contribuyen a las cualidades de la kombucha. Crédito: Lukas Chin (CC-BY-SA 4.0)
Metabarcodificación y microbiomas
Resultados de Clase
Los alumnos quisieron revisar algunos alimentos. Estos incluían salchicha de desayuno de un carrito Halal, “cecina de ternera” de la máquina expendedora, rosbif de la cafetería y salchicha china (lopcheng).

Para obtener más resultados de clase, visite https://openlab.citytech.cuny.edu/dna-barcodes/
Recursos adicionales
- Secuencias de Referencia
- Sykes BC, Mullis RA, Hagenmuller C, Melton TW, Sartori M. Análisis genético de muestras de cabello atribuidas a yeti, bigfoot y otros primates anómalos. Proc Biol Sci. 2014 22 ago; 281 (1789) :20140161. doi: 10.1098/rspb.2014.0161.
- Hollingsworth PM, Graham SW, Little DP (2011) Elegir y usar un código de barras de ADN vegetal. PLOS UNO 6 (5): e19254. https://doi.org/10.1371/journal.pone.0019254
- http://www.dnabarcoding101.org/