1.3: Mutaciones de ADN
- Page ID
- 56607
- Enumere los tipos de mutación.
- Predecir los efectos de mutaciones específicas en regiones codificadoras de genes sobre la proteína codificada por el gen.
- Describir las formas en que surgen las mutaciones, natural y experimentalmente.
Introducción
La mejora de los cultivos se basa en la variación genética, que es sinónimo de variación del ADN. Las mutaciones son la fuente de variación genética que es el motor para la mejora de los rasgos. En general, las mutaciones son eventos raros, y la mayoría son perjudiciales y se seleccionan en contra durante el curso de la evolución. Sin embargo, ocasionalmente las mutaciones crean variación genética que es beneficiosa. Como tal, a menudo es de interés para el mejoramiento de plantas o para estudios genéticos para inducir mutaciones genéticas al exponer semillas o tejido vegetal a ciertos agentes, llamados mutágenos.
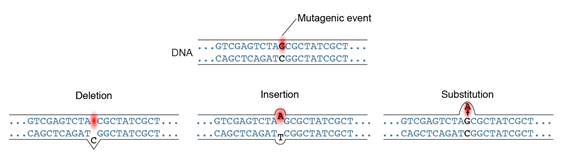
Por definición, una mutación es un cambio heredable en la secuencia de ADN. Esto puede ocurrir de varias maneras: sustitución de una base de ADN, inserción o deleción de una o más bases de ADN, o mediante reordenamientos cromosómicos a gran escala, estos últimos de los cuales no serán considerados aquí. También ha quedado claro en los últimos años que la cromatina lleva información adicional a la secuencia de ADN en forma de modificaciones de bases como la metilación de citosina. Esto se conoce como modificación epigenética y puede afectar la función génica y los rasgos de la planta.
A. Sustituciones de bases de ADN
El tipo de mutación más simple es la sustitución de una base por otra en la secuencia de ADN. Las sustituciones suelen surgir como errores durante la replicación o reparación del ADN. El tipo más común es la transición, donde una pirimidina puede ser sustituida por la otra, o una purina por la otra. El tipo menos común es la transversión, en la que una purina puede ser sustituida por una pirimidina o viceversa. Como se describe en la siguiente sección, estos pueden tener diversos efectos sobre la función génica dependiendo de dónde ocurran en un gen.
B. Inserciones y eliminaciones

Las inserciones o deleciones de ADN son otro tipo de mutaciones que ocurren con bastante frecuencia. Pueden variar en tamaño de una a miles o más bases nucleotídicas. Sus efectos sobre la función génica dependen del tamaño de la inserción o deleción, y de la ubicación relativa al gen. Como se describe a continuación, las inserciones y deleciones que están en múltiplos de 3 se denominan inserciones y deleciones en marco y dan como resultado la adición o deleción de aminoácidos de la secuencia de la proteína. Cuando no están en múltiplos de tres, las inserciones y deleciones conducen a mutaciones de cambio de marco, alterando la secuencia de aminoácidos de la proteína codificada por el gen mutado.

Una fuente común de ADN insertado es de transposones, secuencias de ADN que son capaces de moverse de un sitio genómico e insertarse en otro. Los transposones fueron descubiertos en maíz por Barbara McClintock. Por este descubrimiento, fue galardonada con el Premio Nobel de Fisiología y Medicina en 1983.
Algunos transposones se copian y se multiplican en número dentro de un genoma. Los transposones pueden causar reordenamientos cromosómicos a gran escala y mutaciones genéticas. Una característica de las mutaciones causadas por muchos transposones vegetales es que son inestables. La escisión de un transposón del gen mutado a menudo puede restaurarlo a tipo salvaje o crear un nuevo alelo mutante estable. Los transposones se encuentran en todos los organismos vivos estudiados y son una fuerza impulsora importante en la evolución del genoma.

Efectos de las mutaciones en la función génica
A. Mutaciones en la región codificante
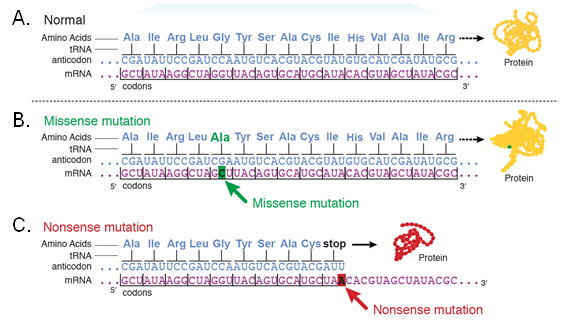
Mutación silenciosa
Una mutación silenciosa es una mutación que da como resultado el cambio de un codón sin un cambio en el aminoácido representado por el codón. Debido a la redundancia del código genético, las sustituciones de bases de ADN pueden no conducir a la incorporación de un aminoácido incorrecto en la proteína.
Mutación sin sentido (sustitución de aminoácidos)
Una mutación sin sentido es una sustitución de base de un solo nucleótido que altera un codón de tal manera que codifica para un aminoácido diferente que se incorpora en la proteína codificada. La sustitución de aminoácidos puede afectar la función de la proteína si ocurre en una porción crítica de la proteína.
Mutación de cambio de marco
Debido a la naturaleza triplete del código genético, una inserción o deleción puede cambiar el marco de lectura para toda la secuencia posterior. Por ejemplo, si una secuencia particular se lee secuencialmente (Figura 4).
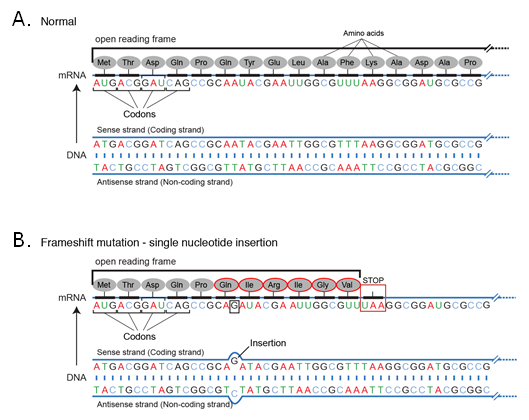
Debido a que la nueva secuencia de codones es diferente de la original, toda la secuencia de aminoácidos se cambia desde el punto de inserción. Se observa un efecto similar para las eliminaciones. Este efecto se observa siempre que el número de bases nucleotídicas insertadas o suprimidas no sea un múltiplo de 3 y por lo general resultará en la pérdida de la función de la proteína.
Adiciones o deleciones de aminoácidos
Por otro lado, si las inserciones o deleciones están en múltiplos de 3, pueden surgir adiciones o deleciones de aminoácidos. Considere la siguiente inserción de 3 bases (XXX):
UUU CCC XXX AAA GGG (codones)
aa1 aa2 aa5 aa3 aa4 (aminoácidos)
Si tales inserciones o deleciones ocurren en regiones importantes de una proteína, podrían afectar la función.
Mutación sin sentido
Una mutación sin sentido surge cuando un codón funcional se cambia a un codón de terminación. Las mutaciones sin sentido pueden causar la terminación prematura de la traducción.
B. Mutaciones en la región reguladora

Promotores y potenciadores controlan aspectos temporales, espaciales y cuantitativos de la expresión génica. Como tal, las mutaciones en estas regiones reguladoras de un gen pueden alterar la regulación de la expresión del gen. Los genes pueden expresarse en momentos, lugares o niveles inapropiados, lo que puede afectar el fenotipo de una planta.
Teosinte, el progenitor del maíz moderno, produce múltiples ramas (Figura 6A) debido a la disminución de la dominancia apical. En teosinte y maíz moderno, la dominancia apical está bajo el control de un gen llamado teosinte ramificado1, tb1 (Doebley et al. 1997). Una inserción de un transposón en la región reguladora del gen tb1 en el maíz moderno provoca que el gen se exprese fuertemente (Doebley et al. 1997; Studer et al. 2011), lo que resulta en un mayor dominio apical y supresión de ramificaciones excesivas (Figura 6B).
C. Mutaciones recesivas vs. Dominantes
Mutaciones recesivas
Las mutaciones recesivas suelen ser el resultado de una pérdida de función. Una mutación de pérdida de función provoca una disminución en la función de un gen. Una pérdida completa de la función se conoce como una mutación nula, mientras que la pérdida parcial de los mutantes de función a menudo se conoce como hipomorfos o “fugas”. Las mutaciones de pérdida de función pueden afectar la expresión génica o la función de la proteína resultante. La mayoría de las mutaciones de pérdida de función son recesivas porque en un heterocigoto, el alelo normal puede proporcionar la función necesaria.
Mutaciones dominantes
Las mutaciones dominantes pueden afectar a los genes de varias maneras diferentes. Una clase general es la ganancia de mutaciones de función. Estos abarcan varios tipos diferentes.
- Mutantes de sobreexpresión. Como su nombre lo indica, el gen afectado se expresa en niveles que son inapropiadamente altos, provocando que se produzca demasiado producto génico.
- Las mutaciones neomórficas surgen cuando una mutación provoca un efecto cualitativamente nuevo no visto con alelos normales. Esto puede ocurrir cuando una mutación afecta la regulación de un gen de tal manera que se expresa en un nuevo tiempo o lugar en la planta. También puede ocurrir cuando una mutación altera las propiedades de la proteína codificada. Por ejemplo, un factor de transcripción podría mutarse para reconocer una secuencia promotora diferente, o una enzima podría reconocer un nuevo sustrato
- Las mutaciones negativas dominantes alteran un producto génico de tal manera que el producto génico mutante interfiere con la función del normal. Esto puede ocurrir a nivel de proteína. Por ejemplo, una proteína podría funcionar como un complejo multimérico y la presencia de una subunidad mutante podría comprometer la función de todo el complejo. Los negativos dominantes también pueden ocurrir a nivel de ARN, por ejemplo, si una inserción, deleción o reordenamiento hace que se transcriba la cadena incorrecta, creando un efecto antisentido o de supresión de ARNi.
Efectos de las mutaciones sobre los rasgos
Ejemplo de una mutación de pérdida de función: la función génica es necesaria para un rasgo
La dulzura es un rasgo importante de calidad en el maíz (maíz). Uno de los mutantes de pérdida de función utilizados para mejorar el dulzor en el maíz es sugary1 (su1). El gen Su1 se expresa en el endospermo (Lertrat y Pulam, 2007), y codifica una enzima que se requiere para la biosíntesis normal del almidón (Rahman et al. 1998). En el mutante su1, con biosíntesis defectuosa del almidón, la concentración de azúcar es 3 veces mayor que la del tipo silvestre, lo que hace que este mutante sea valioso para el maíz dulce (Lertrat y Pulam, 2007). Además, los granos su1 acumulan un polisacárido altamente ramificado llamado fitoglicógeno, lo que le da a los granos una textura cremosa. Se desarrollan nuevos híbridos comerciales de maíz dulce utilizando una combinación de mutantes de biosíntesis de almidón (Nelson y Pan, 1995; Lertrat y Pulam, 2007).
Ejemplo de una mutación de ganancia de función: la función génica es suficiente para un rasgo

La hormona del crecimiento vegetal, giberelina (GA) regula la altura de la planta, por lo tanto, una planta que no produce GA, o no responde a GA tendrá un fenotipo enano (Figura 7). A nivel molecular, la respuesta celular a GA involucra varios procesos, incluyendo la destrucción de proteínas represoras, denominadas DELA.
El “gen de la revolución verde” Altura reducida-1 (Rht-1) del trigo codifica un represor DELLA de señalización de GA (Peng et al. 1999). Se requiere un tramo de cinco aminoácidos denominado dominio DELLA para la destrucción del represor en respuesta a GA. Las mutaciones en el dominio DELLA de los genes Rht-1 dan como resultado un producto proteico que es resistente a la degradación, lo que lleva a que no se señale una respuesta de GA. La falta de señal de la presencia de GA en los mutantes rht-1 es responsable de su baja estatura. Cuando el gen rht-1 mutante se transforma en arroz, el resultado también son plantas cortas, lo que demuestra que la nueva función de la proteína DELLA es suficiente para inducir enanismo (Peng et al. 1999).
Generación de mutaciones
Debido a que las mutaciones son la última fuente de variación genética para la reproducción y los estudios genéticos, a veces es de interés generar nuevas mutaciones. Nuevas mutaciones de ADN (Figura 8) pueden surgir espontáneamente o pueden ser inducidas por métodos experimentales.
Las mutaciones espontáneas surgen “naturalmente”
Es decir, no se hizo ningún esfuerzo particular por parte del científico para aumentar la tasa de mutación. Las mutaciones espontáneas generalmente ocurren a tasas muy bajas, del orden de 1 por gen por millón de gametos.
Errores en la replicación del ADN
Muy raramente, se incorporan bases incorrectas o se omiten bases de una cadena de ADN durante la síntesis. Tales errores pueden conducir a sustituciones, inserciones o deleciones espontáneas. Por ejemplo, el deslizamiento de la hebra debido a la formación de un bucle.
Mutágenos ambientales
Existen varios agentes químicos presentes en el ambiente con potencial de dañar el ADN y crear mutaciones. Estos incluyen compuestos naturales, así como contaminantes artificiales. Algunos modifican las bases nucleotídicas del ADN a través de la desaminación (por ejemplo, ácido nitroso), mientras que otros promueven reacciones oxidativas que pueden dañar el ADN (por ejemplo, ozono).
La radiación es otro tipo de mutágeno ambiental. La radiación ionizante (por ejemplo, rayos X) puede romper secuencias de ADN y promover reordenamientos cromosómicos. Los rayos ultravioleta menos potentes pueden penetrar en la célula y promover la formación de dímeros de pirimidina que pueden inhibir la replicación y transcripción.
La mutagénesis experimental implica aplicar un agente mutagénico a una planta o parte de la planta para generar una mutación. Para que la mutación sea útil, debe ser heredable, lo que impone limitaciones a qué tejidos se pueden tratar. Después del tratamiento mutagénico, se debe implementar un esquema de reproducción posterior para generar líneas de reproducción puras de genotipos apropiados para mostrar fenotipos mutantes. Estos dependerán de la especie y del diseño de la mutagénesis.
Mutagénesis química
Un mutágeno químico de uso común para la biología experimental de plantas y el mejoramiento de mutaciones es el metanosulfonato de etilo (EMS). El compuesto se conoce como agente alquilante e induce una transición de CG a TA.
Mutagénesis de semillas
Este es el método más común de mutagénesis química en la mayoría de las especies. Las semillas se remojan en solución EMS, se plantan y se dejan florecer y sembrar la semilla. Durante la mutagénesis EMS, cada célula del embrión será mutagenizada de forma independiente y la semilla resultante y la planta posterior serán quiméricas. Estas semillas se pueden sembrar y las plantas se pueden cribar para detectar rasgos de interés. Para las especies autopolinizadoras, la reproducción requerida es muy simple, mientras que para las especies dioicas sería mucho más elaborada e intensiva en mano de obra.
Mutágenesia Polen
Para algunas especies como el maíz, la mutagénesis de polen es el método de elección. El polen se trata con EMS y se aplica a las sedas de una planta aceptora. El beneficio es que la semilla resultante no es quimérica sino que todas las células del embrión portan el mismo genoma paterno mutagenizado heredado del grano de polen.
Mutagénesis insercional
La idea detrás de la mutagénesis insercional es que una pieza de ADN móvil o introducida a veces puede insertarse en un gen, inactivando o modificando así la función del gen. El enfoque es útil en la clonación de genes porque el ADN insertado sirve como una “etiqueta” que puede usarse para localizar el gen de interés, y posteriormente aislar ese gen.
Otra estrategia utilizada por los investigadores es la mutagénesis con ADN-T del plásmido Ti de Agrobacterium tumefaciens. Un mayor número de plantas (hasta 10,000 para Arabidopsis) se transforman con ADN-T para generar mutantes al azar.
Similar al ejemplo de mutagénesis de transposón anterior, un gen puede ser “etiquetado” por un ADN-T y la etiqueta utilizada para aislar el gen. Sin embargo, las inserciones de ADN-T son estables y, por lo tanto, alteran el gen permanentemente.
En los últimos años, los mutágenos insercionales se han diseñado de muchas maneras innovadoras. Por ejemplo, se han diseñado promotores fuertes en transposones de tal manera que si el transposón se inserta cerca de un gen, puede causar una fuerte expresión transcripcional de ese gen. Tales enfoques de “etiquetado de activación” han tenido éxito en la identificación de muchos genes que no eran susceptibles a otros enfoques mutacionales.
Las mutaciones alteran los pares de bases A-T y G-C en el ADN. Una mutación en una secuencia codificante puede alterar la secuencia y función del producto proteico. Una mutación de cambio de marco cambia el marco de lectura a través de inserciones o deleciones para producir un producto completamente novedoso. Una mutación puntual por otro lado altera solo el aminoácido representado por el codón en el que existe la mutación. Las mutaciones en regiones reguladoras podrían alterar la expresión de un gen. Las mutaciones han sido herramientas poderosas para estudios genéticos así como para estudiar funciones biológicas, por ejemplo, crecimiento y desarrollo. La ocurrencia natural de mutaciones puede potenciarse experimentalmente aplicando agentes denominados mutágenos. Los investigadores han utilizado ciertos mutágenos para inducir mutaciones para estudiar la función génica y aplicar nuevos rasgos para la mejora de los cultivos.
Actividad 1
Para esta actividad, puede ser útil referirse a la tabla de códigos genéticos.
Para la siguiente secuencia de ADN:
5′ ATG TTG GAG AAG GTT GAA ACT TTC 3′
Escribir la cadena de codificación
3′ TAC AAG CTC TTC CAA CTT TGA AAG 5′
Escribir la secuencia de ARNm a partir de la secuencia de ADN dada
AUG UUC GAG AAG GUU GAA ACU UUG
Escribir la secuencia peptídica resultante
Met-Phe-Glu-Lys-Val-Glu-Thr-Leu
Si el tercer triplete de la secuencia de ADN original se muta a GA A para dar como resultado la siguiente secuencia
5′ ATG TTG GAA AAG GTT GAA ACT TTC 3′
¿Cuál será la secuencia de ARNm de la secuencia de ADN mutada?
AUG UUC GAA AAG GUU GAA ACU UUG
¿Cuál será la secuencia peptídica de la secuencia de ADN mutada?
Met-Phe-Glu-Lys-Val-Glu-Thr-Leu
¿Qué tipo de mutación de región codificante es esta? Da una razón para tu respuesta.
- Silencioso
- Sustitución de aminoácidos
- Cambio de marco
- Tonterías
Si el sexto triplete de la secuencia de ADN original se muta a T AA para dar como resultado la siguiente secuencia
5′ ATG TTG GAG AAG GTT TAA ACT TTC 3′
¿Cuál será la secuencia de ARNm de la secuencia de ADN mutada?
AUG UUC GAG AAG GUU UAA ACU UUG
¿Cuál será la secuencia peptídica de la secuencia de ADN mutada?
Met-Phe-Glu-Lys-Val
¿Qué tipo de mutación de región codificante es esta? Da razón para tu respuesta.
- Silencioso
- Sustitución de aminoácidos
- Cambio de marco
- Tonterías
La secuencia de ADN original se muta para dar como resultado la siguiente secuencia
5′ ATG TTG GAG AAG GTT CGA AAC TTT C 3′
¿Cuál será la secuencia de ARNm de la secuencia de ADN mutada?
AUG UUC GAG AAG GUU CGA AAC UUU C
¿Cuál será la secuencia peptídica de la secuencia de ADN mutada?
Met-Phe-Glu-Lys-Val-Arg-Asn-Phe
¿Qué tipo de mutación de región codificante es esta? Da una razón para tu respuesta.
- Silencioso
- Sustitución de aminoácidos
- Cambio de marco
- Tonterías
1. Un gen vegetal hipotético codifica una proteína con la siguiente secuencia de aminoácidos:
Met-Leu-Lys-Thr-Phe-Val-Glu
Una mutación de un solo nucleótido altera la secuencia de aminoácidos a:
Met-Tyr-Asp-Pro-Gly-Ala-Lys-Ile-Arg
Describir el tipo de mutación y la posición del codón que se ve afectado.
2. Para la siguiente secuencia de ADN
3′ A TG GCC GGC AAT CAA CTA TAT TG A 5′
1 24 (número de nucleótidos)
a. Escribir la cadena de codificación
b. Escribir la cadena de ARNm
c. Escribir la cadena proteica
d. Dar la secuencia de aminoácidos alterada de la proteína para cada una de las siguientes mutaciones:
i. Una transición en el nucleótido 11
ii. Una transición en el nucleótido 13
iii. Una deleción de un solo nucleótido en el nucleótido 7
iv. Un cambio de T a A en el nucleótido 15
v. Una adición de ACC después del nucleótido 6
vi. Una transición en el nucleótido 9
Referencias
Doebley et al. 1997. La evolución de la dominancia apical en el maíz. Naturaleza 386:485-488.
Lertrat, K., Pulam, T. 2007. Cría para aumentar la dulzura en maíz dulce. Int. J. Planta. Raza. 1:27-30.
Nelson, O., Pan, D. 1995. Síntesis de almidón en endospermas de maíz. Annú. Rev. Fisiol Vegetal. Planta Mol. Biol. 46:475-496.
Peng, J., Richards, D.E., Hartley, N.M., Murphy, G.P., Devos, K.M., Flintman, J.E., Beales, J., Fish, L.J., Worland, A.J., Pelica, F., Sudhakar, D., Christou, P., Snape, J.W., Gale, M.D., Harberd, N.D. P. 1999. Los genes de la “revolución verde” codifican moduladores de respuesta de giberelina mutante. Naturaleza 400:256-261.


