1.4: PCR y electroforesis en gel
- Page ID
- 56627
Al finalizar esta lección de PCR, los alumnos podrán:
- Enumere los 5 componentes químicos de una reacción PCR y describa sus roles.
- Enumere las funciones de los 3 ciclos de temperatura que se repiten durante una reacción de PCR.
- Describir el proceso de observación de resultados e interpretación de los resultados de un experimento de PCR.
- Enumerar los posibles usos de la PCR en pruebas genéticas y en investigación.
Visión general
La técnica de laboratorio de reacción en cadena de la polimerasa se utiliza en una variedad de aplicaciones para hacer copias de una secuencia de ADN específica. Esta lección describe cómo funciona una reacción PCR, lo que logra y sus requisitos básicos para el éxito. Se dan ejemplos de resultados de interpretación. Se explican las fortalezas, debilidades y aplicaciones de PCR a la biotecnología vegetal.
El descubrimiento de la PCR
En 1983, Kary Mullis conducía por una carretera de montaña californiana a altas horas de la noche. Como biólogo molecular, el Dr. Mullis estaba imaginando una mejor manera de estudiar el ADN. Este pensamiento nocturno condujo a una forma revolucionaria de hacer copias de laboratorio de moléculas de ADN (Saiki et al. 1985, Mullis 1990). En las décadas posteriores, la reacción en cadena de la polimerasa o PCR, se ha convertido en el método estándar utilizado para detectar secuencias específicas de ADN o ARN. La venta de equipos y kits de reactivos para PCR es un negocio multimillonario porque la detección de ADN y ARN es información crítica en muchas aplicaciones.
Replicación in vitro vs. in vivo
La PCR es un proceso In Vitro; una serie de reacciones químicas que ocurren fuera de una célula viva. Esta técnica de laboratorio se modela a partir de un proceso in vivo, la capacidad natural de la célula viva para replicar el ADN durante los ciclos celulares normales (ver Lección sobre ADN: El material genético). Cada célula viva hace una copia duplicada de cada cromosoma antes de que la célula esté lista para dividirse. La Figura 1 a continuación ilustra las partes clave de la replicación de ADN in vivo que son la base para el éxito de la PCR.
Existen otras enzimas que juegan un papel importante en la replicación in vivo. Sin embargo, la PCR funciona como un proceso de replicación de ADN in vitro al usar solo una de estas enzimas. Mullis imaginó un reactivo químico y un paso de cambio de temperatura en el método que pudiera realizar el trabajo de las otras dos enzimas. Cabe señalar que debido a que el Dr. Kerry Mullis había aprendido sobre los detalles de la replicación in vivo del ADN, pudo crear esta ciencia cambiando el método in vitro.
Antes de leer la descripción de los componentes y procesos de PCR, ver este video puede ayudarte a visualizar la importancia de cada paso,
Nombre y Componentes Químicos de la PCR
El nombre 'reacción en cadena de la polimerasa' representa la naturaleza del proceso. 'Polimerasa' porque la ADN Polimerasa III es necesaria para construir nuevas cadenas de ADN, al igual que en una célula viva. 'Reacción en cadena' describe ciclos repetitivos de replicación que se dirigen a un segmento específico de un cromosoma y utilizan una progresión de “copia de las copias” en cada ciclo que duplica la cantidad de copias de ADN de un segmento específico de ADN presente en cada ciclo. En tan solo 20 ciclos de la reacción en cadena, se pueden producir más de un millón (2 20) copias de ese segmento específico de ADN. Esto es suficiente ADN para ver a simple vista. El objetivo de la PCR es hacer millones de copias de un segmento específico de ADN que todos se originan a partir de una sola muestra de ADN.
Los cinco componentes químicos que deben agregarse a un tubo de ensayo para que funcione la reacción PCR, incluyen un molde de ADN, enzima ADN polimerasa III, cebadores de ADN monocatenarios, nucleótidos y tampón de reacción.
- El molde de ADN es una muestra de ADN que contiene la secuencia diana de ADN para copiar.
- ADN pol III. Hay dos requisitos para una enzima ADN polimerasa adecuada para PCR. Primero, la enzima debe tener una buena tasa de actividad alrededor de 75°C; en segundo lugar, la enzima debe ser capaz de soportar temperaturas de 95-100°C sin desnaturalizar y perder actividad.
- Dos imprimaciones. Los cebadores son oligonucleótidos cortos de ADN, generalmente de alrededor de 20 pares de bases de longitud. Debido a que el propósito de la PCR es amplificar una sección específica de ADN en el genoma, como un gen conocido, entonces deben usarse cebadores de secuencias específicas. El genetista que planea la reacción PCR diseñará un cebador directo para unirse a una cadena y un cebador inverso que complementa y se une a la otra cadena. El proceso de diseño de cebadores para seleccionar cebadores directos e inversos requiere un pensamiento genético apropiado y se describe más adelante en esta lectura.
- Los cuatro desoxirribonucleótidos trifosfatos (dNTPs) diferentes. La adenina (A), la guanina (G), la citosina (C) y la timina (T) son necesarias para proporcionar los bloques de construcción para la replicación del ADN. La ADN polimerasa agregará cada base complementaria a la nueva cadena de ADN en crecimiento de acuerdo con la secuencia de la cadena original siguiendo emparejamientos normales A-T y C-G.
- Finalmente, un tampón de reacción. Esto crea un pH estable y proporciona el cofactor Mg 2 + necesario para la actividad de ADN pol III.
Tres ciclos de temperatura
Una visión clave para el éxito de la PCR como un proceso de replicación de ADN in vitro que genera millones de copias de secuencias específicas fue un ciclo de tres temperaturas que logra tres partes de replicación del ADN: desnaturalización del molde bicatenario, reasociación de los cebadores con el hebras simples y extensión de la síntesis de nuevas cadenas por ADN pol III.
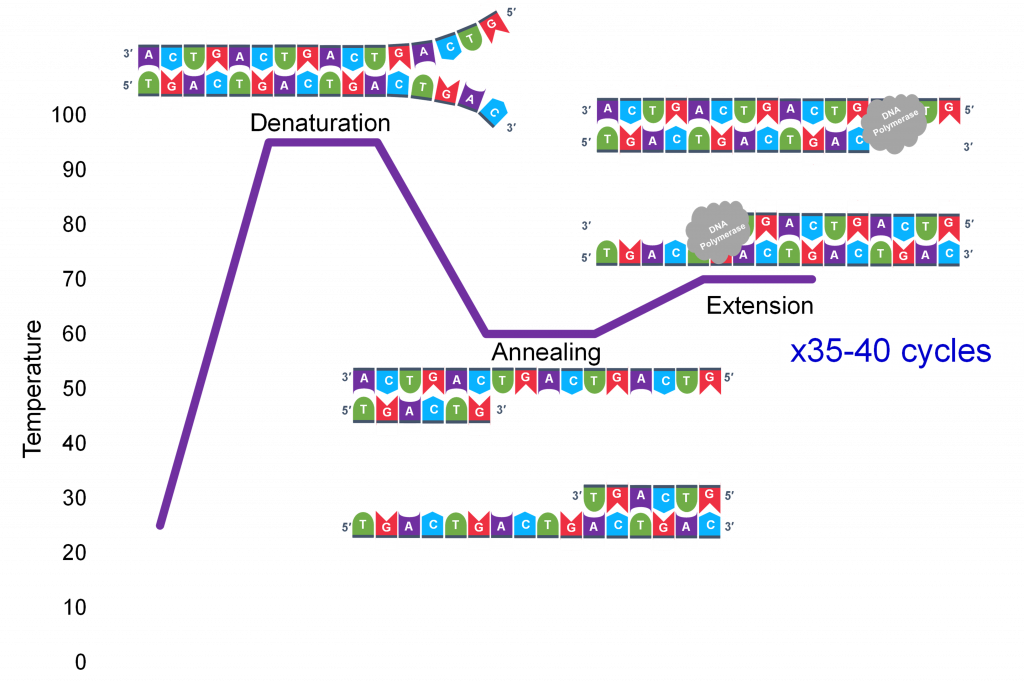
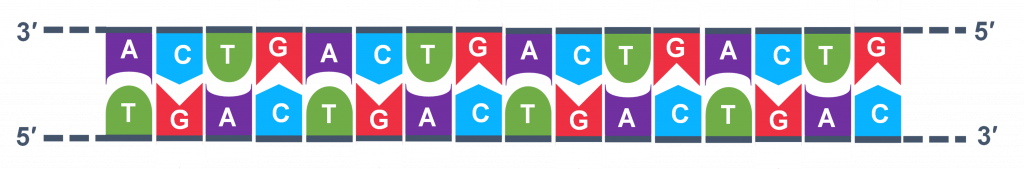
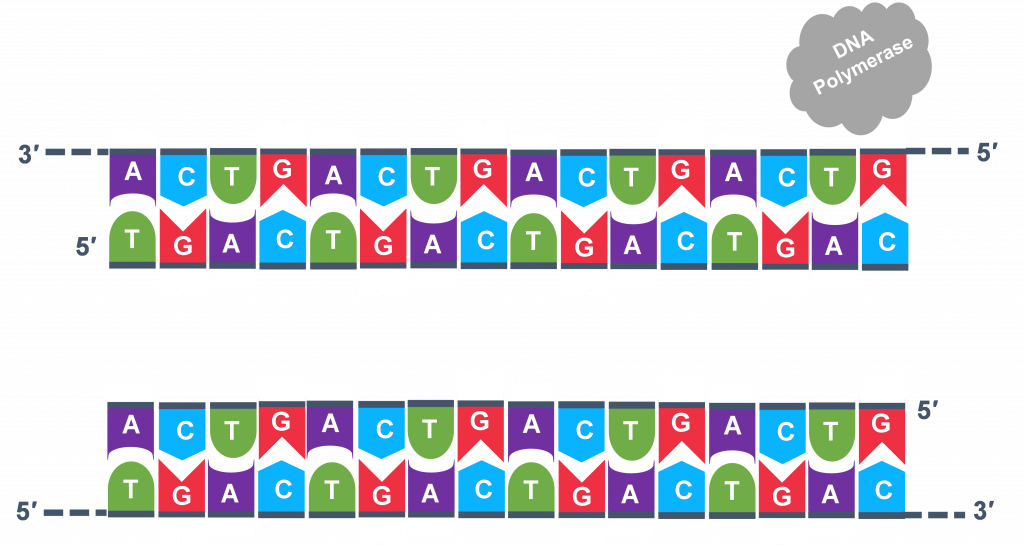
En general, una sola ejecución de PCR sufrirá 25-35 ciclos. El primer paso para un solo ciclo es el paso de desnaturalización, en el que la molécula molde de ADN bicatenario (Figura 2) se hace monocatenaria (Figura 3) . La temperatura para esta etapa está típicamente en el rango de 95-100°C, cerca de ebullición. El alto calor rompe los enlaces de hidrógeno entre las cadenas pero no rompe los enlaces azúcar-fosfato que mantienen juntos los nucleótidos de una sola cadena (Figura 3 ).
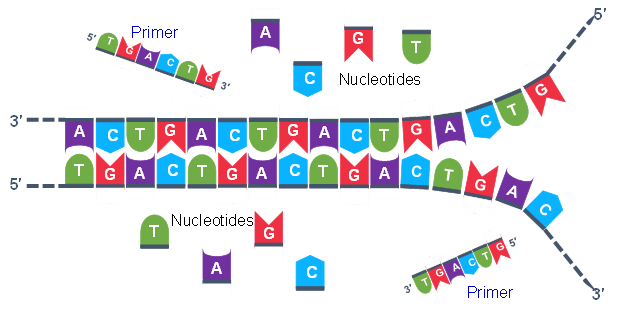
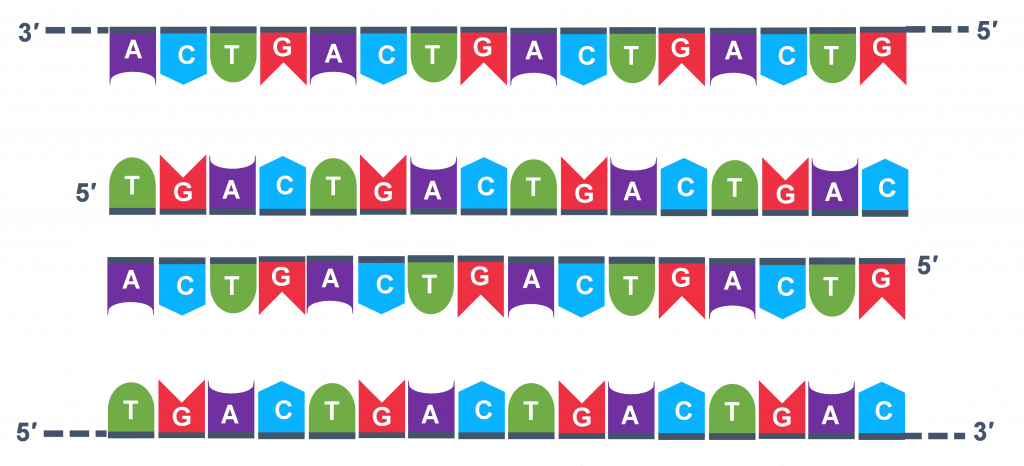
Miles de copias de los cebadores monocatenarios y los nucleótidos individuales se agregaron al tubo de ensayo antes de comenzar los ciclos. Tanto los cebadores como los nucleótidos pasarán a formar parte de las nuevas cadenas de ADN. La segunda etapa en la reacción de PCR es enfriar la temperatura en el tubo de ensayo a 45-55°C, esta es la etapa de reasociación de cebadores en la que los cebadores se unen a complementarios en el molde de ADN monocatenario. Los dos cebadores se denominan cebador directo e inverso y se diseñan porque sus secuencias se dirigirán al segmento deseado del molde de ADN para replicación (Figura 4).
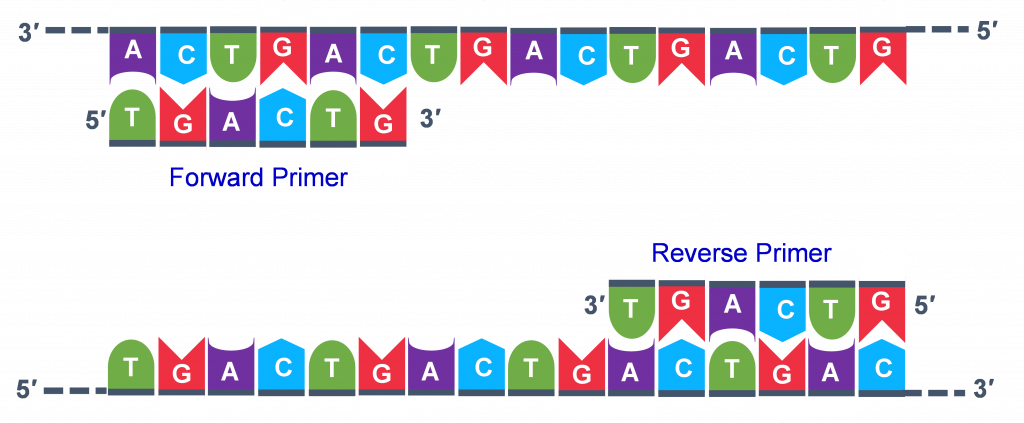
El genetista que planea el análisis de PCR debe “diseñar” los cebadores directo e inverso y luego comprarlos a un proveedor que pueda sintetizar ADN monocatenario que tenga una secuencia y longitud específicas. Los dos criterios más importantes para el diseño de cebadores son los siguientes.
- Un cebador debe tener una secuencia que complemente una de las cadenas molde y el otro cebador debe ser complementario a la otra hebra. AMBAS hebras necesitan ser cebadas para el proceso de replicación.
- Los cebadores deben unirse de manera que sus extremos 3' estén 'apuntando' en la dirección del otro cebador. Esto asegura que la secuencia entre los cebadores se replica en los ciclos de PCR.
Extensión: La etapa final de la PCR es cuando la enzima ADN polimerasa lee el molde y se conecta a nuevos nucleótidos al extremo 3' del cebador, extendiendo una nueva cadena complementaria de ADN (Figura 5). Al finalizar la etapa final y el primer ciclo de PCR, se realizarán dos copias de ADN bicatenario a partir del ADN molde original, duplicando la cantidad de ADN presente. El tubo de ensayo se calienta a alrededor de 75°C , optimizando el ADN pol. III y la cadena de ADN recién sintetizada se extiende a medida que la cadena molde es leída por ADN pol. III . El paso de Extensión se ejecutará durante unos minutos y este paso completará un ciclo de PCR.
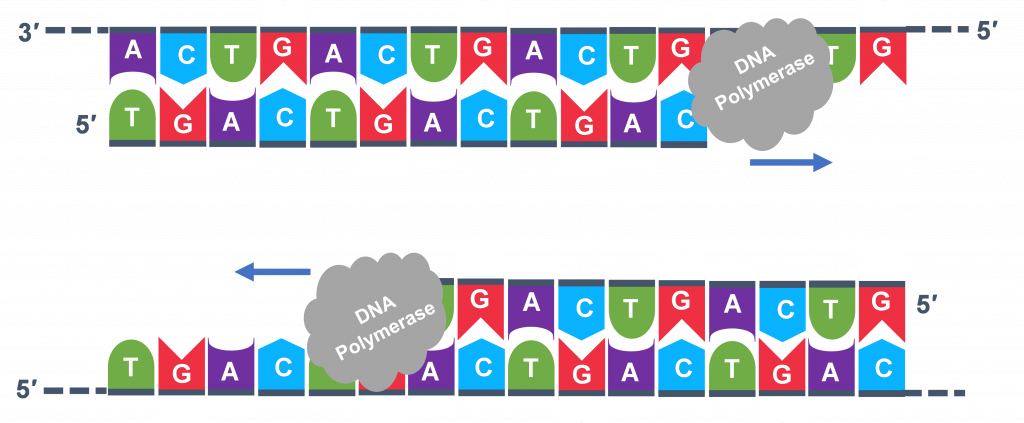
Para el ciclo 2, se repiten las etapas de desnaturalización, recocido y extensión (Figura 6 a, b, c). Esta vez, aunque habrá el doble de moléculas molde de ADN en comparación con lo que había al inicio del ciclo 1. Se están haciendo copias leyendo la plantilla original y las copias se hacen leyendo las copias realizadas en el ciclo anterior. Por lo tanto, si todo está funcionando correctamente, la replicación del ADN en el tubo de ensayo es una reacción en cadena donde al final de un ciclo, hay el doble de la cantidad de esa secuencia de ADN que la que se encontró al inicio del ciclo.
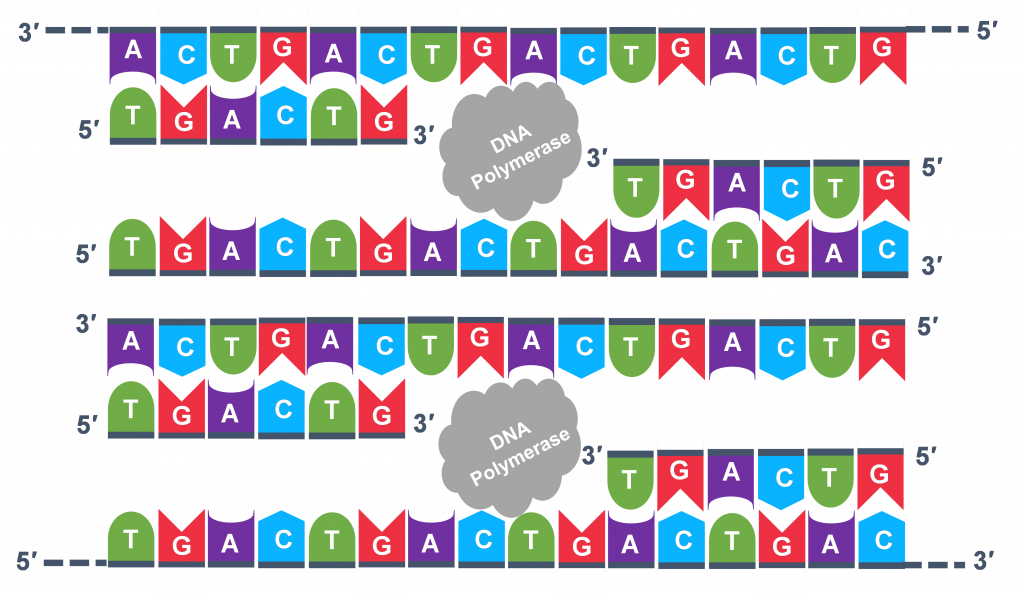
Debido a que miles de copias del cebador directo e inverso se agregan al inicio de la PCR, todos los moldes monocatenarios, tanto el original, las copias en el ciclo 3 y posteriores, como las copias de las copias hechas de ciclos anteriores se cebarán para el paso de extensión de los ciclos.
Termociclador

Cuando se inventó la PCR por primera vez, los científicos utilizaron baños de agua establecidos a diferentes temperaturas y la transferencia manual de tubos de ensayo a intervalos cronometrados para ejecutar la reacción PCR. Una vez que la técnica se convirtió en una tecnología probada para el análisis de ADN, los ingenieros comenzaron a trabajar para crear máquinas de PCR. El instrumento que calienta y enfría las muestras de ADN se denomina termociclador (Figura 7). Cada pequeño tubo o pocillo de muestra en una placa contiene todos los componentes químicos necesarios para una reacción de PCR. La adición de una muestra específica a la mezcla de reacción proporciona el ADN molde. Un termociclador puede programarse para temperaturas específicas y la cantidad de tiempo empleado en cada temperatura. El diseño diseñado de cicladores térmicos para maximizar la replicación precisa del ADN diana en un pequeño volumen de muestra con la cantidad mínima de tiempo puede ser crítico en muchas aplicaciones de PCR.
Taq ADN polimerasa
Cuando el Dr. Kerry Mullis realizó los primeros experimentos de PCR, necesitó agregar una nueva muestra de ADN pol III después de cada paso de desnaturalización. Esto se debió a que la alta temperatura necesaria para desnaturalizar el molde de ADN bicatenario también desnaturalizó la estructura de la proteína pol III del ADN. La enzima ADN pol III comúnmente disponible para los genetistas moleculares era de la bacteria E. coli y esta enzima no tuvo estabilidad a temperaturas cercanas a la ebullición. Afortunadamente, los biólogos habían estado investigando Thermus aquaticus, (Taq) una eubacteria termófila que se encuentra en aguas termales (Chien et al. 1976). La versión Taq de DNA pol III no se desnaturaliza fácilmente en las altas temperaturas requeridas en la PCR; además, tiene una buena eficiencia, capaz de agregar 60 pares de bases/seg a 70°C.Al igual que todas las demás ADN polimerasas, Taq DNA pol III no puede comenzar la replicación del ADN sin la adición de un cebador inicial. Así, el descubrimiento de Taq DNA pol III y la disponibilidad comercial de esta enzima hicieron de la PCR una tecnología más confiable y factible que aceleró su aplicación a la investigación científica y pruebas diagnósticas.
Visualización de los Resultados con Electroforesis
Una vez que se ha completado una reacción de PCR, necesitamos poder ver los resultados. Para ello, se carga una muestra de la mezcla de PCR en un gel de agarosa para electroforesis. El gel de agarosa contiene una matriz de poros que le permite separar fragmentos de ADN en función de sus tamaños. Para obtener detalles sobre cómo configurar y ejecutar un gel de electroforesis, consulte Electroforesis: Cómo los científicos observan fragmentos de ADN

La Figura 8 muestra una imagen de un gel de electroforesis en gel que está funcionando. La caja de la derecha contiene ADN cargado en el gel de agarosa. El gel se colocó en una solución acuosa de electrolitos. Dependiendo del tipo de tinte utilizado, las bandas de color son un tinte que se agregó a la muestra de PCR antes de que se cargara en el pozo de la muestra. Esto permite el seguimiento de la progresión del ADN a través del gel. Conectada a esta unidad de gel hay una fuente de energía eléctrica que proporciona la fuerza para mover el ADN a través del gel. Dado que las moléculas de ADN están cargadas negativamente, migrarán hacia el electrodo rojo positivo. Los fragmentos de ADN más cortos se mueven más rápido que los fragmentos más largos a través de los poros del gel
Después de ejecutar el gel, el ADN se tiñe con un químico que se une específicamente a las moléculas de ADN y luego reflejará un color específico de luz visible o fluorescerá un color específico cuando se ve con luz ultravioleta. Una única 'banda' contiene 1000 fragmentos de ADN individuales, todos de la misma longitud. La Figura 10 ilustra la información visual que se puede obtener de un gel de electroforesis después de que se haya realizado. La corriente eléctrica mueve uniformemente todos los fragmentos de ADN a través de un gel en la misma dirección. Los pocillos de muestra en la parte superior de la imagen del gel establecen así carriles para que las muestras de ADN se muevan.
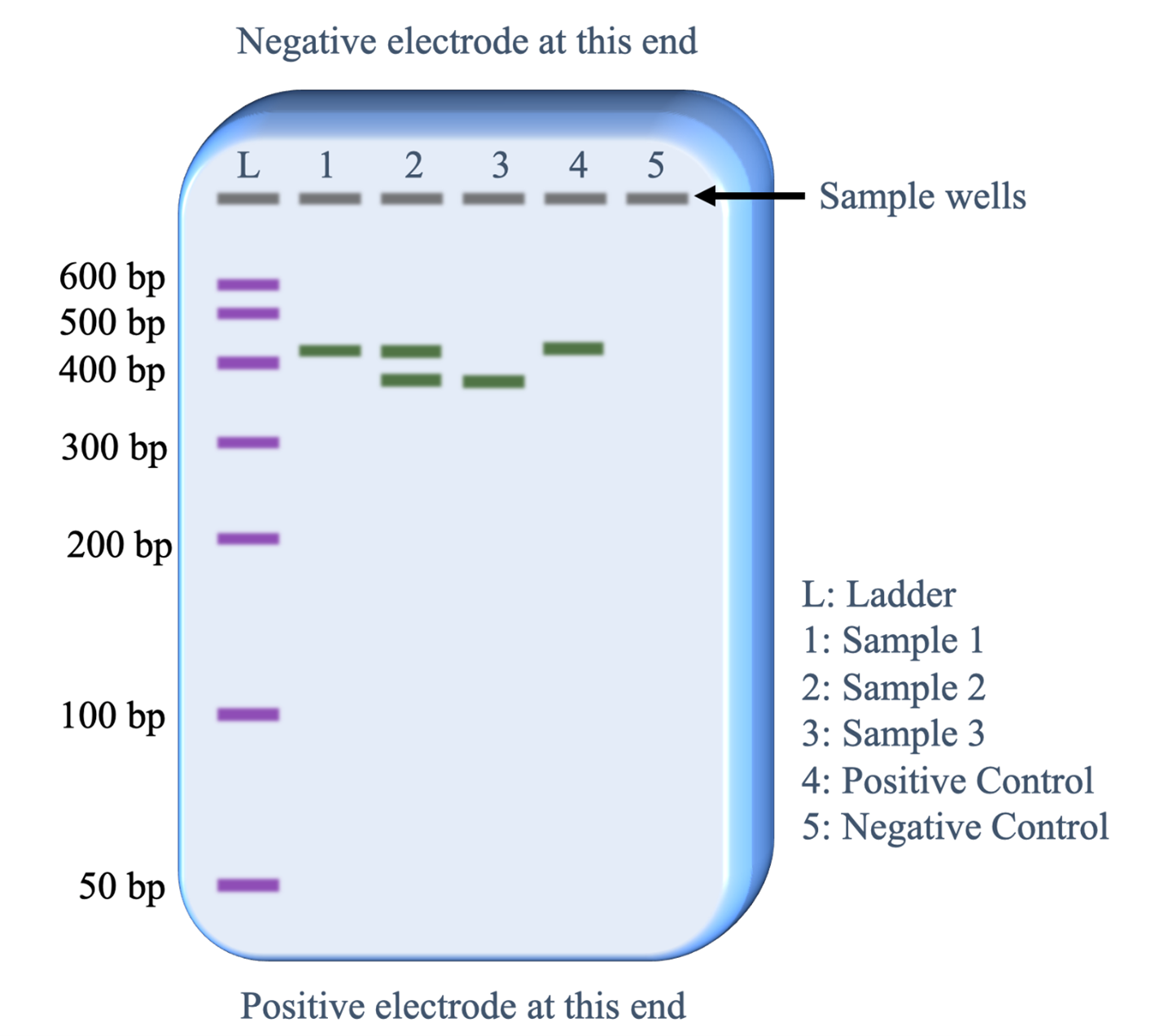
A continuación se muestra una descripción de qué información se revela de cada carril.
- Carril L: Esta se cargó con la escalera de tamaño de ADN que contiene copias de siete longitudes diferentes de fragmentos de ADN. Los vendedores comerciales de la escala de tamaños de ADN proporcionan información sobre las longitudes de cada fragmento en pares de bases (pb). Correr este carril proporciona una estimación de las longitudes de los fragmentos de ADN en los carriles de muestra (1-5).
- Carril 1: La muestra de PCR cargada en este carril tiene copias de una sola longitud de ADN. La longitud es ligeramente superior a 400 pares de bases.
- Carril 2: La muestra de PCR cargada en este carril tiene copias de dos longitudes de ADN. Un fragmento tiene la misma longitud que los fragmentos en el carril 1 y el segundo fragmento es ligeramente inferior a 400 pb.
- Carril 3: La muestra de PCR cargada en este carril tiene copias de un fragmento que tiene la misma longitud que los fragmentos más cortos en el carril 2.
- Carril 4: El control positivo funcionó como se predijo. La muestra se configuró con todos los mismos reactivos que las otras muestras de PCR más un molde de ADN que se sabía que contenía la secuencia dirigida por los cebadores de PCR que generarían un fragmento de 410 pares de bases.
- Carril 5: El control negativo funcionó como se predijo. La muestra se configuró con todos los mismos reactivos que las otras muestras de PCR excepto que no se agregó ADN molde. Por lo tanto, no esperaríamos que la reacción PCR funcionara y la ausencia de una banda de fragmentos de ADN es la esperada.
Debido a que los controles positivos y negativos funcionaron como se esperaba, el biólogo puede estar seguro de que las bandas de ADN observadas en los carriles 1,2 y 3 revelan información genética sobre el individuo que proporciona esa muestra de ADN.
Ventajas de la PCR
La PCR se convirtió rápidamente en el método de elección para muchos tipos de análisis de ADN debido a varias ventajas sobre otros métodos de detección de ADN. El primero; es un procedimiento sencillo de configurar y ejecutar. Menos pasos ahorran tiempo en obtener un resultado de análisis de ADN. El segundo es la sensibilidad. Se puede detectar una cantidad muy pequeña de ADN molde en la muestra. Incluso solo unas pocas células de la piel de un cabello humano contienen suficiente ADN, lo que hace que la PCR sea útil en forense. La tercera es que se puede diseñar para diferenciar con precisión muestras genéticas que son diferentes en tan poco como un solo nucleótido en la secuencia diana. Por último, en aplicaciones donde un gran número de muestras necesitan ser sometidas al mismo análisis de PCR, la automatización y la asistencia robótica permiten el procesamiento de muchas muestras en muy poco tiempo. Por ejemplo, una PCR automatizada sería fundamental para probar a un gran número de personas en horas durante una pandemia.
Limitaciones de la PCR
Hay algunos inconvenientes de usar PCR que uno debe tener en cuenta también. Primero, se requiere la secuencia del gen o región cromosómica a la que se dirige. Esta limitación está disminuyendo rápidamente a medida que la tecnología de secuenciación génica y genómica y el intercambio de esta secuencia a través de bases de datos de Internet ha surgido como la norma en el análisis genético. Debido al tamaño de los genomas de los seres vivos y una alta conservación de las secuencias génicas en muchos organismos, los cebadores diseñados para una prueba de PCR deben probarse empíricamente con los controles adecuados. Los biólogos pueden generar fácilmente ADN falso positivo a partir de PCR que es causado por contaminación o falta de especificidad en el diseño de cebadores. También puede ser necesario optimizar las concentraciones de cada componente químico. Por ejemplo, cambiar la cantidad de molde de ADN, MgCl2 y Taq polimerasa puede afectar tanto a la cantidad como a la calidad de las bandas producidas. Algunos estudios han demostrado que incluso la marca de la polimerasa Taq puede afectar los resultados (Holden et al. 2003). Asimismo, es posible que sea necesario ajustar los ciclos de temperatura para una prueba de PCR específica. Finalmente, como método de replicación de ADN in vitro, la PCR no puede replicar cromosomas enteros.
Para resaltar brevemente los temas de esta lección, recuerde que la PCR es una técnica de laboratorio relativamente fácil que amplifica la cantidad de ADN presente, al igual que lo hacen las células vivas en las etapas iniciales de un ciclo celular. Hay 5 componentes químicos de una reacción de PCR: un molde de ADN, una ADN polimerasa, cebadores, nucleótidos y un tampón. Los 3 pasos de temperatura para un ciclo son los pasos de desnaturalización, apareamiento de cebadores y extensión. Ahora hay muchas variaciones y usos de la PCR que van desde la ciencia forense hasta los estudios genómicos y la identificación de cultivos transgénicos.
Mira estos videos para conocer más sobre PCR:


