2.8: Introducción
- Page ID
- 58709
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
El ADN y el ARN son ambos ácidos nucleicos, que son los ácidos poliméricos aislados del núcleo de las células. El ADN y el ARN se pueden representar como simples cadenas de letras, donde cada letra corresponde a un nucleótido particular, el componente monomérico de los polímeros de ácido nucleico. Aunque esto transmite casi todo el contenido de información de los ácidos nucleicos, no te dice nada sobre las estructuras químicas subyacentes. En este capítulo se revisará la evidencia de que los ácidos nucleicos son el material genético, para luego explorar la estructura química de los ácidos nucleicos.
Hibridaciones Southern blot-
Después de la separación por electroforesis, los fragmentos de ADN se transfieren a una membrana (nylon o nitrocelulosa) y se inmovilizan; esta réplica del patrón de ADN en el gel se denomina “transferencia”. Una sonda marcada específica se hibrida con la transferencia para detectar secuencias relacionadas. Después de lavar la sonda unida no específicamente, los híbridos específicos se detectan por autorradiografía de la transferencia.
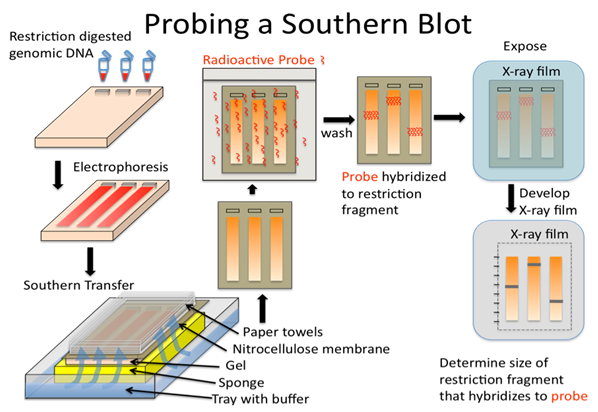
Los sitios de restricción pueden ser utilizados como marcadores genéticos. Se pueden identificar polimorfismos de longitud de fragmentos de restricción (RFLP) que están unidos a un locus particular. Esto se puede utilizar para
- Desarrollar una prueba de diagnóstico para un locus de la enfermedad (por ejemplo, enfermedad de células falciformes)
- Ayudar a aislar el gen.
- Huellas de ADN para loci altamente variables.
Tamaños de ADN y cromosomas, y métodos para resolverlos
La siguiente figura presenta vistas de cromosomas y segmentos de ADN en cuatro escalas diferentes en expansión. El nivel superior compara los tamaños de cromosomas intactos de cuatro de los organismos que vamos a discutir en este curso. Luego se expande la escala sobre el cromosoma III de levadura para que pueda compararse con algunos de los genomas virales y plasmídicos que son de uso común. A continuación, se da una vista de mayor resolución del plásmido pBR322, y finalmente la resolución más alta a la que normalmente nos ocupa, es decir, la secuencia de nucleótidos.

Determinar la secuencia de ADN y ARN
El enfoque básico es generar un conjunto anidado de fragmentos de ADN que inician un sitio común y terminan en A, G, C o T. Estos conjuntos de fragmentos de ADN (marcados) se separan en un gel de poliacrilamida desnaturalizante que tiene una resolución de 1 pb. El patrón resultante permite leer la secuencia. La modificación y degradación química específica de bases, desarrollada por Maxam y Gilbert, fue un enfoque ampliamente utilizado. La escisión específica de nucleótidos del ARN por un conjunto de ARNasas se puede usar para secuenciar ARN. Nos centraremos en el método más común de secuenciación del ADN, el de terminación de cadena específica de nucleótidos.
El método de terminación de cadena de didesoxinucleótidos se desarrolló en el laboratorio de Fred Sanger en Cambridge. Se puede incorporar un didesoxinucleótido2', 3' al ADN, según lo dirigido por la cadena molde. Sin embargo, el 3'-OH que falta impide la polimerización adicional. Por lo tanto, la cadena recién sintetizada de nucleótidos termina en didesoxinucleótidos de terminación de cadena específica de bases. Las reacciones se ejecutan de tal manera que todos los productos terminan en una G, una C, una A o AT, pero todos comienzan en el mismo lugar. Esto genera un conjunto anidado de productos cuya longitud es una medida de la posición de todas las G en una secuencia diana, o todas las C, etc. Así se puede deducir que la secuencia diana es complementaria a, por ejemplo, G en la posición 1, T en la posición 2, C en las posiciones 3 y 4, etc. para cientos de nucleótidos por serie.
Con más detalle, un cebador específico se hibrida con el molde, aguas arriba de la región a secuenciar. La ADN polimerasa catalizará la síntesis de nuevo ADN a partir del extremo 3' de ese cebador (elongación). Por lo tanto, el cebador genera un extremo común a todos los fragmentos del producto. (Esta es la base para el conjunto anidado en este enfoque).
El ADN sintetizado está marcado con un nucleótido radiactivo, como [A35s] desoxi-tio‑ATP, o un colorante fluorescente, a menudo unido al cebador.
Se incluye un terminador de cadena específico de base en cada una de las cuatro reacciones:
- 2',3' didesoxiGTP en la reacción “G”.
- 2',3' didesoxiATP en la reacción “A”.
- 2',3' didesoxiTTP en la reacción “T”.
- 2',3' didesoxiCTP en la reacción “C”.
La ADN polimerasa se alargará de cada cebador hibridado hasta incorporar un didesoxinucleótidos 2', 3'. No se pueden agregar nucleótidos adicionales a este producto, ya que no tiene OH 3', por lo que es un terminador de cadena. Esta terminación ocurre solo en los residuos G (complementarios a los C en el molde) en la reacción “G”, solo en los residuos A en la reacción “A”, etc. Así los productos de cada reacción comprenden un conjunto anidado de fragmentos, con el cebador específico en el extremo 5' y el terminador de cadena específico de base en el extremo 3'. Los productos se resuelven en un gel de secuenciación, se exponen a película de rayos X y se lee la secuencia, como en la Figura 2.30.

El enfoque de terminación de cadena de didesoxinucleótidos es el método utilizado en secuenciadores automatizados. Se incluyen diferentes colorantes fluorescentes de color (generalmente unidos al cebador) en cada reacción específica de base. Por lo tanto, los productos de los cuatro se pueden ejecutar en 1 carril del gel de resolución, permitiendo que se analicen >20 conjuntos de secuenciación a la vez. Un láser escanea continuamente a lo largo de una zona del gel, y registra cuando se detecta una fluorescencia (por ejemplo) roja, verde, azul o amarilla en cada carril, lo que significa que el cebador se extiende a un (por ejemplo) A, G, C o T está pasando por la zona de detección. Estos datos se procesan automáticamente, y se genera una lectura con los picos para cada colorante fluorescente en función del tiempo de ejecución del gel y la secuencia deducida. Un ejemplo de la salida se muestra a continuación en blanco y negro; la salida original es en color (un color diferente para cada nucleótido). La edición manual de la secuencia deducida se puede hacer con base en los datos brutos, pero en proyectos de secuenciación a gran escala, cada región se determina aproximadamente 8 tiempos diferentes y se utiliza otro software para determinar el nucleótido que ocurre con mayor frecuencia en cada posición.
La capacidad de las máquinas de secuenciación automatizadas es extraordinaria. Se utilizan nuevas máquinas que utilizan electroforesis capilar en gel para generar millones de nucleótidos por día en los principales centros de secuenciación. Esta tecnología permite secuenciar rápidamente genomas grandes y complejos, como se discute en el Capítulo 4.

Superenrollamiento de ADN topológicamente restringido
El ADN topológicamente cerrado puede ser circular (círculos covalentemente cerrados) o bucles que están restringidos en la base. El enrollamiento (o envoltura) del ADN dúplex alrededor de su propio eje se llama superbobinado (Figura 2.32 medio).
- Las superbobinas negativas tuercen el ADN alrededor de su eje en la dirección opuesta a las vueltas en sentido horario de la doble hélice derecha (R-H).
- El ADN superenrollado negativamente está subenrollado (y por lo tanto favorece el desenrollado del dúplex).
- El ADN superenrollado negativamente tiene giros de superbobina R-H (Figura 2.32).
- Las superbobinas positivas tuercen el ADN en la misma dirección que las vueltas de la doble hélice R-H.
- El ADN superenrollado positivamente está sobreenrollado (la hélice se enrolla más fuertemente).
- El ADN superenrollado positivamente tiene giros de superbobina L-H.
Los giros en sentido horario de doble hélice R-H (forma A o B) generan una Torsión positiva (T); ver Figura 2.32 izquierda. Las vueltas hacia la derecha (ccw) de la hélice L-H (Z) generan una T negativa.
T = Número de torsión
- Para el ADN de la forma B, es + (# bp/10 pb por torsión)
- Para ADN de forma A, es + (# bp/11 pb por giro)
- Para el ADN Z, es - (# bp/12 pb por giro)
W = Número de retorcimiento es el giro del eje del dúplex de ADN en el espacio
- Molécula relajada W=0
- Superbobinas negativas, W es negativo
- Superbobinas positivas, W es positivo
L = Número de enlace = número total de veces que una hebra de la doble hélice (de una molécula cerrada) rodea (o enlaza) la otra.
\[L = W + T\]
- L no puede cambiar a menos que uno o ambos hilos estén rotos y reformados.
- Un cambio en el número de enlace, DL, se divide entre T y W (Figura 2.32 derecha). Por lo tanto:
\[DL=DW+DT\]
si\(DL = 0\),\(DW=-DT\)

El bromuro de etidio se intercala en el ADN y desenrosca (o desenrolla) el dúplex en -27° por molécula de bromuro de etidio intercalada. Así, la intercalación de 14 moléculas de bromuro de etidio desenroscará el dúplex 378o, es decir, ligeramente más de una torsión completa (que sería de 360°). Para este proceso de intercalación, DL=0, ya que no se rompen ni reforman enlaces covalentes en el ADN. El cambio en la torsión, DT, es negativo y, por lo tanto, DW es positivo. Así, la intercalación del bromuro de etidio puede relajar un círculo superenrollado negativamente, y la intercalación adicional hará que el ADN se superenrolle positivamente (Figura 2.33).

Es útil tener una expresión para superenrollar que sea independiente de la longitud. La densidad superhelicoidal es simplemente el número de vueltas superhelicoidales (S.H.) por giro (o torsión) de doble hélice.
\[\text{Superhelical density} = s= \dfrac{W}{T}\]
Esto es -0.05 para el ADN bacteriano natural. Es decir, en el ADN bacteriano, hay 1 giro S.H. negativo por 200 pb (calculado a partir de 1 giro S.H. negativo por 20 giros = 1 giro S.H. negativo por 200 pb)
El ADN superenrollado negativo tiene energía almacenada que favorece el desenrollado, o una transición de ADN de la forma B al ADN Z.
Para s = -0.05\(\Delta G=-9 Kcal/mole\), lo que favorece el desenrollado
Así, el superenrollamiento negativo podría favorecer el inicio de la transcripción y la iniciación de la replicación.
Topoisomerasas
Las topoisomerasas catalizan un cambio en el número de enlaces de ADN.
- Topo I = enzima de cierre de mellas, puede relajar el ADN superenrollado positivo o negativo, hace una ruptura transitoria en 1 hebra. E. coli Topo I relaja específicamente el ADN superenrollado negativamente. Timo de ternero Topo I trabaja en ADN superenrollado tanto negativa como positivamente.
- Topo II = girasa: utiliza la energía de la hidrólisis de ATP para introducir superbobinas negativas. Su mecanismo de acción es hacer una rotura transitoria de doble cadena, pasar un ADN dúplex a través de la rotura y luego volver a sellar la ruptura.
Medir un cambio en el número de enlace
Se puede medir un cambio en el número de enlaces (DL) por sedimentación, electroforesis o microscopía electrónica, como se ilustra en la Figura 2.34.


Colaboradores y Atribuciones
- Ross C. Hardison, T. Ming Chu Profesor de Bioquímica y Biología Molecular (La Universidad Estatal de Pensilvania)

