10: Transcripción: ARN polimerasas
- Page ID
- 58480
Recordemos el Dogma Central de la biología molecular: el ADN se transcribe en ARN, el cual se traduce en proteína. Cubriremos el material en ese orden, ya que esa es la dirección que fluye la información.
Introducción
El pirofosfato liberado se escinde en la célula a 2 Pi, una reacción energéticamente favorable que impulsa la reacción en la dirección de la síntesis. En presencia de exceso de PPi, puede ocurrir la reacción inversa de pirofosforólisis. La síntesis siempre procede en una dirección 5' a 3' (con respecto a la cadena de ARN en crecimiento). La plantilla se lee en una dirección de 3' a 5'.
B. Estructura de la ARN polimerasa de E. coli
1. Esta ARN polimerasa sintetiza todas las clases de ARN
ARNm, ARNr, ARNt
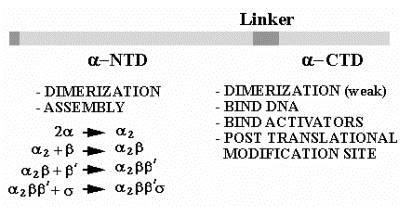
2. Se compone de cuatro subunidades.
a. Núcleo y holoenzima
A2bb's A2bb' + s
Holoenzima = a2bb's = core + s = puede iniciar la transcripción con precisión como el sitio apropiado, según lo determinado por el promotor
Núcleo = a2bb' = puede alargar una cadena de ARN en crecimiento
Un promotor puede definirse de dos maneras.
- La secuencia de ADN requerida para una iniciación precisa y específica de la transcripción
- La secuencia de ADN a la que se une la ARN polimerasa para iniciar con precisión la transcripción.
b. Subunidades
| Subunidad | Tamaño | Gene | Función |
|---|---|---|---|
| b' | 160 kDa | RPoC | b' + b forman el centro catalítico. |
| b | 155 kDa | RPoB | b' + b forman el centro catalítico. |
| a | 40 kDa | RPoA | ensamblaje enzimático; también se une a la secuencia UP en el promotor |
| s | 70 kDa (general) | RPOD | confiere especificidad para el promotor; se une a los sitios -10 y -35 en el promotor |
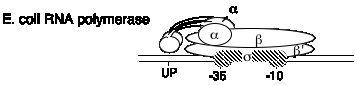
Mecanismo de la ARN polimerasa de C. E. coli
Modo de acción de los factores
La presencia del factor s provoca que la holoenzima ARN polimerasa sea selectiva en la elección del sitio de inicio. Esto se logra principalmente a través de efectos sobre la tasa de disociación de la ARN polimerasa del ADN.
- a. El núcleo tiene una fuerte afinidad por las secuencias generales de ADN. La t 1/2 para la disociación del complejo de núcleo-ADN es de aproximadamente 60 min. Esto es útil durante la fase de elongación, pero no durante la iniciación.
- b. La holoenzima tiene una afinidad reducida por el ADN general; disminuye aproximadamente 10 veces 4 veces. La t 1/2 para la disociación de la holoenzima del ADN general se reduce a aproximadamente 1 seg.
- c. La holoenzima tiene una afinidad mucho mayor por las secuencias promotoras.La t 1/2 para la disociación de la holoenzima de las secuencias promotoras es del orden de horas.
Eventos al inicio de la transcripción
- a. La holoenzima ARN polimerasa se une al promotor para formar un complejo cerrado; en esta etapa no hay desenrollado del ADN.
- b. El complejo polimerasa-promotor se somete a la transición cerrada a abierta, que es una fusión o desenrollado de aproximadamente 12 pb.
- c. Los nucleótidos iniciadores pueden unirse a la enzima, como lo dirigen sus nucleótidos complementarios en la cadena molde de ADN, y la enzima catalizará la formación de un enlace fosfodiéster entre ellos. Este complejo de polimerasa-DNA-ARN se conoce como el complejo ternario.
- d. Durante el inicio abortivo, la polimerasa cataliza la síntesis de transcritos cortos de aproximadamente 6 nucleótidos de longitud y luego los libera.
- e. Esta fase termina cuando el ARN naciente de ~6 nucleótidos se une a un segundo sitio de unión de ARN en la enzima; este segundo sitio es distinto del centro catalítico. Esta unión se asocia con “restablecer” el centro catalítico para que la enzima catalice ahora la síntesis de oligonucleótidos de 7-12 de longitud.
- f. La enzima ahora se transloca a una nueva posición en la plantilla. Durante este proceso sigma sale del complejo. Un cambio conformacional en la enzima asociada con sigma que sale del complejo permite que el “pulgar” se envuelva alrededor de la plantilla de ADN, bloqueando la procesividad. Así, la enzima central cataliza la síntesis de ARN durante la elongación, la cual continúa hasta que se encuentran “señales” que indican terminación.
Figura 3.1.8. Acontecimientos al inicio
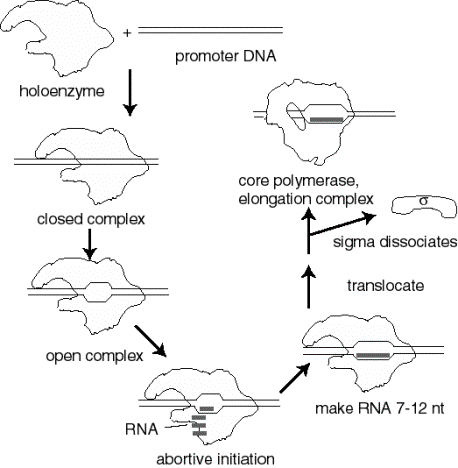
3. Ciclo de transcripción
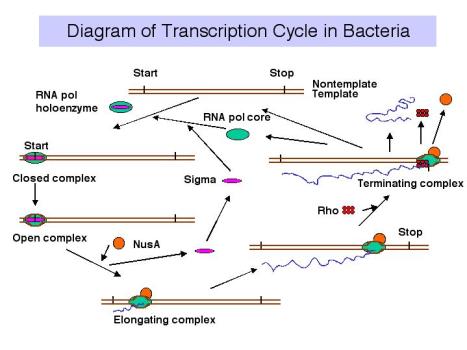
4. Sitios en el núcleo de la ARN polimerasa
a. La enzima cubre aproximadamente 60 pb de ADN, con una burbuja de transcripción de aproximadamente 17 pb desenrollada.
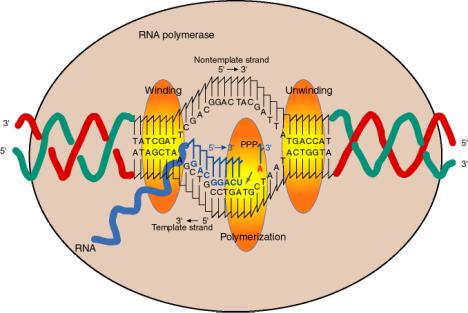
d El nucleótido entrante (NTP) que se agregará a la cadena de ARN en crecimiento se une adyacente al extremo 3' de la cadena de ARN en crecimiento, como lo indica el molde, en el sitio activo para la polimerización.
e. El nucleótido entrante se une a la cadena de ARN en crecimiento por ataque nucleofílico del OH 3' sobre el fosforilo a de la NTP, con liberación de pirofosfato.
f. la reacción progresa (la enzima se mueve) aproximadamente 50 nts por seg. Esto es mucho más lento que la velocidad de replicación (alrededor de 1000 nts por segundo).
g. Si el molde está limitado topológicamente, el ADN por delante de la ARN polimerasa se sobreenrolla (giros superhelicoidales positivos) y el ADN detrás de la ARN polimerasa se enrolla (giros superhelicoidales negativos).
El efecto del desenrollado del molde de ADN por la ARN polimerasa es disminuir T en 1 por cada 10 pb desenrollado. Así DT = -1, y desde DL = 0, luego DW = +1 por cada 10 pb desenrollados. Este efecto del incremento de W se ejercerá en el ADN por delante de la polimerasa.
El efecto de rebobinar la plantilla de ADN por la ARN polimerasa es justo lo contrario, por supuesto. T aumentará en 1 por cada 10 pb rebobinados. Así DT= +1, y desde DL = 0, luego DW = -1 por cada 10 pb rebobinados. Este efecto de la disminución de W se ejercerá en el ADN detrás de la polimerasa, ya que ahí es donde se está produciendo el rebobinado.
5. Inhibidores: reactivos útiles y pistas para funcionar
a. Rifamicinas, por ejemplo rifampicina: se unen a la subunidad b para bloquear la iniciación. El fármaco evita la adición del 3er o 4º nucleótido, de ahí que el proceso de iniciación no pueda completarse.
¿Cómo sabemos que el sitio de acción de la rifampicina es la subunidad b? Mutaciones que confieren resistencia al mapa de rifampicina al gen rpoB.
b. Streptolydigins: se unen a la subunidad b para inhibir la elongación de la cadena.
Estos efectos de las rifamicinas y estreptolidiginas, y el hecho de que actúan sobre la subunidad b, argumentan que la subunidad b es necesaria para la adición de nucleótidos a la cadena en crecimiento.
c. La heparina, un polianión, se une a la subunidad b' para evitar la unión al ADN in vitro
D. ARN polimerasas eucariotas
1. Los eucariotas tienen 3 ARN polimerasas diferentes en sus núcleos.
- a. Cada ARN polimerasa nuclear es una proteína grande con aproximadamente 8 a 14 subunidades. MW es aproximadamente 500,000 para cada uno.
- b. Cada polimerasa tiene una función diferente:
| ARN polimerasa | localización | sintetiza | efecto de una -amanitina |
|---|---|---|---|
| ARN polimerasa I | nucleolo | Pre-rRNA | ninguno |
| ARN polimerasa II | nucleoplasma | Pre-ARNm algunos ARNsnR | inhibida por bajas concentraciones (0.03 mg/ml) |
| ARN polimerasa III | nucleoplasma | Pre-ARNt, otros ARN pequeños algunos ARNsnR | inhibida por altas concentraciones (100 mg/ml) |
2. Estructuras de subunidades
a. Se han aislado los genes y proteínas codificadas para las subunidades de las ARN polimerasas de levadura y se han determinado las secuencias, y se han realizado algunos análisis funcionales.
b. Algunas de las subunidades son homólogas a las ARN polimerasas bacterianas: Las dos subunidades más grandes son homólogos de b y b'. La subunidad de aproximadamente 40 kDa es el homólogo de a.
c. Algunas subunidades son comunes a las tres ARN polimerasas.
d. Ejemplo de ARN polimerasa II de levadura:
| Tamaño aproximado (kDa) | subunidades por polimerasa | rol/comentario | |
|---|---|---|---|
| 220 | 1 | relacionados con b' | catalizador? |
| 130 | 1 | relacionados con b | catalizador? |
| 40 | 2 | relacionados con un | montaje? |
| 35 | < 1 | ||
| 30 | 2 | común a todos 3 | |
| 27 | 1 | común a todos 3 | |
| 24 | < 1 | ||
| 20 | 1 | común a todos 3 | |
| 14 | 2 | ||
| 10 | 1 |
e. La subunidad más grande tiene un dominio carboxi-terminal (CTD) con una estructura inusual: repeticiones en tándem de la secuencia Tyr‑Ser‑Pro‑Thr‑Ser‑Pro‑Thr. La enzima de levadura tiene 26 repeticiones en tándem y la enzima de mamífero tiene aproximadamente 50. Estos pueden fosforilarse en Ser y Thr para dar un CTD altamente cargado.
- El ARN Pol IIa no está fosforilado en el CTD.
- El ARN Pol IIo se fosforila en el CTD.

4. ARN polimerasas en cloroplastos (plastidios) y mitocondrias
- a. La ARN polimerasa que se encuentra en los plastidios está codificada en el cromosoma plastidio. En algunas especies la ARN polimerasa mitocondrial está codificada por el ADN mitocondrial.
- b. Estas ARN polimerasas organelares están mucho más relacionadas con las ARN polimerasas bacterianas que con las ARN polimerasas nucleares. Este es un argumento fuerte a favor de que los orígenes de estos orgánulos sean bacterianos, apoyando el modelo endo-simbiotnt para la adquisición de estos orgánulos en eucariotas.
- c. Estas ARN polimerasas catalizan la transcripción específica de genes organelares.
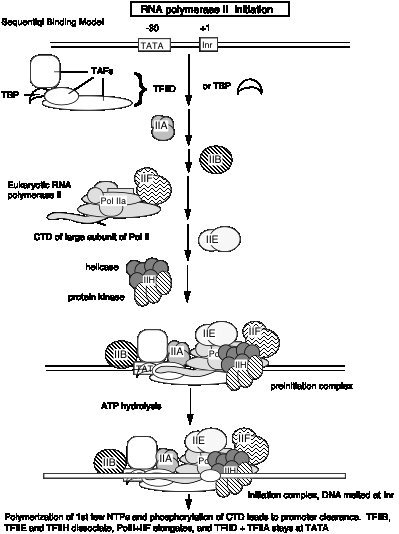
E. Factores generales de transcripción para la ARN polimerasa eucariota II
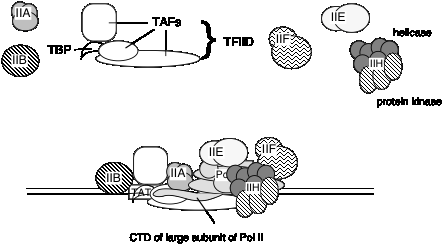
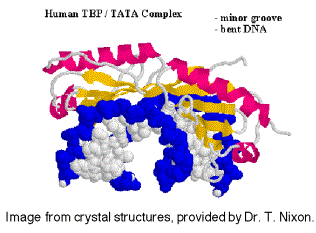
No se sabe si el mismo conjunto de TAF están en el TFIID para todos los promotores transcritos por la ARN polimerasa II, o si algunos se usan solo para ciertos tipos de promotores. El TFIID es el único factor de transcripción general específico de secuencia que se ha caracterizado hasta ahora, y se une en el surco menor del ADN. También se utiliza en promotores sin TATA, por lo que aún se está investigando el papel de la unión específica de la secuencia.
3. Resumen de los factores generales de transcripción para la ARN polimerasa II.
Factores para la ARN polimerasa II (células humanas)
| Factor | No. de subunidades | Molecular masa (kDa) | Funciones | Funciones para Reclutar: |
|---|---|---|---|---|
| TFIID: TBP | 1 | 38 | Reconocer el promotor del núcleo (TATA) | TFIIB |
| TFIID: TAF | 12 | 15-250 | Reconocer promotor central (no TATA); Regulación positiva y negativa | ARN Pol II? |
| TFIIA | 2 | 12, 19, 35 | Estabilizar la unión de TBP-ADN; Anti-represión | |
| TFIIB | 1 | 35 | Seleccionar sitio de inicio para RNA Pol II | ARN poliI-TFIIF |
| ARN Pol II | 12 | 10-220 | Catalizar la síntesis de ARN | TFIIE |
| TFIIF | 2 | 30, 74 | ARN diana poliI al promotor; desestabiliza interacciones no específicas entre poliI y ADN | |
| TFIIE | 2 | 34, 57 | Modula las actividades de helicasa, ATPasa y quinasa de TFIIH; ¿potenciar directamente la fusión del promotor? | TFIIH |
| TFIIH | 9 | 35-89 | ¿Helicasa para fundir promotor; CTD quinasa; aclaramiento del promotor? |
Roeder, R.G. (1996) TIBS 21:327-335.
4. El TFIIH es un factor de transcripción multisubunidad que también participa en la reparación del ADN.
Subunidades del factor humano
| Gene | Masa molecular de proteína (kDa) | Función/Estructura | Rol propuesto |
|---|---|---|---|
| XPB | 89 | helicasa, pistas 3' a 5' | Desenrollar dúplex para transcripción/reparación |
| XPD | 80 | helicasa, pistas 5' a 3' | Desenrollar dúplex, Reparar |
| P62 | 62 | desconocido | |
| P52 | 52 | desconocido | |
| P44 | 44 | Dedo Zn | Se une al ADN |
| P34 | 34 | Dedo Zn | |
| MAT1 | 32 | Factor de montaje CDK | |
| Ciclin H | 38 | Compañero de ciclina para CDK7/MO15 | |
| CDK7/MO15 | 32 | Proteína quinasa | Quinasa para CTD |

Cuadro 3.1.6. Holoenzima ARN polimerasa II y mediador

Estos estudios muestran que la ARN polimerasa II puede existir en varios estados o complejos diferentes. Uno se encuentra en un holocomplejo muy grande que contiene al mediador. En este estado, iniciará con precisión la transcripción cuando esté dirigida por TFIID, y responderá a activadores (Cuadro 3.1.6). El subcomplejo mediador parece ser capaz de disociarse y reasociarse con la ARN polimerasa II y los GTFs. En efecto, esta reasociación podría ser el paso que se ensayó en la identificación del mediador. Sin mediador, la ARN polimerasa II más los GTFs pueden iniciar la transcripción en el lugar correcto (según las indicaciones del TFIID), pero no responden a los activadores. En ausencia de GTFs, la ARN polimerasa II es capaz de transcribir moldes de ADN, pero no iniciará la transcripción en el sitio correcto. De ahí que sea competente para el alargamiento pero no para la iniciación.
Cuadro 3.1.7. Ampliar las funciones de la ARN polimerasa II
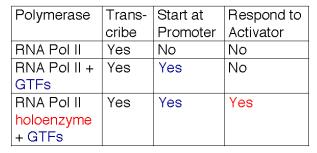
Fig.3.1.17
![]()
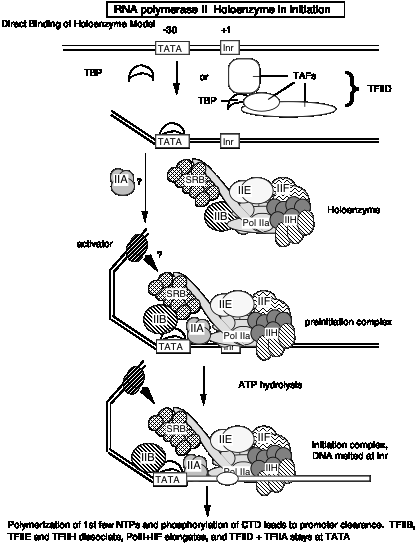
Si la holoenzima es la enzima primaria involucrada en el inicio de la transcripción en células eucariotas, entonces la vía de ensamblaje progresivo observada in vitro (ver sección d anterior) puede ser de poca relevancia in vivo. Quizás la holoenzima se unirá a promotores simplemente marcados por la unión de TBP (o TFIID) a la caja TATA, en contraste con el modelo de ensamblaje progresivo que tiene un mecanismo de ensamblaje ordenado más extenso. En ambos modelos, la unión a TBP o TFIID es el paso inicial en el ensamblaje del complejo de preiniciación. Sin embargo, en este punto no se puede descartar la posibilidad de que la holoenzima se use en algunos promotores, y el ensamblaje progresivo ocurre en otros.
7. Dianas para las proteínas activadoras
Las dianas para las proteínas activadoras transcripcionales pueden ser algún componente del complejo de iniciación. Una línea de investigación apunta a los TAF en TFIID así como TFIIB como dianas para los activadores. Así, los activadores podrán facilitar el montaje ordenado del complejo de iniciación mediante el reclutamiento de GTFs. Sin embargo, la holoenzima contiene el “mediador” o complejo SRB que puede mediar la respuesta a los activadores. Así, los activadores pueden servir para reclutar la holoenzima al promotor. Se requieren estudios adicionales para establecer si uno u otro es correcto, o si estos son caminos separados para la activación.
F. Factores generales de transcripción para ARN polimerasas eucariotas I y III
1. Factores generales de transcripción para la ARN polimerasa I
a. El promotor central cubre el sitio de inicio de la transcripción, más un elemento de control aguas arriba localizado aproximadamente 70 pb más 5'.
b. El factor UBF1 se une a una secuencia rica en G+C tanto en el elemento control aguas arriba como en el promotor central.
c. Un complejo multisubunidad llamado SL1 se une al complejo UBF1-ADN, nuevamente tanto en los elementos aguas arriba como en el núcleo.
d. Una de las subuntis de SL1 es TBP, ¡la proteína de unión a TATA de TFIID!
e. La ARN polimerasa I luego se une a este complejo de DNA+UBF1+SL1 para iniciar la transcripción en el nucleótido correcto y el alargado para hacer pre-ARNr.
2. Factores generales de transcripción para ARN Pol III
- a. Las secuencias de control interno son características de genes transcritos por ARN Pol III (ver más adelante).
- b. TFIIIA: se une a la región de control interno de genes que codifican ARN 5S (promotor interno tipo 1)
- c. TFIIIC: se une a regiones de control interno de genes para ARN 5S (junto con TFIIIA) y para ARNt (promotores internos tipo 2)
- d. TFIIIB: La unión de TFIIIC dirige a TFIIIB a unirse a secuencias (-40 a +11) que se superponen al sitio de inicio para la transcripción. Una subunidad de TFIIIB es TBP, aunque no se requiere caja TATA para la transcripción. TFIIIA y TFIIIC ahora pueden eliminarse sin afectar la capacidad de la ARN polimerasa III para iniciar la transcripción. Así, TFIIIA y TFIIIC son factores de ensamblaje, y TFIIIB es el factor de iniciación.
Figura 3.1.18.
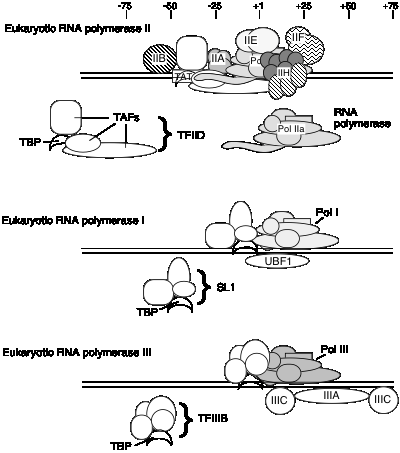
e. La ARN polimerasa III se une al complejo de TFIIIB+ADN para iniciar la transcripción de manera precisa y eficiente.
3 Factor de transcripción utilizado por las 3 ARN Pol'ases: TBP
La TBP parece desempeñar un papel común en la dirección de la ARN polimerasa (I, II y III) para que se inicie en el lugar correcto. Los factores multisubunitarios que contienen TBP (TFIID, SL1 y TFIIIB) pueden servir como factores de posicionamiento para sus respectivas polimerasas.


