14: Traducción (Síntesis de proteínas)
- Page ID
- 58522
Un recordatorio: el ARNm codifica el polipéptido con cada aminoácido designado por una cadena de tres nucleótidos. Los ARNt sirven como adaptadores para traducir del lenguaje de los ácidos nucleicos al de las proteínas. Los ribosomas son las fábricas para la síntesis de proteínas.
A. ARNt
1. Los ARN de transferencia, o ARNt, sirven como adaptadores para alinear los aminoácidos apropiados en los moldes de ARNm.
Figura 3.5.1.

2. Estructura primaria de los ARNt
a. Los ARNt son cortos, siendo de sólo 73 a 93 nts de largo.
b. Todos los ARNt tienen el trinucleótido CCA en el extremo 3'.
- El aminoácido está unido a la A terminal del CCA.
- En la mayoría de los genes de ARNt procariotas, el CCA está codificado en el extremo 3' del gen. Ningún gen de ARNt eucariota conocido codifica el CCA, sino que es agregado postranscripcionalmente por la enzima ARNt nucleotidiltransferasa.
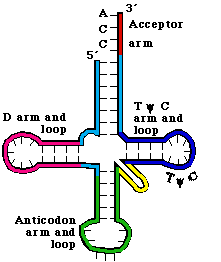
a. Los ARNt tienen 4 brazos con 3 bucles (ver Figura 3. 5.2. para ARNt de fenilalanina de levadura)
b. El brazo aceptor de aminoácidos está formado por apareamiento de bases complementario entre los 7 nts iniciales del ARNt y un segmento corto cerca del extremo 3'. Nuevamente, el aminoácido se añadirá a la A terminal.
c. El brazo D termina en el bucle D. Contiene varias dihidrouridinas, las cuales se abrevian como “D”.
d. El brazo anticodón termina en bucle anticodón. El anticodón se localiza en el centro del bucle. Se alineará de 3' a 5' con el ARNm (lectura de 5' a 3').
e. El bucle variable varía en tamaño en diferentes ARNt. La diferencia de tamaño entre los ARNt de 73 nt versus 93 nt se encuentra en el bucle variable.
f. El brazo TyC se llama así por este motivo altamente conservado que se encuentra en el bucle.
4. La estructura terciaria del ARNt es una “L grasa”. (Ver Fig 3.5.3.)
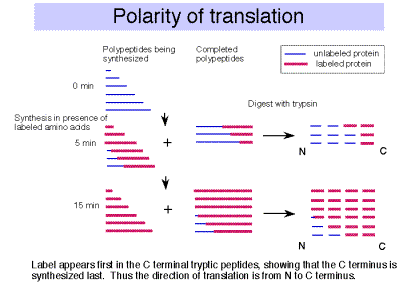
F. La polaridad de la traducción es desde el extremo amino (N) hasta el extremo caboxi (C).
Esto fue demostrado en un experimento clásico por Dintzis.
- Las proteínas que se traducen activamente se marcaron con aminoácidos radiactivos por un breve tiempo (corto en relación con el tiempo requerido para completar la síntesis).
- Se recolectaron polipéptidos completos, se digirieron con tripsina y se determinó la cantidad de radiactividad en los fragmentos trípticos.
- Los fragmentos trípticos del extremo C-terminal del polipéptido tuvieron radiactividad en los primeros tiempos de marcaje.
- A medida que se incrementó el periodo de marcaje (pulso más largo), se marcaron fragmentos trípticos más cercanos al extremo N terminal.
- Esto muestra que la dirección del crecimiento del polipéptido es desde el N teminus hasta el extremo C, es decir, la traducción comienza en el aminoácido N-terminal. Esto corresponde al crecimiento de la cadena de ARNm en una dirección 5' a 3'.
- Tenga en cuenta que este protocolo experimental también se utiliza para mapear los orígenes de replicación, tal y como lo cubrimos en la Segunda Parte del curso.
b. IF3 = Factor de iniciación 3
- Un factor antiasociación; previene la asociación entre las subunidades ribosómicas grandes y pequeñas.
- También debe estar asociada con la subunidad pequeña para que forme un complejo de iniciación, es decir, para que la subunidad pequeña se una correctamente al ARNm y a FMET-ARNf.
- Se disocia antes de la unión de la subunidad grande.
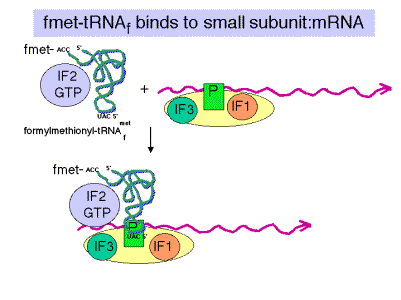
c. IF2
- Lleva fMET-ARNf al sitio P parcial en la subunidad pequeña.
- Al menos en eucariotas, lo hace en un complejo ternario con IF2, FMET‑TrNAF y GTP. En bacterias, el GTP puede unirse al complejo de iniciación por separado. [En algunos textos, como MBOG, p. 412, el complejo GTP-IF2 se une a la subunidad 30S por separado de fMET-ARNf. ¿Cómo probarías las diferencias en estos dos modelos?]
- IF2 activa una actividad GTPasa en la subunidad pequeña. El cambio resultante en la conformación puede permitir que la subunidad grande se una.
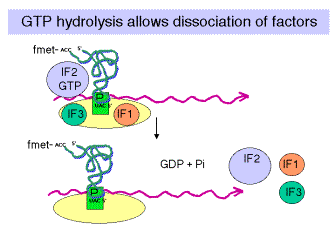
3. La unión de la subunidad 50S (grande) al complejo de iniciación da un ribosoma completo listo para la fase de elongación de la traducción. Tenga en cuenta que F-Met-Trnafmet se posiciona en el sitio P. Se ha reconocido el iniciador AUG en el ARNm.
4. Identificación del iniciador AUG en eucariotas
a. Bases alrededor de AUG influyen en la eficiencia de la iniciación.
- Los efectos más importantes son de una purina 3 nt antes de AUG y una G después de ella. El contexto preferido es R NN AUG G.
- La secuencia consenso para un gran número de ARNm es GCC R CC AUG, pero estos otros nucleótidos tienen poco efecto en los experimentos de mutagénesis.
a. Modelo de escáner modificado
(1) El ARNm se “prepara” para unirse al ribosoma por la acción del factor de iniciación eucariota 4, abreviado eIF4 (Figura 3.5.16). eIF4 es un factor de múltiples subunidades; incluye una proteína de unión a cap, eIF4F, que reconoce la estructura de casquete 5'. También incluye las proteínas eIF4A y eIF4B. Estas son las ARN helicasas, que desenrollan estructuras secundarias en la región 5' no traducida del ARNm a expensas de la hidrólisis de ATP.
Luego, el ARNm se une a la subunidad ribosómica pequeña. El Met-ARNt ya ha sido llevado a la subunidad ribosómica pequeña por eIF2, en un complejo con GTP.
eIF3 mantiene la subunidad ribosómica pequeña separada de la subunidad grande durante el proceso de unión al ARNm.
(2) La subunidad pequeña, con factores asociados, escanea a lo largo del ARNm hasta alcanzar (generalmente) el primer AUG. Los factores eIF1 y eIF1a ayudan a mover el complejo de preiniciación al inicio AUG.
Figura 3.5.16.
H. El ciclo de elongación durante la traslación
1. Unión de aminoacil-ARNt al sitio A
Revisión reciente: Weijland, A. y A. Parmeggiani (1994) TIBS 19:188-193. Schroeder, R. (1994) Naturaleza 370:597.
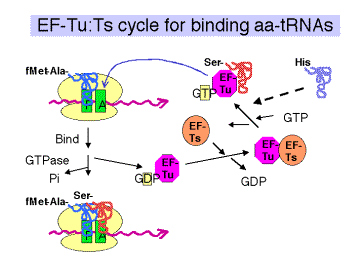
a. Factor de elongación EF‑Tu
- El complejo ternario de aminoacil-ARNt, EF‑Tu y GTP lleva el aminoacil-ARNt al sitio A en el ribosoma 70S (fig. 3.5.17).
- Después de que el aminoacil-ARNt se deposita en el sitio A del ribosoma, el GTP se escinde a GDP + Pi. El complejo binario de EF‑Tu y PIB se disocia del ribosoma.
- Este es uno de los muchos ejemplos de proteínas de unión a nucleótidos de guanina que son activas cuando GTP está unido e inactivas cuando el PIB está unido.
El modelo general es que el estado unido a GTP de EF-Tu adopta una conformación con una alta afinidad por aminoacil-ARNt. La conformación (forma, densidad de carga, etc.) del complejo ternario resultante (que contiene EF-Tu, GTP y aminoacil-ARNt) permite entonces que se una al sitio A del ribsosoma. La hidrólisis de GTP para formar GDP y fosfato inorgánico hace que el EF-Tu adopte una conformación diferente. El aminoacil-ARNt ahora tiene una menor afinidad por EF-Tu en el estado unido al GDP, y presumiblemente una mayor afinidad por el sitio A en el ribosoma, por lo que permanece en el ribosoma cuando EF-Tu en el estado unido al GDP se disocia (tanto del aminoacil-ARNt como del ribosoma).
Figura 3.5.17.
(4) EF‑Tu es una de las proteínas más abundantes en E. coli, con 70,000 copias por célula. Esto es casi igual al número de aminoacil-ARNt por célula, por lo que es probable que la mayoría de los aminoacil-ARNt estén en el complejo ternario cuando la concentración de GTP es suficientemente alta.
b. GTP
- Requerido para la unión de aminoacil-ARNt.
- La hidrólisis promueve la disociación del complejo EF‑Tu más PIB del ribosoma.
c. EF‑Ts
- Ayudas en el reciclaje de EF‑Tu por intercambio GDP‑GTP.
- Los EF‑T se unen a EF-tu complejados con el PIB, provocando disociación del PIB. El GTP ahora se puede unir al complejo EF‑Tu‑TS, lo que hace que los EF‑T se disocien y dejan el EF-Tu complejado con GTP. Este último complejo binario está listo para unirse a otro aminoacil-ARNt.
d. El antibiótico kirromicina impide la liberación de EF‑Tu‑PIB, bloqueando así la elongación. Esto demuestra que se debe completar un paso antes de que pueda darse el siguiente, e ilustra la importancia del ciclo EF‑TU‑GTP/PIB.
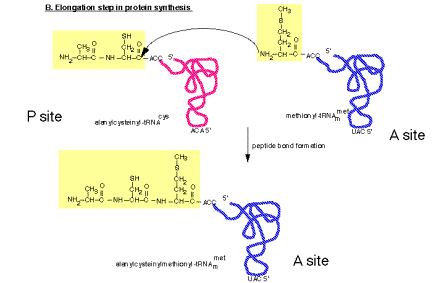
2. Péptidil transferasa en la subunidad ribosómica grande
a. La reacción de peptidil transferasa se produce por desplazamiento nucleofílico. El grupo amino del aminoacil-ARNt (posición n) ataca al grupo carboxilo “C-terminal” del peptidil-ARNt (posición n-1 en el ARNm). Esto da como resultado la escisión del enlace éster de peptidil-ARNt de alta energía, proporcionando así la energía libre para impulsar la reacción. Los productos resultantes de la reacción son ARNt desacilado en el sitio P y peptidil-ARNt en el sitio A.
Figura 3.5.18. Reacción de peptidil-transferasa
b. Papel del ARNr en la catálisis
Es probable que el ARNr proporcione el centro catalítico para la actividad peptidil-transferasa, con quizás algunas proteínas ribosómicas ayudando a mantener el ARNr en la conformación correcta para la catálisis. Esta conclusión se sustenta en varias líneas de investigación, algunas de las cuales se enumeran a continuación.
- Ninguna proteína, sola o en combinación con otras proteínas, ha demostrado catalizar la formación de enlaces peptídicos.
- Las regiones específicas del ARNr 16S (en la subunidad pequeña) interactúan con las regiones anticodón del ARNt tanto en los sitios A como P. Por el contrario, el ARNr 23S en la subunidad grande interactúa con el extremo CCA del peptidil-ARNt, colocándolo así en la ubicación correcta para la peptidil-transferasa.
- Los antibióticos eritromicina y cloranfenicol bloquean la peptidil transferasa. Algunas mutaciones que confieren resistencia a ellas se mapean a la secuencia de ARNr 23S (otras mapean algunas proteínas ribosómicas 50S).
- Una preparación que consiste en ARNr 23S y algunos restos de proteínas de subunidad grande conserva la actividad peptidil-transferasa. Para mayor información, ver Noller et al. (1992) Resistencia inusual de peptidil-transferasa a procedimientos de extracción de proteínas. Ciencia 256:1416-1419.
- Se pueden seleccionar ARN de ribozima que catalizan la formación de enlaces peptídicos. En este experimento, los investigadores iniciaron con un pool de 1.3 '1015 ARN diferentes de 72 nucleótidos, flanqueados por regiones constantes. Permiten que esta gran población de ARN catalice una formación de enlaces peptídicos que agrega un aminoácido marcado con biotinilo (en un imitador químico de un sitio P) a un aminoácido conectado al ARN (en un imitador químico de un sitio A). Los ARN que catalizaron con éxito la reacción fueron extremadamente raros, pero ahora estaban unidos covalentemente a un marcador de biotina. De esta manera, podrían seleccionarse de la población mediante la unión a estreptavidina. Se utilizó PCR para amplificar los ARN exitosos y el procedimiento se repitió 19 veces. En este punto, los investigadores caracterizaron 9 ARN que catalizaron la reacción. Encontraron que estos ARN incrementaron la velocidad de reacción en un factor de 106 sobre la reacción no catalizada.
- La estructura tridimensional del ribosoma muestra que el sitio activo está compuesto por ARN. Se ha determinado la estructura de un ribosoma cristalizado con un inhibidor dirigido al sitio activo, así como la estructura sin el inhibidor. Esto permitió a los investigadores ver con precisión dónde se encuentra el sitio activo de la peptidil-transferasa dentro de la estructura. Solo se ve ARN alrededor de este sitio. La proteína más cercana está a 20 Angstroms de distancia, demasiado lejos para participar en la catálisis.
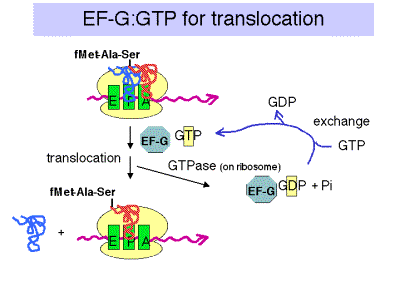
3. Translocación
a. La etapa de translocación mueve el ribosoma otros 3 nucleótidos aguas abajo (un codón) y mueve el peptidil-ARNt al sitio P (posición n), el ARNt desacilado sale a través del sitio E y el sitio A (posición n+1) está vacante para otra ronda de elongación.
b. Factor de Alargamiento G = EF‑G
- Esta es otra proteína muy abundante, con alrededor de 20 mil copias por célula, lo que equivale al número de ribosomas.
- EF‑G‑GTP se une al ribosoma para ayudar a la translocación, y se libera tras la hidrólisis de GTP (la GTPasa es de algún componente ribosómico).
- Estudios estructurales recientes (de A. Dahlberg y colegas) muestran que EF-G en el estado unido a GTP tiene una forma similar a la del complejo ternario de EF-Tu, GTP y aminoacil-ARNt. Al igual que este último complejo ternario, EF-G en el estado unido a GTP también tiene una alta afinidad por el sitio A en el ribosoma. Esto puede ayudar a impulsar el movimiento del peptidil-ARNt desde el sitio A al sitio P, reemplazándolo con EF-G (GTP) en el sitio A.
c. La hidrólisis de GT P es necesaria para la disociación de EF‑G después de la translocación. La GTPasa es parte del ribosoma, no EF-G.
Figura 3.5.20.
d. La acción del ácido fusídico reveló la necesidad de liberar EF‑G‑PIB. En presencia de ácido fusídico, EF‑G‑GTP se une al ribosoma, el GTP se hidroliza y el ribosoma mueve tres nucleótidos. Pero el complejo ribosoma-EF‑G‑PIB se estabiliza con este compuesto y se detiene la traducción.
e. Los ribosomas no pueden unirse simultáneamente a EF‑Tu y EF‑G. EF‑Tu debe terminar su acción antes de que EF‑G pueda actuar, y EF‑G debe completar su ciclo antes de que EF‑Tu pueda actuar de nuevo para introducir otro aminoacil-ARNt.
f. Efecto de la toxina de la difteria
- El análogo eucariota a EF‑G es eEF2, que también es una translocasa dependiente de la hidrólisis de GTP. También está bloqueado por el ácido fusídico.
- La toxina de Diptheria catalizará la adición de ADP-ribosa (del sustrato NAD+) a eEF2, inactivándola así. El objetivo para ADP-ribosilación es histidina modificada que se encuentra en EEF2 de muchas especies.
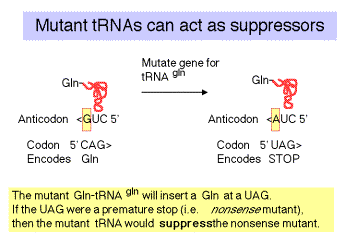
c. El “lado negativo” de la supresión sin sentido es que el ARNt supresor puede actuar en cualquier codón ámbar, por lo tanto compite con los factores liberadores en el reconocimiento de los codones de terminación normales. Cuando se usa el ARNt supresor en lugar de liberar factores, la traducción avanza más abajo del ARNm de lo que se supone, lo que lleva a la producción de proteínas aberrantes. Las cepas supresoras de E. coli pueden estar bastante enfermas (es decir, no crecen tan bien como las cepas de tipo silvestre).
d. Otros dos supresores ámbar están codificados por el gen supD, que codifica un ARNt que insertará Ser en una UAG, y suPF, que insertará Tyr.
3. Supresores de sentido erróneo: Se trata de ARNt mutantes que conducen a la inserción de un aminoácido que es compatible con el aminoácido de tipo silvestre (alterado por la mutación original).
4. Supresores del desplazamiento de rameshift F: Estos son ARNt mutantes cuyo anticodón se ha expandido (¿o contraído?) para que coincida con la mutación que altera la longitud en el ARNm.
Por ejemplo, considere una mutación original 5'GGG ‑> 5'GGGG (inserte una G).
Un supresor de desplazamiento de marco también tendría una C adicional en el anticodón.
anticodón de ARNt wt 3'CCC ‑‑> ARNt supresor 3'CCCC.


