15: Control positivo y negativo de la expresión génica
- Page ID
- 58757
Operones
Un operón es un grupo de genes regulados coordinadamente. Incluye genes estructurales (que generalmente codifican enzimas), genes reguladores (codificantes, por ejemplo, activadores o represores) y sitios reguladores (como promotores y operadores). El tipo de control se define por la respuesta del operón cuando no hay proteína reguladora presente. En el caso del control negativo, los genes en el operón se expresan a menos que sean desconectados por una proteína represora. Así, el operón se encenderá constitutivamente (los genes se expresarán) cuando el represor esté inactivado. En el caso del control positivo, los genes se expresan solo cuando está presente una proteína reguladora activa, por ejemplo, un activador. Así, el operón se apagará cuando la proteína reguladora positiva esté ausente o inactivada.
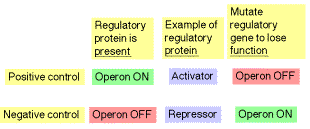
Cuadro 4.1.1. Control positivo vs negativo
Operones catabólicos versus biosintéticos
Las vías catabólicas catalizan la descomposición de los nutrientes (el sustrato para la vía) para generar energía, o más precisamente ATP, la moneda de energía de la célula. En ausencia del sustrato, no hay razón para que las enzimas catabólicas estén presentes, y el operón que las codifica es reprimido. En presencia del sustrato, cuando se necesitan las enzimas, el operón es inducido o desreprimido.
| Operón codifica | Ausencia de | Efecto | Presencia de | Efecto |
|---|---|---|---|---|
| enzimas catabólicas | sustrato | reprimida | sustrato | desreprimidos (inducidos) |
| enzimas biosintéticas | producto | indujo | producto | reprimida |
Por ejemplo, el operón lac codifica las enzimas necesarias para la captación (lactosa permeasa) y la descomposición inicial de la lactosa (el disacárido B-D-galactosil-1->4-D-glucosa) en galactosa y glucosa (catalizada por b-galactosidasa). Estos monosacáridos se descomponen en lactato (principalmente vía glucólisis, produciendo ATP), y de lactato a CO2 (a través del ciclo del ácido cítrico), produciendo NADH, que se alimenta a la cadena de transporte de electrones para producir más ATP (fosforilación oxidativa). Esto puede proporcionar la energía para que viva la célula bacteriana. Sin embargo, las enzimas iniciales (lactosa permeasa y b-galactosidasa) solo son necesarias, y solo se expresan, en presencia de lactosa y en ausencia de glucosa. En presencia del sustrato lactosa, el operón se enciende, y en su ausencia, el operón se apaga.
Las vías anabólicas o biosintéticas utilizan energía en forma de ATP y equivalentes reductores en forma de NAD (P) H para catalizar la síntesis de componentes celulares (el producto) a partir de materiales más simples, por ejemplo, síntesis de aminoácidos a partir de ácidos dicarboxílicos pequeños (componentes de la ciclo del ácido cítrico). Si la célula ya tiene bastante del producto (en presencia del producto), no se necesitan las enzimas que catalizan su síntesis, y se reprime el operón que las codifica. En ausencia del producto, cuando la célula necesita hacer más, se induce el operón biosintético. Por ejemplo, el operón trp codifica las enzimas que catalizan la conversión del ácido corismico en triptófano. Cuando la concentración celular de Trp (o Trp-TrnaTP) es alta, el operón no se expresa, pero cuando los niveles son bajos, se expresa el operón.
Operones inducibles versus reprimibles
Los operones inducibles se encienden en respuesta a un metabolito (una pequeña molécula sometida al metabolismo) que regula el operón. Por ejemplo, el operón lac se induce en presencia de lactosa (a través de la acción de un subproducto metabólico alolactosa). Los operones reprimibles se apagan en respuesta a una pequeña molécula reguladora. Por ejemplo, el operón trp es reprimido en presencia de triptófano. Tenga en cuenta que en este uso, los términos son definidos por la respuesta a una molécula pequeña. Aunque lac es un operón inducible, veremos condiciones bajo las cuales es reprimido o inducido (vía desrepresión).
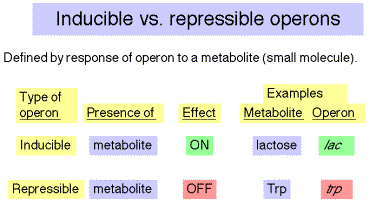
Cuadro 4.1.3.
Mapa del operón E. colilac
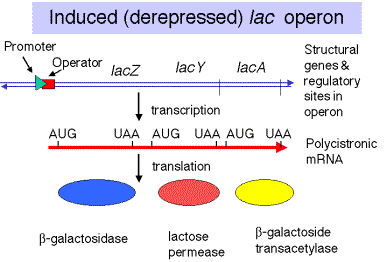
- Promotores = p = sitios de unión para ARN polimerasa a partir de los cuales inicia la transcripción. Existen promotores separados para el gen lacI y los genes lacZyA.
- Operador = o = sitio de unión para represor; se solapa con el promotor para lacZyA.
- Represor codificado por el gen lacI
- Genes estructurales: lacZyA
LacZ codifica b-galactosidasa, que escinde el discárido lactosa en galactosa y glucosa.
LacY codifica la lactosa permeasa, una proteína de membrana que facilita la captación de lactosa.
LacA codifica b-galactósido transacetilasa; la función de estas enzimas en el catabolismo de la lactosa no se entiende (al menos por mí)
C. Control negativo
El operón lac está bajo control negativo y positivo. Los mecanismos para estos se considerarán por separado.
1. En control negativo, los genes lacZyA son desactivados por represor cuando el inductor está ausente (señalando una ausencia de lactosa). Cuando el tetrámero represor se une a o, lacZya no se transcribe y por lo tanto no se expresa.
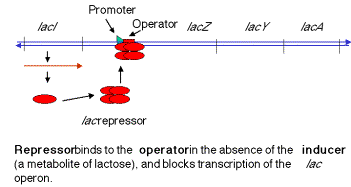
2. Cuando el inductor está presente (señalando la presencia de lactosa), se une a la proteína represora, alterando así su conformación, disminuyendo su afinidad por o, el operador. La disociación del complejo represor-inductor permite transcribir y expresar lacZya.
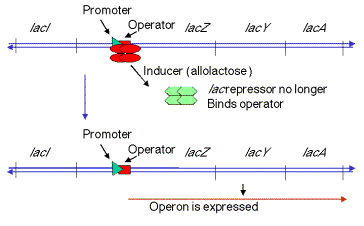
Inductores
El inductor natural (o antirepresor), es la alolactosa, un análogo de la lactosa. Se elabora como un subproducto metabólico de la reacción catalizada por la b-galactosidasa. Por lo general, esta enzima cataliza la escisión de lactosa a galactosa + glucosa, pero ocasionalmente catalizará una isomerización para formar alolactosa, en la que la galacosa se une a C6 de glucosa en lugar de C4.
Un inductor gratuito inducirá el operón pero no será metabolizado por las enzimas codificadas; de ahí que la inducción se mantenga por más tiempo. Uno de los más comunes utilizados en el laboratorio es un análogo sintético de la lactosa llamado isopropiltiogalactósido (IPTG). En este compuesto el enlace b-galactosidico es a un tiol, que no es un sustrato eficiente para la b-galactosidasa.
E. Mutantes reguladores
Las mutaciones reguladoras afectan la cantidad de todas las enzimas codificadas por un operón, mientras que las mutaciones en un gen estructural afectan solo la actividad del polipéptido codificado (único).
Mutantes represores
- a. Las cepas de tipo silvestre (LacI+) son inducibles.
- b. La mayoría de las cepas con un represor defectuoso (lacI-) son constitutivas, es decir, producen las enzimas codificadas por el operón lac incluso en ausencia del inductor.
- c. Las cepas con represor que no es capaz de interactuar con el inductor (LacI S) no son inducibles. Dado que el inductor no puede unirse, el represor permanece en el operador e impide la expresión del operón incluso en presencia de inductor.
- d. Deducciones basadas en fenotipos de mutantes
| b-galactosidasa | transacetilasa | ||||
|---|---|---|---|---|---|
| Genotipo | -IPTG | +IPTG | -IPTG | +IPTG | Conclusión |
| I+Z+A+ | <0.1> | 100 | <1> | 100 | Inducibles |
| I+Z-A+ | <0.1> | <0.1> | <1> | 100 | LacZ codifica b-galactosidasa |
| I-Z+A+ | 100 | 100 | 90 | 90 | Constitutivo |
| I+Z-A+ /F 'I-Z+A+ | <0.1> | 100 | <1> | 200 | I+ > I- en trans |
| ISZ+A+ | <0.1> | <1> | <1> | <1> | No inducible |
| ISZ+A+ /F 'I+Z+A+ | <0.1> | 1 | <1> | 1 | Es >. I+ en trans |
- El operón de tipo silvestre es inducible por IPTG.
- Una mutación en lacZ afecta solo a la b-galactosidasa, no a la transacetilasa (u otros productos del operón), demostrando que lacZ es un gen estructural.
- Una mutación en lacI afecta a ambas enzimas, de ahí que lacI sea un gen regulador. Ambos se expresan en ausencia del inductor, de ahí que el operón se exprese constitutivamente (la cepa muestra un fenotipo constitutivo).
- En una cepa merodiploide, en la que una copia del operón lac está en el cromosoma y otra copia está en un factor F', se puede probar el dominio de un alelo sobre otro. El LacI + de tipo silvestre es dominante sobre LacI- en trans. En una situación en la que el único gen lacZ funcional se encuentra en el mismo cromosoma que lacI-, el lacI funcional todavía causa represión en ausencia de inductor.
- El alelo LacI S no es inducible.
- En un merodiploide, el alelo LacI S es dominante sobre el tipo silvestre en tran s.
e. El hecho de que el producto del gen lacI sea de acción trans significa que es una molécula difusible que puede codificarse en un cromosoma pero actuar sobre otro, como el cromosoma F' en el ejemplo (d) anterior. De hecho, el producto del gen lacI es una proteína represora.
2. Mutantes de operador
a. Los defectos en el operador conducen a la expresión constitutiva del operón, de ahí que se puedan aislar mutaciones constitutivas del operador, abreviadas o c. El o + de tipo silvestre es inducible.
b. Las mutaciones en el operador son de acción cis; solo afectan la expresión de genes estructurales en el mismo cromosoma.
(1) El merodiploide I + o c Z +/I + o + Z - [esta es una abreviatura de LacI + o c LacZ +/LacI + o + LacZ -] expresa b-galactosidasa constitutivamente. Así o c es dominante a o + cuando o c está en cis a LacZ+.
(2) El merodiploide I + o c Z -/I + o + Z + es inducible para la expresión de b-galactosidasa. Así o + es dominante a o c cuando o + está en cis a lacZ +.
(3) El alelo de o que está en cis al gen informador activo (es decir, en el mismo cromosoma que lacZ + en este caso) es aquel cuyo fenotipo se ve. Por lo tanto, el operador actúa en cis, y esta propiedad se conoce como cis- dominancia. Como en la mayoría de los casos de secuencias reguladoras cis, estos son sitios en el ADN que se requieren para la regulación. En este caso el operador es un sitio de unión para la proteína represora de acción trans.
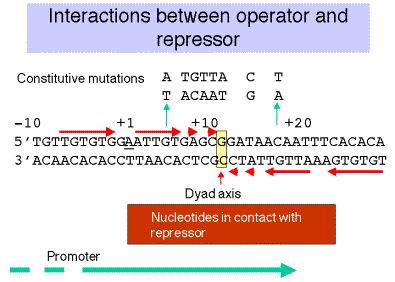
Interacciones entre Operador y Represor
Secuencia de operador
El operador solapa el inicio del sitio de transcripción y el promotor. Tiene una simetría de díada centrada en +11. Tal simetría díada se encuentra comúnmente dentro de los sitios de unión para proteínas simétricas (el represor es un homotetrámero). La secuencia de ADN que consitituta al operador se definió por la posición de las mutaciones o C, así como los nucleótidos protegidos de la reacción con, por ejemplo, DMS, tras la unión del represor.
2. Represor
a. Purificación
(1) Aumentar la cantidad de represor en el material de partida por sobreexpresión.
Una célula de tipo silvestre tiene solo alrededor de 10 moléculas del tetrámero represor. El aislamiento y purificación de la proteína se vio muy favorecido por el uso de una cepa mutante con mutaciones promotoras para LacI, por lo que muchas más copias de la proteína estuvieron presentes en cada célula. Esta estrategia general de sobreproducción de la proteína es ampliamente utilizada en esquemas de purificación. Ahora el gen para la proteína se clona en un vector de expresión, de modo que el huésped (bacteria en este caso) produce una gran cantidad de la proteína, a menudo una fracción sustancial de la proteína bacteriana total.
(2) Ensayos para represor
[1] Unión de IPTG radiomarcado (inductor gratuito) al represor
[2] Unión de la secuencia de ADN operadora radiomarcada al represor. Esto puede ser monitoreado por la capacidad del complejo proteína-ADN para unirse a nitrocelulosa (mientras que un fragmento de ADN operador mutante radiomarcado, o c, más represor no se unirá). Los ensayos de desplazamiento de movilidad electroforética se usarían ahora en muchos casos.
[3] Esta capacidad de secuencias particulares para unirse con alta afinidad a la proteína deseada se explota frecuentemente para aislar rápidamente la proteína. El sitio de unión puede sintetizarse como oligonucleótidos dúplex. Estos se ligan entre sí para formar multímeros, los cuales luego se unen a un sustrato sólido en una columna. La proteína de unión al ADN deseada puede aislarse entonces por cromatografía de afinidad, usando el sitio de unión en el ADN como ligando de afinidad.
b. El represor funcional aislado es un tetrámero; cada uno de los cuatro monómeros es el producto del gen lacI (es decir, es un homotetrámero).
c. El dominio de unión a ADN del represor lac se pliega en un dominio hélice-giro-hélice. Examinaremos este dominio estructural en más en el Capítulo III. Es uno de los dominios de unión a ADN más comunes en procariotas, y un dominio estructural similar (el homeodominio) se encuentra en algunos reguladores transcripcionales eucariotas.
3. Puntos de contacto entre represor y operador
a. La investigación de los puntos de contacto entre el represor y el operador utilizó las mismas técnicas que discutimos anteriormente para mapear el sitio de unión de la ARN polimerasa en el promotor, por ejemplo, ensayos de desplazamiento de movilidad electroforética (¿se une el fragmento de ADN?) , huellas de DNasa (¿dónde se une la proteína?) y ensayos de interferencia de metilación (¿metilación de qué purinas evitarán la unión?). Esquemas alternativos permitirán identificar sitios en los que la metilación es prevenida o potenciada por la unión del represor. Estas técnicas proporcionan una definición bioquímica del operador = sitio de unión para represor.
b. Los puntos de contacto clave (ver Figura 4.1.4. ):
(1) están dentro de la simetría de la díada.
(2) coinciden (en muchos casos) con nucleótidos que cuando mutan conducen a la expresión constitutiva. Obsérvese que este último es una definición genética del operador, y coincide con el operador bioquímico definido.
(3) tienden a distribuirse simétricamente alrededor del eje de la díada (+11).
(4) se encuentran en gran parte en una cara de la doble hélice del ADN.
c. El solapamiento parcial entre el operador y el promotor inicialmente sugirió un modelo de interferencia estérica para explicar el mecanismo de represión. Mientras un represor estuviera unido al operador, la polimerasa no pudo unirse al promotor. Pero, como se explorará en el próximo capítulo, no es así. La ARN polimerasa puede unirse al promotor lac incluso cuando el represor es boudn al operador lac. Sin embargo, la polimerasa no puede iniciar la transcripción cuando se yuxtapone al represor.
4. Desplazamiento conformacional en el represor cuando el inductor se une
a. El represor tiene dos dominios diferentes, uno que se une al ADN (“cabeza” que contiene el dominio hélice-vuelta-hélice) y otro que se une al inductor (y otras subunidades) (llamado el “núcleo). Estos están conectados por una región de “bisagra”.
b. Estos dominios estructurales pueden distinguirse por los fenotipos de mutaciones que ocurren en ellos.
LacI-d evita la unión al ADN, conduce a la expresión constitutiva.
LacI S evita la unión del inductor, conduce a un fenotipo no inducible.
c. La unión del inductor al “núcleo” provoca un desplazamiento alostérico en el represor de manera que la “cabeza” ya no es capaz de formar un complejo de alta afinidad con el ADN, y el represor puede disociarse (ir a uno de los muchos sitios inespecíficos competidores).
Control positivo: “represión de catabolitos”
1. Represión catabolita
a. Incluso las bacterias pueden ser quisquillosas con lo que comen. La glucosa es la fuente preferida de carbono para E. coli; la bacteria consumirá la glucosa disponible antes de utilizar fuentes alternativas de carbono, como lactosa o aminoácidos.
b. La glucosa conduce a la represión de la expresión de lac y algunos otros operones catabólicos. A este fenómeno se le llama represión catabolita.
2. Se necesitan dos componentes para esta forma de regulación
a. AMPc
[1] En presencia de glucosa, el [cAMP] dentro de la célula disminuye de 10-4 M a 10-7 M. Un alto [AMPc] aliviará la represión de catabolitos.
[2] La síntesis de cAMP es catalizada por la adenilato ciclasa (producto del gen cya)
ATP® AMP+ PP i
b. Proteína Activadora de Catabolitos = CAP
[1] Producto del gen cap, también llamado crp (proteína receptora cAMP).
[2] Es un dímero
[3] Se une a AMPc, y luego el complejo cAMP CAP se une al ADN en sitios específicos
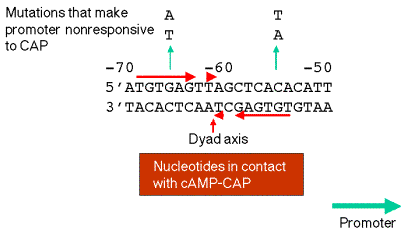
3. Sitio de unión para cAMP
a. En el operón lac, el sitio de unión es una región de aproximadamente 20 pb ubicada justo aguas arriba del promotor, de -52 a -72.
b. El pentámero TGTGA es un elemento esencial en el reconocimiento. Para el operón lac, el sitio de unión es una díada con esa secuencia en ambos lados de la díada.
c. Los puntos de contacto entre cAMP y el ADN son cercanos o coincidentes con mutaciones que hacen que el promotor lac ya no responda a cAMP-cap.
d. Camp-cap se une en una cara de la hélice.
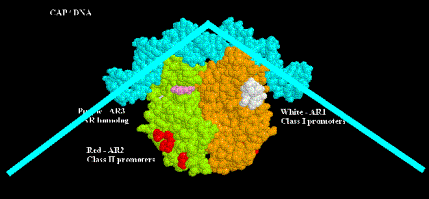
Figura 4.1.5. Sitio de unión para cAMP
4. La unión de cAMP-cap a su sitio potenciará la eficiencia de la iniciación de la transcripción en el promotor
a. El promotor lac no es un promotor particularmente fuerte. La secuencia a -10, TATGTT, no coincide con el consenso (TATAAT) en dos posiciones.
b. En presencia de cAMP, la ARN polimerasa iniciará la transcripción de manera más eficiente.
c. El promotor lac UV5 es una mutación promotora positiva en la que la región -10 coincide con el consenso. El operón lac impulsado por el promotor UV5 logrará un alto nivel de inducción sin cAMP, pero el promotor de tipo silvestre requiere cAMP-cap para inducción de alto nivel.
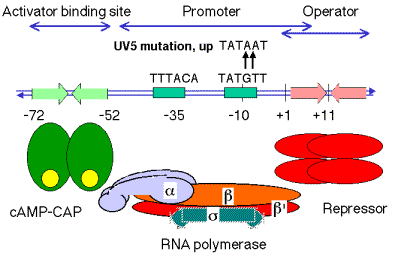
5. Modo de acción de Camp-cap
a. Interacción positiva directa con la ARN polimerasa. El extremo C de la subunidad a es necesario para que la ARN polimerasa sea activada por cAMP-cap. Esto se explorará con más detalle en el Capítulo 16.
b. Camp-cap dobla el ADN alrededor de 90 o.
Algunas generalidades
- Los represores, activadores y polimerasas interactúan principalmente con una cara de la doble hélice del ADN.
- Las proteínas reguladoras, como activadores y represores, son frecuentemente simétricas y se unen a secuencias simétricas en el ADN.
- Las ARN polimerasas no son simétricas, y los promotores a los que se unen también son asimétricos. Esto confiere direccionalidad a la transcripción.


