3.3: Regulación enzimática
- Page ID
- 55288
null Objetivos de aprendizaje
- Comparar y contrastar el control genético de la actividad enzimática (síntesis enzimática) en bacterias con el control de la actividad enzimática a través de la inhibición por retroalimentación.
- Comparar y contrastar un operón inducible con un operón reprimible y dar un ejemplo de cada uno.
- Compara cómo la presencia o ausencia de triptófano afecta al operón trp.
- Compara cómo la presencia o ausencia de lactosa afecta al operón lac.
- Compara cómo la presencia o ausencia de un inductor afecta a los activadores.
- Describa brevemente cómo los ARN pequeños pueden regular la actividad enzimática.
- Defina lo siguiente:
- represor
- inductor
- activador
- potenciador
- RNAs pequeños
- Comparar y contrastar la inhibición competitiva con la inhibición no competitiva.
En las células vivas, hay cientos de enzimas diferentes trabajando juntas de manera coordinada. Las células vivas no sintetizan ni descomponen más material del requerido para el metabolismo y crecimiento normales. Todo esto requiere mecanismos de control precisos para activar y desactivar las reacciones metabólicas. Existe una enorme diversidad en los mecanismos que utilizan las bacterias para regular la síntesis enzimática y la actividad enzimática. Para casi cada paso entre la activación de un gen y la reacción enzimática final de ese producto génico existe algún mecanismo bacteriano para la regulación de ese paso. Aquí veremos varios ejemplos bien estudiados.
Control Genético de la Síntesis Enzimática a través de la Represión, Inducción o Mejora
El control genético de la actividad enzimática se refiere al control de la transcripción del ARNm necesario para la síntesis de una enzima. En las células procariotas, esto implica la inducción, represión o potenciación de la síntesis enzimática por proteínas reguladoras que pueden unirse al ADN e inducir, bloquear o potenciar la función de la ARN polimerasa, la enzima requerida para la transcripción. Las proteínas reguladoras son a menudo parte de un operón o un regulón. Un operón es un conjunto de genes transcritos como un mensaje policistrónico que es controlado colectivamente por una proteína reguladora. Un regulón es un conjunto de genes relacionados controlados por la misma proteína reguladora pero transcritos como unidades monocistrónicas. Las proteínas reguladoras pueden funcionar como represores, activadores o potenciadores.
a. Represores
Los represores son proteínas reguladoras que bloquean la transcripción del ARNm. Hacen esto uniéndose a una porción de ADN llamada operador (a menudo a los operadores se les llama cajas ahora) que se encuentra aguas abajo de un promotor. La unión de la proteína reguladora al operador impide que la ARN polimerasa se una al promotor y transcriba la secuencia codificante de las enzimas. Esto se denomina control negativo y es principalmente n en reacciones biosintéticas donde una bacteria solo hace una molécula como un aminoácido particular cuando ese aminoácido no está presente en la célula.
Los represores son proteínas alostéricas que tienen un sitio de unión para una molécula específica. La unión de esa molécula al sitio alostérico del represor puede alterar la forma del represor que, a su vez, afecta su capacidad de unirse al ADN. Esto puede funcionar de una de dos maneras:
1. Algunos represores se sintetizan en una forma que por sí misma no puede unirse al operador. Esto se conoce como un sistema reprimible. La unión de una molécula llamada corepresora, sin embargo, altera la forma de la proteína reguladora a una forma que puede unirse al operador y posteriormente bloquear la transcripción. Un ejemplo de este tipo de sistema reprimible es el operón trp en Escherichia coli que codifica las cinco enzimas en la ruta para la biosíntesis del aminoácido triptófano. En este caso, la proteína represora codificada por el gen regulador trp, normalmente no se une a la región operadora del operón trp y se elaboran las cinco enzimas necesarias para sintetizar el aminoácido triptófano (Figura\(\PageIndex{1}\) A y Figura\(\PageIndex{1}\) B).
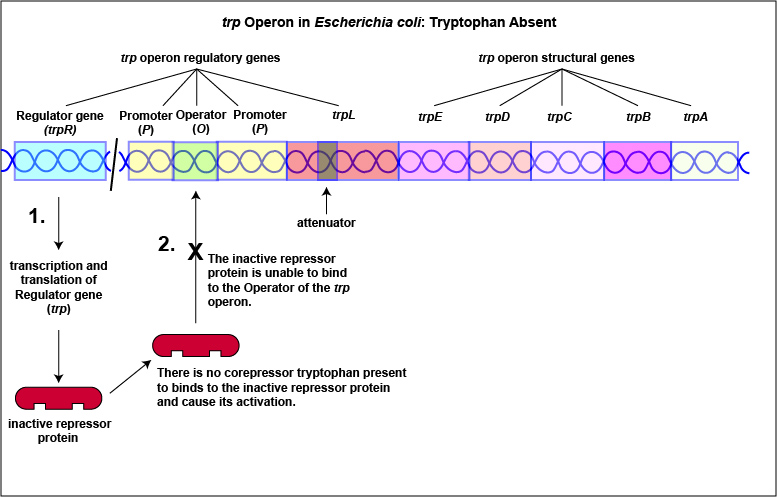
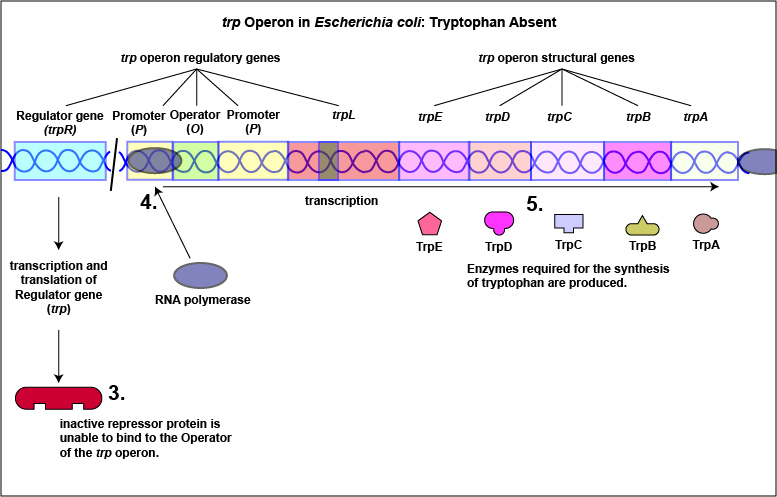
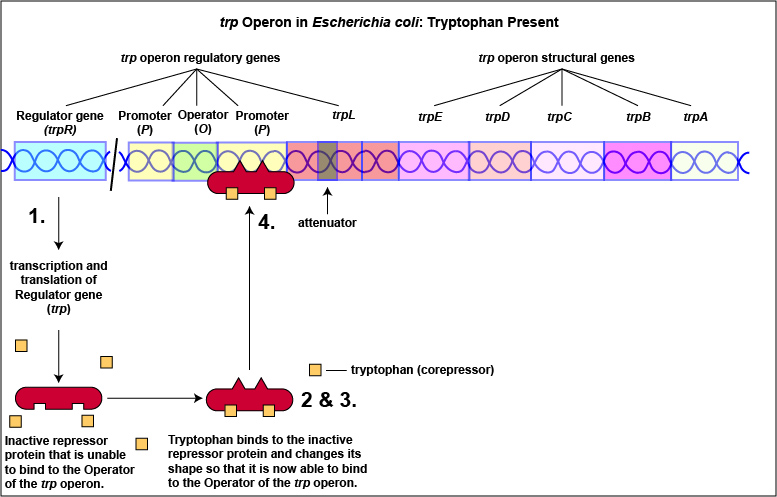
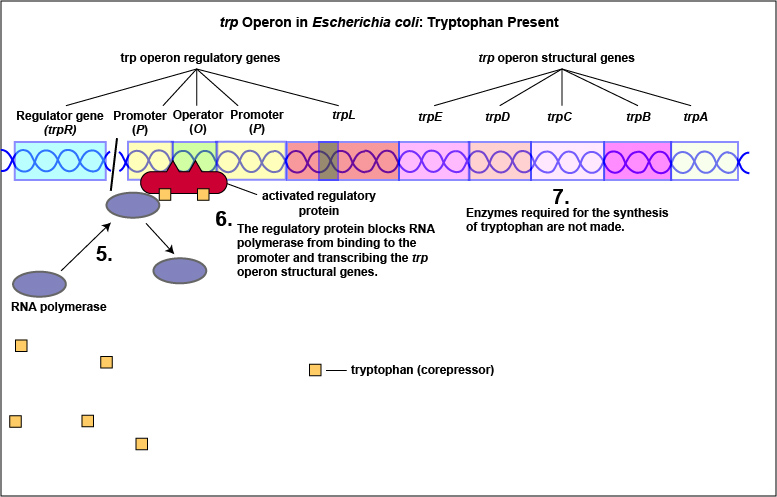
El triptófano, el producto final de estas reacciones enzimáticas, sin embargo, funciona como un corepresor. Una vez que se ha sintetizado suficiente triptófano, la célula necesita terminar su síntesis. El triptófano es capaz de unirse a un sitio en la proteína represora alostérica, cambiando su forma y permitiéndole interactuar con la región operadora trp. Una vez que el represor se une al operador, la ARN polimerasa es incapaz de unirse al promotor y transcribir los genes para la biosíntesis de triptófano. Por lo tanto, cuando hay suficiente triptófano, se desactiva la transcripción de las enzimas que permiten su biosíntesis (Figura\(\PageIndex{2}\) A y Figura\(\PageIndex{2}\) B).
Además de la represión, la expresión del operón trp también está regulada por la atenuación. El gen trpL codifica una secuencia líder de ARNm que controla la expresión del operón a través de la atenuación. Este ARNm de secuencia líder consiste en los dominios 1, 2, 3 y 4. El dominio 3 puede emparejarse con el dominio 2 o el dominio 4.
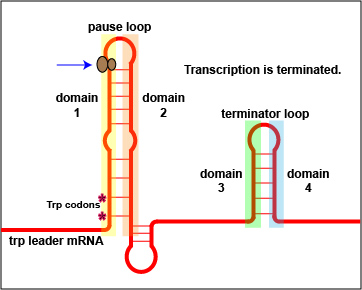
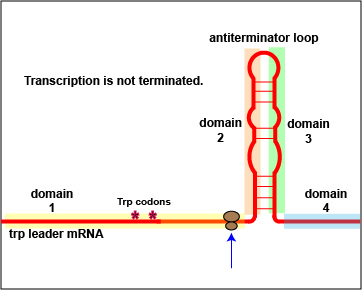
A altas concentraciones de triptófano, los dominios 3 y 4 se emparejan de tal manera que forman estructuras de tallo y bucle que bloquean la transcripción del resto del ARNm de la secuencia líder y posteriormente, la transcripción de los genes estructurales para la biosíntesis de triptófano (Figura\(\PageIndex{3}\) A). Sin embargo, a bajas concentraciones de triptófano, los dominios 3 y 2 se emparejan. Este emparejamiento permite la transcripción completa del ARNm de la secuencia líder, así como la de los genes estructurales para la biosíntesis de triptófano (Figura\(\PageIndex{3}\) B).
2. Otros represores se sintetizan en una forma que se une fácilmente al operador y bloquea la transcripción. Sin embargo, la unión de una molécula llamada inductor altera la forma de la proteína reguladora de una manera que ahora bloquea su unión al operador y así permite la transcripción. Esto se conoce como un sistema inducible.
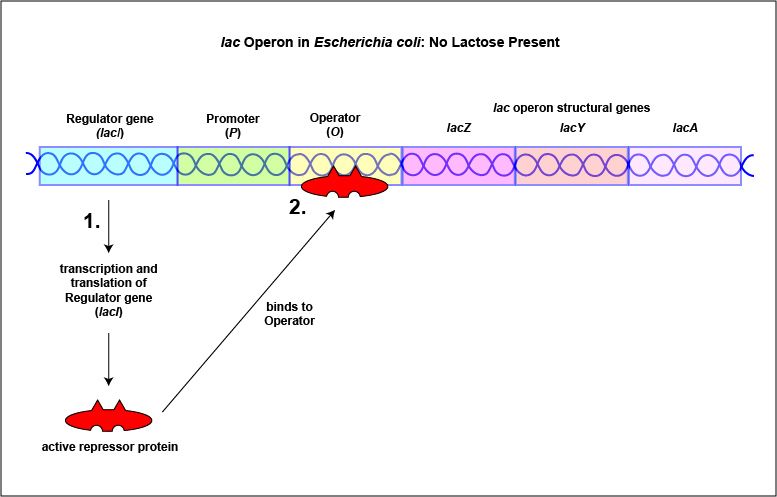
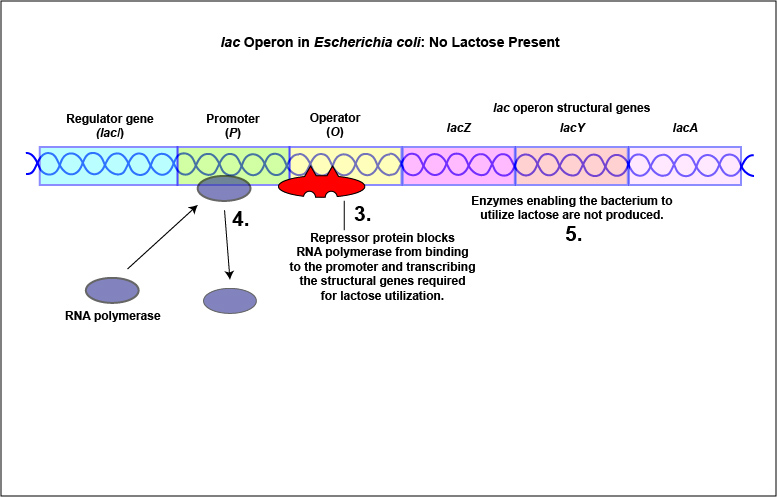
Un ejemplo de sistema inducible es el operón lac que codifica las tres enzimas necesarias para la degradación de la lactosa por E. coli. E. coli solo sintetizará las enzimas que requiere para utilizar lactosa si ese azúcar está presente en el ambiente circundante. En este caso, la lactosa funciona como inductor. En ausencia de lactosa, la proteína represora activa se une al operador y la ARN polimerasa es incapaz de unirse al promotor y transcribir los genes para la utilización de lactosa. Como resultado, no se sintetizan las enzimas necesarias para la utilización de lactosa (Figura\(\PageIndex{4}\) A y Figura\(\PageIndex{4}\) B). Cuando la lactosa, el inductor, está presente, un metabolito de la lactosa llamado alolactosa se une a la proteína represora alostérica y hace que cambie de forma de tal manera que ya no sea capaz de unirse al operador. Ahora la ARN polimerasa es capaz de transcribir los tres genes estructurales del operón lac y la bacteria es capaz de sintetizar las enzimas requeridas para la utilización de lactosa (Figura\(\PageIndex{5}\) A y Figura\(\PageIndex{5}\) B).
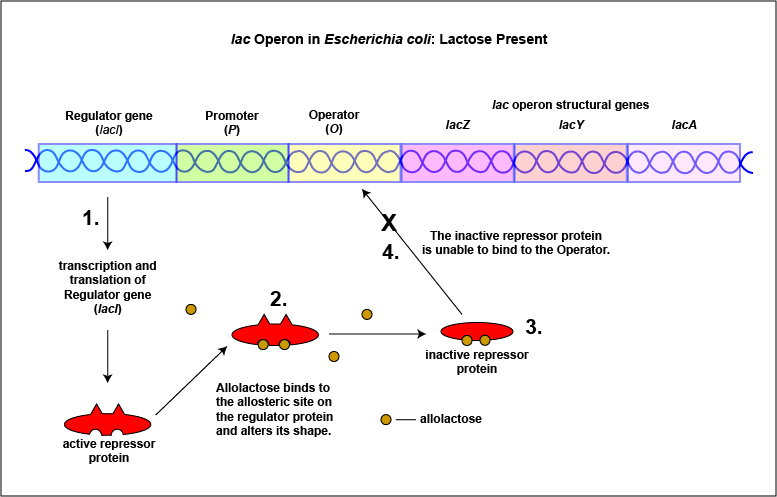
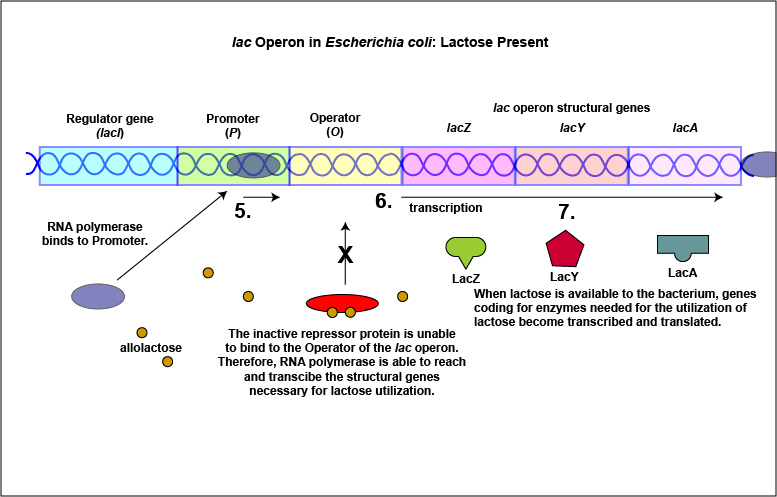
b. Activadores
Los activadores son proteínas reguladoras que promueven la transcripción del ARNm. Los activadores controlan los genes que tienen un promotor al que la ARN polimerasa no puede unirse. El promotor se encuentra adyacente a un segmento de ADN llamado el sitio de unión al activador. El activador es una proteína alostérica sintetizada en una forma que normalmente no puede unirse al sitio de unión al activador. Como resultado, la ARN polimerasa es incapaz de unirse al promotor y transcribir los genes (Figura\(\PageIndex{6}\)). Sin embargo, la unión de una molécula llamada inductor al activador altera la forma del activador de una manera que ahora le permite unirse al sitio de unión del activador. La unión del activador al sitio de unión al activador, a su vez, permite que la ARN polimerasa se una al promotor e inicie la transcripción (Figura\(\PageIndex{7}\) A y Figura\(\PageIndex{7}\) B). Esto se denomina control positivo y es principalmente n en reacciones catabólicas donde una bacteria solo produce enzimas para el catabolismo de un sustrato cuando ese sustrato está disponible para la célula.
c. Potenciadores
Los potenciadores son proteínas reguladoras que se unen al ADN ubicado a cierta distancia del operón que controlan al trabajar con proteínas dobladoras de ADN. Las proteínas de unión al ADN doblan el ADN de una manera que ahora permite que el potenciador interactúe con el promotor de tal manera que la ARN polimerasa ahora puede unirse e iniciar la transcripción (Figura\(\PageIndex{8}\)).
2. Control Genético de la Síntesis de Enzimas mediante Reconocimiento de Promotores y Superbobinado de ADN
a. Reconocimiento del promotor: Los factores sigma específicos que se unen a la ARN polimerasa determinan qué operón se transcribirá.
b. Superbobinado de ADN: El superenrollamiento de ADN puede cambiar la forma terciaria de una molécula de ADN de su forma normal a una que tiene un giro zurdo llamado Z-ADN. Las actividades de algunos promotores se disminuyen con Z-DNA mientras que otros se incrementan.
3. Control Genético de la Síntesis de Enzimas mediante el Control Traslacional de la Síntesis
a. Interferencia de ARN (ARNi)
La interferencia de ARN (ARNi) es un proceso mediante el cual pequeños ARN reguladores no codificantes (ARNnc) tales como microARN (miRNAs) regulan la expresión génica. Estos ARNnc son moléculas reguladoras que son complementarias a una porción temprana del extremo 5' del ARNm que codifica la enzima. Cuando el ARN pequeño se une al ARNm por emparejamiento de bases complementarias, los ribosomas no pueden unirse al ARNm bloqueando su traducción. Como resultado, la enzima no se elabora (Figura\(\PageIndex{9}\)). En las bacterias, estos ARNnc a menudo se denominan ARN pequeños (ARNs); en las células animales, las células vegetales y los virus a menudo se les llama microARN (miARN).
b. Proteínas ribosómicas (r-proteínas)
Las proteínas ribosómicas se unen al ARNr para formar subunidades ribosómicas. Debido a que la secuencia de bases nucleotídicas para el ARNm que codifica las proteínas r tiene similitudes con la del ARNr al que se une esa proteína r durante la formación de la subunidad, las proteínas r que aún no están incorporadas en las subunidades ribosómicas pueden unirse a ese ARNm y bloquear la traducción
4. Control de la actividad de la enzima (inhibición de retroalimentación).
La actividad enzimática puede controlarse mediante inhibición competitiva e inhibición no competitiva.
a. Con lo que se denomina inhibición no competitiva, el inhibidor es el producto final de una vía metabólica que es capaz de unirse a un segundo sitio (el sitio alostérico) en la enzima. La unión del inhibidor al sitio alostérico altera la forma del sitio activo de la enzima, evitando así la unión del primer sustrato en la vía metabólica. De esta manera, se apaga el camino (Figura\(\PageIndex{10}\)).
b. En el caso de lo que se denomina inhibición competitiva, el inhibidor es el producto final de una reacción enzimática. Ese producto final también es capaz de reaccionar con el sitio activo de la enzima y evita que la enzima se una a su sustrato normal. Como resultado, el producto final ya no se sintetiza (Figura\(\PageIndex{11}\)).
Resumen
- En las células vivas hay cientos de enzimas diferentes trabajando juntas de manera coordinada, y dado que las células no sintetizan ni descomponen más material del requerido para el metabolismo y crecimiento normales, se requiere una regulación enzimática precisa para activar y desactivar las reacciones metabólicas.
- Existe una enorme diversidad en los mecanismos que utilizan las bacterias para regular la síntesis enzimática y la actividad enzimática.
- Las formas en que las enzimas pueden controlarse o regularse incluyen controlar la síntesis de la enzima (control genético) y controlar la actividad de la enzima (inhibición por retroalimentación).
- En procariotas, el control genético de la actividad enzimática incluye la inducción o represión de la síntesis enzimática por proteínas reguladoras que pueden unirse al ADN y bloquear o potenciar la función de la ARN polimerasa, la enzima requerida para la transcripción.
- Un operón es un conjunto de genes controlados colectivamente por una proteína reguladora.
- Las proteínas reguladoras pueden funcionar como represoras o activadoras.
- Los represores son proteínas reguladoras que bloquean la transcripción del ARNm al evitar que la ARN polimerasa transcriba la secuencia codificante de las enzimas.
- Algunos represores, como en el caso del operón trp, se sintetizan en una forma que por sí misma no puede unirse al operador. Esto se conoce como un sistema reprimible. La unión de una molécula llamada corepresora, sin embargo, altera la forma de la proteína reguladora a una forma que puede unirse al operador y posteriormente bloquear la transcripción.
- Algunos represores, como en el caso del operón lac, se sintetizan en una forma que se une fácilmente al operador y bloquea la transcripción. Sin embargo, la unión de una molécula llamada inductor altera la forma de la proteína reguladora de una manera que ahora bloquea su unión al operador y así permite la transcripción. Esto se conoce como un sistema inducible.
- Los activadores son proteínas reguladoras que promueven la transcripción de ARNm al permitir que la ARN polimerasa transcriba la secuencia codificante de las enzimas.
- Los potenciadores son proteínas reguladoras que se unen al ADN ubicado a cierta distancia del operón que controlan al trabajar con proteínas dobladoras de ADN. Las proteínas dobladoras de ADN doblan el ADN de una manera que ahora permite que el potenciador interactúe con el promotor de tal manera que la ARN polimerasa ahora pueda unirse e iniciar la transcripción
- Las bacterias también utilizan el control traslacional de la síntesis enzimática. Un método es que las bacterias produzcan moléculas de ARN no codificantes (ARNnc) que sean complementarias al ARNm que codifica la enzima, y cuando el ARN pequeño se une al ARNm por emparejamiento de bases complementarias, los ribosomas no pueden unirse al ARNm, el ARNm no se transcribe y se traduce en proteína, y la enzima no se hace. En las bacterias, estos ARNnc a menudo se denominan ARN pequeños (ARNs).
- La inhibición de retroalimentación controla la actividad de la enzima en lugar de su síntesis y puede ser no competitiva o competitiva.
- En el caso de inhibición no competitiva, el inhibidor es el producto final de una vía metabólica que es capaz de unirse al sitio alostérico en la enzima. La unión del inhibidor al sitio alostérico altera la forma del sitio activo de la enzima, evitando así la unión del primer sustrato en la vía metabólica. De esta manera, se apaga el camino.
- En el caso de lo que se denomina inhibición competitiva, el inhibidor es el producto final de una reacción enzimática. Ese producto final también es capaz de reaccionar con el sitio activo de la enzima y evita que la enzima se una a su sustrato normal. Como resultado, el producto final ya no se sintetiza.


