13.1B: Estructura de anticuerpos
- Page ID
- 55464
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Describir una molécula de anticuerpo.
- Dibujar la estructura de “figura de palo” de IgG, indicando la porción Fab (región variable) y la porción Fc (región constante).
- Anotar las funciones de las porciones Fab y Fc de un anticuerpo.
- Declarar qué se entiende por la actividad biológica de un anticuerpo.
- Comparar la estructura de IgM e IgA secretora con la de IgG.
En esta sección veremos la estructura de los anticuerpos. Existen cinco clases o isotipos de anticuerpos humanos:
- inmunoglobulina G (IgG),
- inmunoglobulina M (IgM),
- inmunoglobulina A (IgA),
- inmunoglobulina D (IgD), y
- inmunoglobulina E (IgE).
Los anticuerpos más simples, como IgG, IgD e IgE, son macromoléculas en forma de “Y” llamadas monómeros. Un monómero está compuesto por cuatro cadenas glicoproteínicas: dos cadenas pesadas idénticas y dos cadenas ligeras idénticas. Las dos cadenas pesadas tienen un alto peso molecular que varía con la clase de anticuerpo. Las cadenas ligeras vienen en dos variedades: kappa o lambda y tienen un peso molecular menor que las cadenas pesadas. Las cuatro cadenas de glicoproteínas están conectadas entre sí por enlaces disulfuro (S-S) y enlaces no covalentes (Figura\(\PageIndex{1}\)).
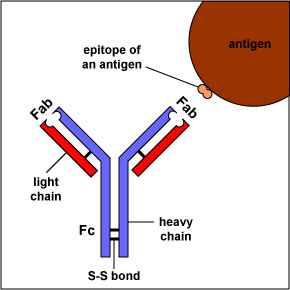
Los enlaces S-S adicionales pliegan las cadenas de glicoproteínas individuales en varios dominios globulares distintos (Figura\(\PageIndex{2}\)). El área donde la parte superior de la “Y” se une a la parte inferior se llama bisagra. Esta área es flexible para permitir que el anticuerpo se una a pares de epítopos a distintas distancias en un antígeno.
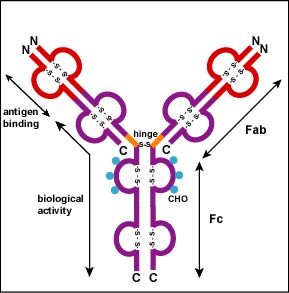
“Las dos puntas del monómero “" Y "” se denominan fragmentos de unión a antígeno o porciones Fab del anticuerpo (Figuras 1-3).” Los primeros 110 aminoácidos o el primer dominio tanto de la cadena pesada como de la cadena ligera de la región Fab del anticuerpo proporcionan especificidad para unirse a un epítopo en un antígeno. La secuencia de aminoácidos de este primer dominio tanto de la cadena ligera como de la cadena pesada muestra una tremenda variación de anticuerpo a anticuerpo y constituye la región variable (región V). Esto se debe a que cada linfocito B, temprano en su desarrollo, se programa genéticamente a través de una serie de reacciones de corte y empalme de genes para producir un Fab con una forma tridimensional única capaz de encajar algún epítopo con una forma correspondiente.
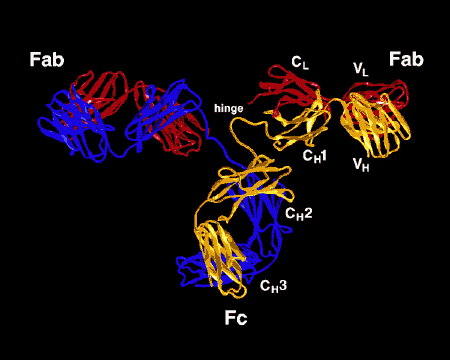
Figura\(\PageIndex{3}\): Dibujo en cinta de la molécula de anticuerpo IgG2a, Dibujo de cinta de la primera molécula de anticuerpo intacta jamás cristalizada (IgG2a). La porción Fab del anticuerpo tiene especificidad para unirse a un epítopo de un antígeno. La porción Fc dirige la actividad biológica del anticuerpo.
Los diversos genes que empalman las células determinan el orden de aminoácidos de la porción Fab tanto de la cadena ligera como de la pesada; la secuencia de aminoácidos determina la forma tridimensional final (Figura\(\PageIndex{4}\)). Por lo tanto, diferentes moléculas de anticuerpos producidas por diferentes linfocitos B tendrán diferentes órdenes de aminoácidos en las puntas del Fab para darles formas únicas para unirse al epítopo. El sitio de unión al antígeno es lo suficientemente grande como para contener un epítopo de aproximadamente 5-7 aminoácidos o 3-4 residuos de azúcar. Los epítopos se unen a la porción Fab del anticuerpo por enlaces reversibles no covalentes.
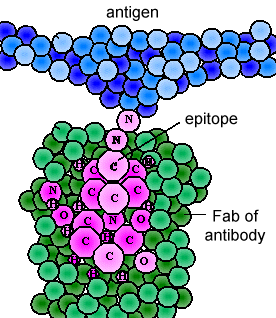
La parte inferior de la “Y”, la región C terminal de cada cadena de glicoproteína, se denomina porción Fc. La porción Fc, así como un dominio tanto de la cadena pesada como ligera de la región Fab tiene una secuencia de aminoácidos constante y se conoce como la región constante (región C) del anticuerpo y define la clase y subclase de cada anticuerpo. La porción Fc es responsable de la actividad biológica del anticuerpo (Figuras 1-3), sin embargo, la porción Fc solo se vuelve biológicamente activa después de que el componente Fab se haya unido a su antígeno correspondiente. Dependiendo de la clase y subclase de anticuerpo, las actividades biológicas de la porción Fc de los anticuerpos incluyen la capacidad de:
- Activar la vía clásica del complemento (IgG e IgM); ver Figura\(\PageIndex{5}\).
- Activar la vía del complemento de lectina y la vía alternativa del complemento (IgA)
- Se unen a receptores en fagocitos (IgG); ver Figura\(\PageIndex{6}\).
- Se unen a receptores en mastocitos, basófilos y eosinófilos (IgE); ver Fig. 7 y Figura\(\PageIndex{8}\).
- Se unen a receptores en células NK (IgG); ver Figura\(\PageIndex{9}\).
- Determinar la distribución tisular de los anticuerpos, es decir, a qué tipos de tejidos pueden ir las moléculas de anticuerpos.
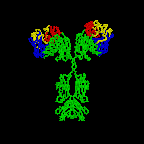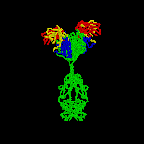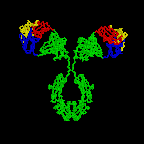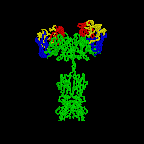
“Las moléculas de anticuerpos individuales en forma de “" Y "” se denominan monómeros y pueden unirse a dos epítopos idénticos.” Los anticuerpos de las clases IgG, IgD e IgE son monómeros.
Dos clases de anticuerpos son más complejas. IgM (ver Figura\(\PageIndex{10}\)) es un pentámero, que consiste en 5 moléculas similares a “Y” conectadas en sus porciones Fc por una “J” o cadena de unión. La IgA secretora (ver Figura\(\PageIndex{11}\)) es un dímero que consiste en 2 moléculas similares a “Y” conectadas en sus porciones Fc por una cadena “J” y estabilizadas para resistir la digestión enzimática en las secreciones corporales por medio de un componente secretor.
Resumen
- Existen 5 clases o isotipos de anticuerpos humanos o inmunoglobulinas: IgG, IgM, IgA, IgD e IgE.
- Los anticuerpos más simples, como IgG, IgD e IgE, son macromoléculas en forma de “Y” llamadas monómeros y están compuestas por cuatro cadenas glicoproteicas: dos cadenas pesadas idénticas y dos cadenas ligeras idénticas.
- “Las dos puntas del monómero “" Y "” se denominan fragmentos de unión a antígeno o porciones Fab del anticuerpo y estas porciones proporcionan especificidad para unirse a un epítopo en un antígeno.”
- Al principio de su desarrollo, cada linfocito B se programa genéticamente a través de una serie de reacciones de corte y empalme génico para producir un Fab con una forma tridimensional única capaz de encajar algún epítopo con una forma correspondiente.
- La porción Fc solo se vuelve biológicamente activa después de que el componente Fab se haya unido a su antígeno correspondiente. Las actividades biológicas incluyen activar las vías del complemento y unirse a receptores en fagocitos y otras células de defensa para promover la inmunidad adaptativa.
- IgM es un pentámero, que consiste en 5 monómeros unidos en sus porciones Fc.
- La IgA es un dímero, que consiste en 2 monómeros unidos en sus porciones Fc.
Preguntas
Estudie el material en esta sección y luego escriba las respuestas a estas preguntas. No se limite a hacer clic en las respuestas y escríbelas. Esto no pondrá a prueba tu comprensión de este tutorial.
- Describir una molécula de anticuerpo. (ans)
- Coinciden con lo siguiente:
_____ La región del anticuerpo que proporciona especificidad para unirse a un epítopo sobre un antígeno. (ans)
_____ La región del anticuerpo que es responsable de la actividad biológica del anticuerpo. (ans)
_____ Compuesto por cuatro cadenas de glicoproteínas. Hay dos cadenas pesadas idénticas que tienen un alto peso molecular y dos cadenas ligeras idénticas. (ans)
_____ Un pentámero, que consiste en 5 moléculas similares a “Y” conectadas en sus porciones Fc por una “J” o cadena de unión. (ans)
_____ Un dímero que consiste en 2 moléculas similares a “Y” conectadas en sus porciones Fc por una cadena “J” y estabilizadas para resistir la digestión enzimática. (ans)
- IgM
- IgA secretora
- IgG
- Fab
- Fc
- Opción múltiple (ans)


