7.13A: Estrategias utilizadas en proyectos de secuenciación
- Page ID
- 60741
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Las estrategias utilizadas para secuenciar genomas incluyen el método de Sanger, secuenciación de escopeta, final por pares y secuenciación de próxima generación.
OBJETIVOS DE APRENDIZAJE
Compare las diferentes estrategias utilizadas para la secuenciación del genoma completo: método de Sanger, secuenciación de escopeta, secuenciación de pares y secuenciación de próxima generación
Claves para llevar
Puntos Clave
- El método de Sanger es una técnica básica de secuenciación que utiliza didesoxinucleótidos marcados fluorescentemente (ddNTPs) durante la replicación del ADN, lo que da como resultado múltiples cadenas cortas de ADN replicado que terminan en diferentes puntos, con base en donde se incorporó el ddNTP.
- La secuenciación de escopeta es un método que corta aleatoriamente fragmentos de ADN en trozos más pequeños y luego, con la ayuda de una computadora, toma los fragmentos de ADN, los analiza en busca de secuencias superpuestas y vuelve a ensamblar toda la secuencia de ADN.
- La secuenciación de pares es un tipo de secuenciación de escopeta que se usa para genomas más grandes y analiza ambos extremos de los fragmentos de ADN para superponerse.
- La secuenciación de próxima generación es un tipo de secuenciación automatizada y se basa en un software sofisticado para la secuenciación rápida del ADN.
Términos Clave
- fluoróforo: una molécula o grupo funcional que es capaz de fluorescencia
- cóntigo: un conjunto de segmentos de ADN superpuestos, derivados de una única fuente de material genético, de los cuales se puede deducir la secuencia completa
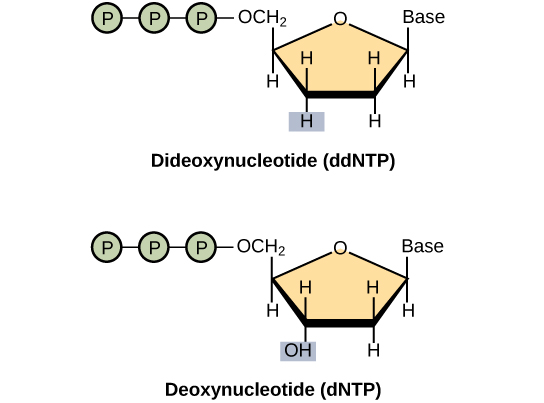
- didesoxinucleótidos: cualquier nucleótido formado a partir de un desoxinucleótidos por pérdida de un segundo grupo hidroxilo del grupo desoxirribosa
Estrategias utilizadas en proyectos de secuenciación
La técnica básica de secuenciación utilizada en todos los proyectos de secuenciación modernos es el método de terminación de cadena (también conocido como el método didesoxilo), que fue desarrollado por Fred Sanger en la década de 1970. El método de terminación de cadena implica la replicación del ADN de un molde monocatenario con el uso de un cebador y un desoxinucleótidos regulares (dNTP), que es un monómero, o una sola unidad, de ADN. El cebador y dNTP se mezclan con una pequeña proporción de didesoxinucleótidos marcados fluorescentemente (ddNTPs). Los ddNTPs son monómeros a los que les falta un grupo hidroxilo (-OH) en el sitio en el que otro nucleótido generalmente se une para formar una cadena. Cada ddNTP está marcado con un color diferente de fluoróforo. Cada vez que se incorpora un ddNTP en la cadena complementaria en crecimiento, termina el proceso de replicación del ADN, lo que da como resultado múltiples cadenas cortas de ADN replicado que terminan cada una en un punto diferente durante la replicación. Cuando la mezcla de reacción se procesa por electroforesis en gel después de ser separada en cadenas simples, las múltiples cadenas de ADN recién replicadas forman una escalera debido a sus diferentes tamaños. Debido a que los ddNTPs están marcados fluorescentemente, cada banda en el gel refleja el tamaño de la cadena de ADN y el ddNTP que terminó la reacción. Los diferentes colores de los ddNTPs marcados con fluoróforo ayudan a identificar el ddNTP incorporado en esa posición. La lectura del gel sobre la base del color de cada banda en la escalera produce la secuencia de la hebra plantilla.

Estrategias tempranas: Secuenciación de escopeta y secuenciación de extremos por pares
En el método de secuenciación de escopeta, varias copias de un fragmento de ADN se cortan aleatoriamente en muchas piezas más pequeñas (algo así como lo que sucede con un cartucho de disparo redondo cuando se dispara desde una escopeta). Todos los segmentos se secuencian mediante el método de secuenciación en cadena. Después, con la ayuda de una computadora, se analizan los fragmentos para ver dónde se superponen sus secuencias. Al emparejar secuencias superpuestas al final de cada fragmento, se puede reformar la secuencia de ADN completa. Una secuencia más grande que se ensambla a partir de secuencias más cortas que se superponen se llama cóntigo. Como analogía, considera que alguien tiene cuatro copias de una fotografía de paisaje que nunca antes habías visto y no sabes nada de cómo debería aparecer. Luego, la persona rasga cada fotografía con sus manos, de manera que se presentan piezas de diferentes tamaños a partir de cada copia. Luego, la persona mezcla todas las piezas y te pide que reconstruya la fotografía. En una de las piezas más pequeñas se ve una montaña. En una pieza más grande, se ve que la misma montaña está detrás de un lago. Un tercer fragmento muestra solo el lago, pero revela que hay una cabaña en la orilla del lago. Por lo tanto, al mirar la información superpuesta en estos tres fragmentos, se sabe que la imagen contiene una montaña detrás de un lago que tiene una cabaña en su orilla. Este es el principio detrás de la reconstrucción de secuencias de ADN completas usando secuenciación de escopeta.
Originalmente, la secuenciación de escopeta solo analizó un extremo de cada fragmento en busca de superposiciones. Esto fue suficiente para secuenciar genomas pequeños. Sin embargo, el deseo de secuenciar genomas más grandes, como el de un ser humano, condujo al desarrollo de una secuenciación de escopeta de doble cañón, más formalmente conocida como secuenciación de pares finales. En la secuenciación de pares de extremos, ambos extremos de cada fragmento se analizan para determinar el solapamiento. La secuenciación por pares es, por lo tanto, más engorrosa que la secuenciación de escopeta, pero es más fácil reconstruir la secuencia porque hay más información disponible.
Secuenciación de próxima generación
Desde 2005, las técnicas de secuenciación automatizada utilizadas por los laboratorios están bajo el paraguas de la secuenciación de próxima generación, que es un grupo de técnicas automatizadas utilizadas para la secuenciación rápida del ADN. Estos secuenciadores automatizados y de bajo costo pueden generar secuencias de cientos de miles o millones de fragmentos cortos (de 25 a 500 pares de bases) en el lapso de un día. Se utiliza un software sofisticado para gestionar el engorroso proceso de poner todos los fragmentos en orden.


