11.5: Mutaciones
- Page ID
- 54596
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Comparar mutaciones puntuales y mutaciones de cambio de marco
- Describir las diferencias entre las mutaciones sin sentido, sin sentido y silenciosas
- Describir las diferencias entre la reparación de luz y oscuridad
- Explicar cómo actúan los diferentes mutágenos
- Explicar por qué se puede utilizar la prueba de Ames para detectar carcinógenos
- Analizar secuencias de ADN e identificar ejemplos de tipos de mutaciones
Una mutación es un cambio heredable en la secuencia de ADN de un organismo. El organismo resultante, llamado mutante, puede tener un cambio reconocible en el fenotipo en comparación con el tipo silvestre, que es el fenotipo más comúnmente observado en la naturaleza. Un cambio en la secuencia de ADN se confiere al ARNm a través de la transcripción, y puede conducir a una secuencia de aminoácidos alterada en una proteína en la traducción. Debido a que las proteínas llevan a cabo la gran mayoría de las funciones celulares, un cambio en la secuencia de aminoácidos en una proteína puede conducir a un fenotipo alterado para la célula y el organismo.
Efectos de las mutaciones en la secuencia de ADN
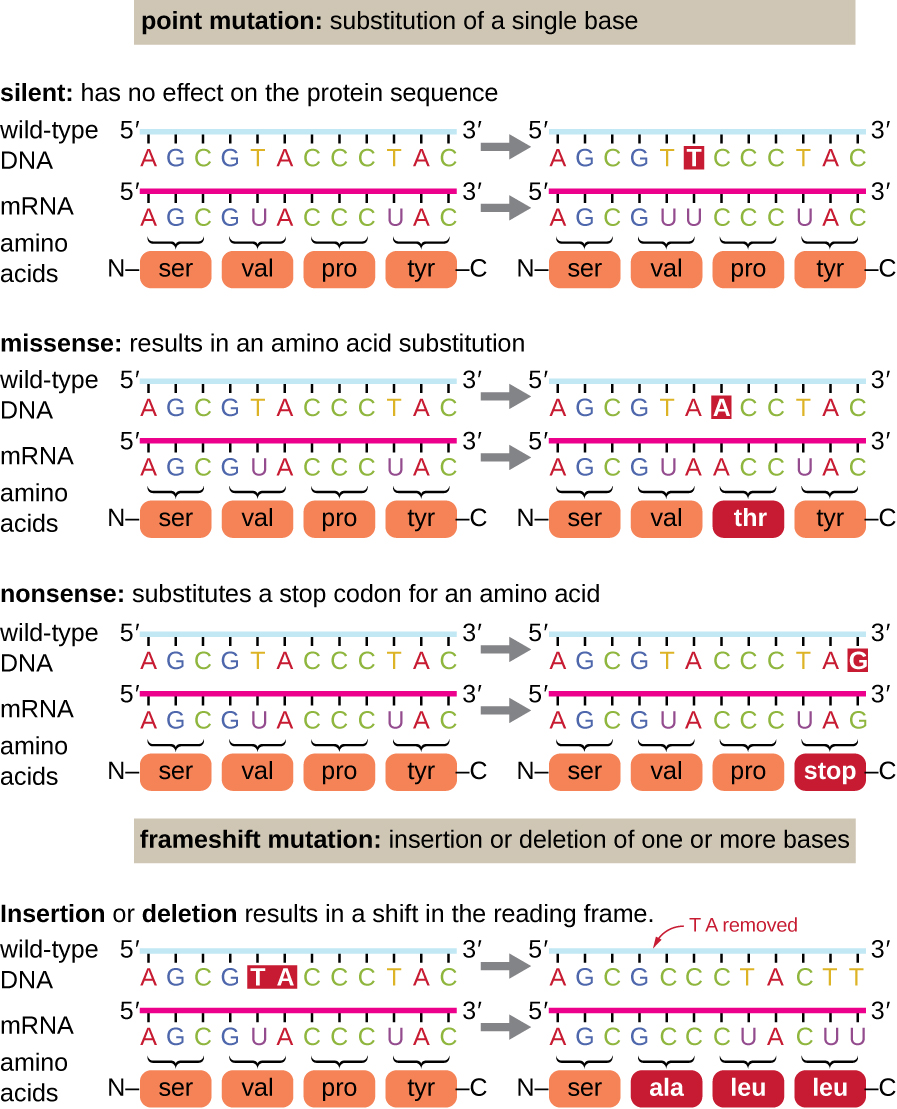
Existen varios tipos de mutaciones que se clasifican de acuerdo a cómo se altera la molécula de ADN. Un tipo, llamado mutación puntual, afecta a una sola base y más comúnmente ocurre cuando una base es sustituida o reemplazada por otra. Las mutaciones también resultan de la adición de una o más bases, conocidas como inserción, o la eliminación de una o más bases, conocidas como deleción.
Ejercicio\(\PageIndex{1}\)
¿Qué tipo de mutación ocurre cuando un gen tiene dos nucleótidos menos en su secuencia?
Efectos de las mutaciones en la estructura y función de las proteínas
Las mutaciones puntuales pueden tener una amplia gama de efectos sobre la función de las proteínas (Figura\(\PageIndex{1}\)). Como consecuencia de la degeneración del código genético, una mutación puntual generalmente dará como resultado que el mismo aminoácido se incorpore al polipéptido resultante a pesar del cambio de secuencia. Este cambio no tendría ningún efecto sobre la estructura de la proteína, y así se llama una mutación silenciosa. Una mutación sin sentido da como resultado que se incorpore un aminoácido diferente en el polipéptido resultante. El efecto de una mutación sin sentido depende de cuán químicamente diferente sea el nuevo aminoácido del aminoácido de tipo silvestre. La ubicación del aminoácido cambiado dentro de la proteína también es importante. Por ejemplo, si el aminoácido cambiado es parte del sitio activo de la enzima, entonces el efecto de la mutación sin sentido puede ser significativo. Muchas mutaciones de sentido erróneo dan como resultado proteínas que aún son funcionales, al menos hasta cierto punto. A veces, los efectos de las mutaciones de sentido erróneo solo pueden ser evidentes bajo ciertas condiciones ambientales; tales mutaciones de sentido erróneo se denominan mutaciones condicionales. En raras ocasiones, una mutación sin sentido puede ser beneficiosa. Bajo las condiciones ambientales adecuadas, este tipo de mutación puede dar al organismo que la alberga una ventaja selectiva. Otro tipo más de mutación puntual, llamada mutación sin sentido, convierte un codón que codifica un aminoácido (un codón sentido) en un codón de terminación (un codón sin sentido). Las mutaciones sin sentido dan como resultado la síntesis de proteínas que son más cortas que las de tipo silvestre y normalmente no funcionales.
Las deleciones e inserciones también causan diversos efectos. Debido a que los codones son tripletes de nucleótidos, las inserciones o deleciones en grupos de tres nucleótidos pueden conducir a la inserción o deleción de uno o más aminoácidos y pueden no causar efectos significativos sobre la funcionalidad de la proteína resultante. Sin embargo, las mutaciones de desplazamiento de marco, causadas por inserciones o deleciones de varios nucleótidos que no son un múltiplo de tres, son extremadamente problemáticas debido a que resulta un cambio en el marco de lectura (Figura\(\PageIndex{1}\)). Debido a que los ribosomas leen el ARNm en codones tripletes, las mutaciones de desplazamiento de marco pueden cambiar cada aminoácido después del punto de la mutación. El nuevo marco de lectura también puede incluir un codón de terminación antes del final de la secuencia codificante. En consecuencia, las proteínas elaboradas a partir de genes que contienen mutaciones de cambio de marco casi siempre son infuncionales.
Ejercicio\(\PageIndex{2}\)
- ¿Cuáles son las razones por las que un cambio de nucleótido en un gen de una proteína podría no tener ningún efecto sobre el fenotipo de ese gen?
- ¿Es posible que una inserción de tres nucleótidos juntos después del quinto nucleótido en un gen codificante de proteínas produzca una proteína que sea más corta de lo normal? ¿Cómo o cómo no?
Una mutación beneficiosa
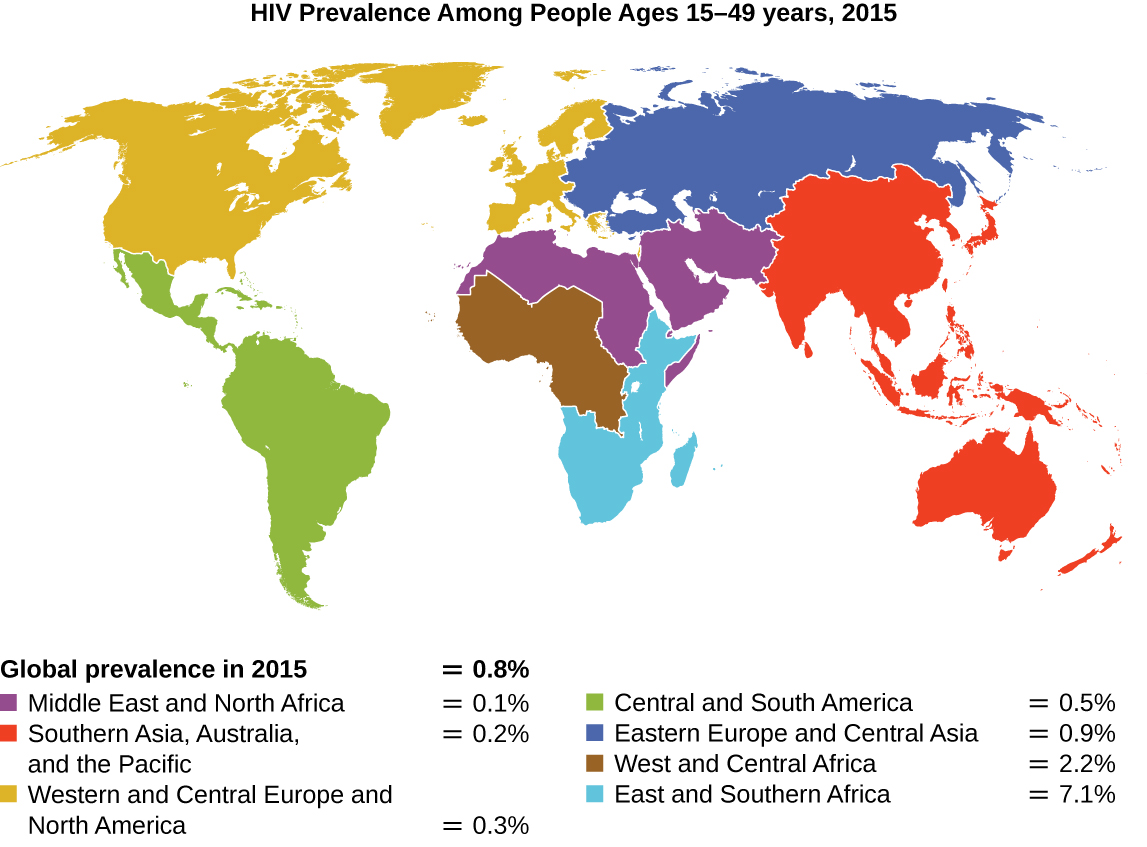
Desde que se reportó el primer caso de infección por el virus de la inmunodeficiencia humana (VIH) en 1981, casi 40 millones de personas han muerto por infección por VIH, 1 el virus que causa el síndrome de inmunodeficiencia adquirida (SIDA). El virus se dirige a las células T auxiliares que desempeñan un papel clave en la unión de la respuesta inmune innata y adaptativa, infectando y matando las células normalmente involucradas en la respuesta del cuerpo a la infección. No existe cura para la infección por VIH, pero se han desarrollado muchos medicamentos para ralentizar o bloquear la progresión del virus. Aunque los individuos de todo el mundo pueden estar infectados, la prevalencia más alta entre las personas de 15 a 49 años se encuentra en el África subsahariana, donde casi una persona de cada 20 está infectada, lo que representa más del 70% de las infecciones a nivel mundial 2 (Figura\(\PageIndex{2}\)). Desafortunadamente, esto también es una parte del mundo donde las estrategias de prevención y los medicamentos para tratar la infección son los que más faltan.
En los últimos años, el interés científico se ha despertado por el descubrimiento de unos pocos individuos del norte de Europa que son resistentes a la infección por VIH. En 1998, el genetista estadounidense Stephen J. O'Brien de los Institutos Nacionales de la Salud (NIH) y colegas publicaron los resultados de su análisis genético de más de 4,000 individuos. Estos indicaron que muchos individuos de ascendencia euroasiática (hasta 14% en algunos grupos étnicos) tienen una mutación por deleción, llamada CCR5-delta 32, en el gen que codifica CCR5. El CCR5 es un correceptor que se encuentra en la superficie de las células T que es necesario para que muchas cepas del virus entren en la célula hospedadora. La mutación conduce a la producción de un receptor al que el VIH no puede unirse de manera efectiva y, por lo tanto, bloquea la entrada viral. Las personas homocigóticas para esta mutación tienen una susceptibilidad muy reducida a la infección por VIH, y las que son heterocigóticas también tienen cierta protección contra la infección.
No está claro por qué las personas de ascendencia del norte de Europa, específicamente, portan esta mutación, pero su prevalencia parece ser más alta en el norte de Europa y disminuye constantemente en las poblaciones a medida que uno se mueve hacia el sur. Las investigaciones indican que la mutación ha estado presente desde antes de que apareciera el VIH y puede haber sido seleccionada en poblaciones europeas como resultado de la exposición a la peste o viruela. Esta mutación puede proteger a los individuos de la peste (causada por la bacteria Yersinia pestis) y la viruela (causada por el virus de la variola) porque este receptor también puede estar involucrado en estas enfermedades. La edad de esta mutación es un tema de debate, pero estimaciones sugieren que apareció entre 1875 años y hace 225 años, y puede haberse extendido desde el norte de Europa a través de invasiones vikingas.
Este emocionante hallazgo ha llevado a nuevas vías en la investigación del VIH, incluida la búsqueda de medicamentos para bloquear la unión de CCR5 al VIH en individuos que carecen de la mutación. Si bien es posible realizar pruebas de ADN para determinar qué individuos portan la mutación CCR5-delta 32, existen casos documentados de individuos homocigotos para la mutación que contraen VIH. Por esta razón, las pruebas de ADN para la mutación no son ampliamente recomendadas por los funcionarios de salud pública para no incentivar conductas de riesgo en quienes portan la mutación. Sin embargo, inhibir la unión del VIH a CCR5 sigue siendo una estrategia válida para el desarrollo de terapias farmacológicas para los infectados con el VIH.
Causas de las Mutaciones
Los errores en el proceso de replicación del ADN pueden provocar mutaciones espontáneas. La tasa de error de la ADN polimerasa es de una base incorrecta por mil millones de pares de bases replicados. La exposición a mutágenos puede provocar mutaciones inducidas, que son diversos tipos de agentes químicos o radiación (Tabla\(\PageIndex{1}\)). La exposición a un mutágeno puede aumentar la tasa de mutación más de 1000 veces. Los mutágenos suelen ser también carcinógenos, agentes que causan cáncer. Sin embargo, mientras que casi todos los carcinógenos son mutagénicos, no todos los mutágenos son necesariamente carcinógenos.
Mutágenos Químicos
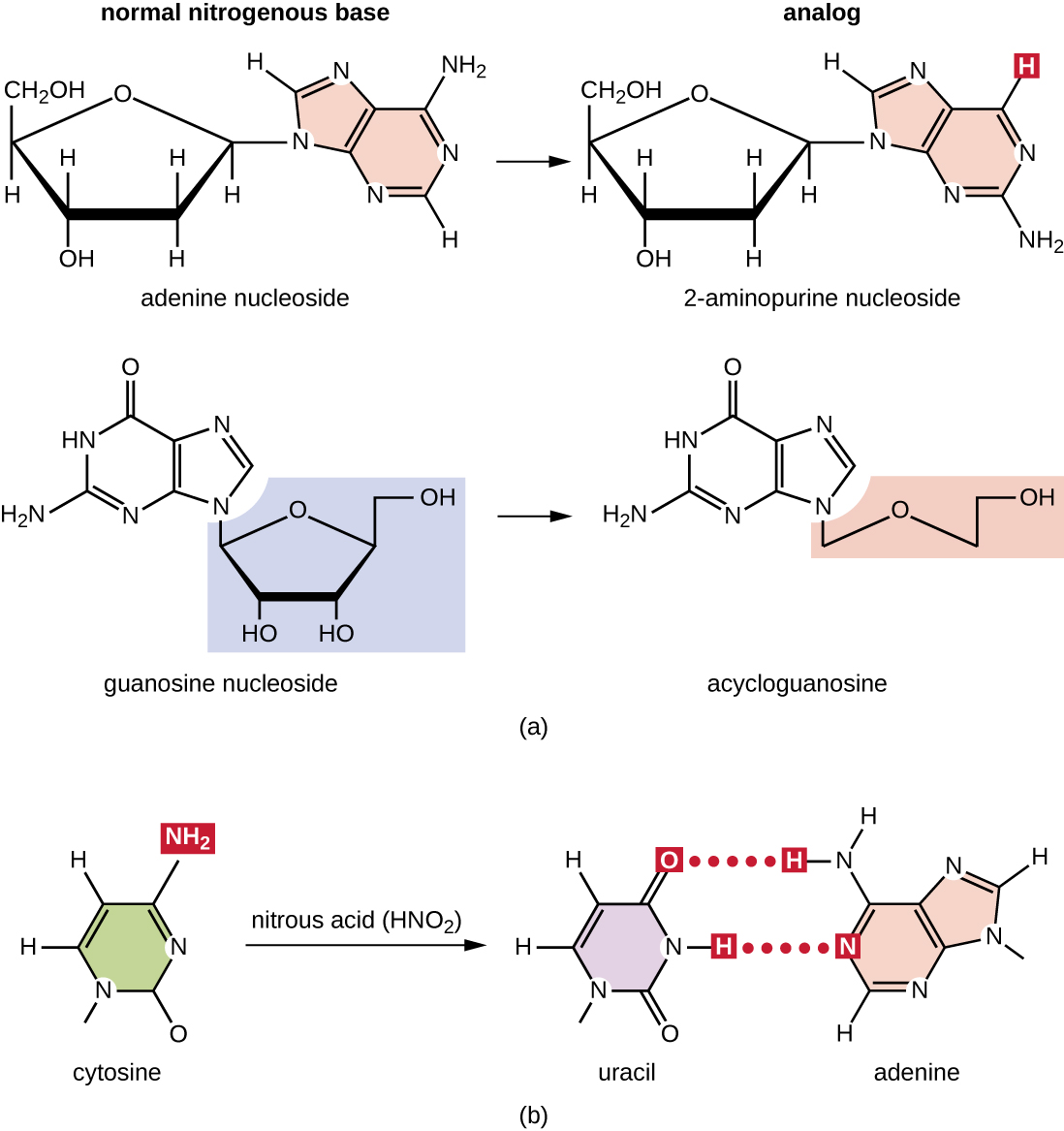
Diversos tipos de mutágenos químicos interactúan directamente con el ADN ya sea actuando como análogos de nucleósidos o modificando bases nucleotídicas. Los químicos llamados análogos de nucleósidos son estructuralmente similares a las bases nucleotídicas normales y pueden incorporarse al ADN durante la replicación (Figura\(\PageIndex{3}\)). Estos análogos de bases inducen mutaciones porque a menudo tienen reglas de apareamiento de bases diferentes a las bases que reemplazan. Otros mutágenos químicos pueden modificar bases normales de ADN, dando como resultado diferentes reglas de emparejamiento de bases. Por ejemplo, el ácido nitroso desamina la citosina, convirtiéndola en uracilo. El uracilo luego se empareja con adenina en una ronda posterior de replicación, lo que resulta en la conversión de un par de bases GC en un par de bases AT. El ácido nitroso también desamina la adenina en hipoxantina, que se empareja con citosina en lugar de timina, resultando en la conversión de un par de bases TA en un par de bases CG.
Los mutágenos químicos conocidos como agentes intercalantes funcionan de manera diferente. Estas moléculas se deslizan entre las bases nitrogenadas apiladas de la doble hélice del ADN, distorsionando la molécula y creando un espaciamiento atípico entre los pares de bases nucleotídicas (Figura\(\PageIndex{4}\)). Como resultado, durante la replicación del ADN, la ADN polimerasa puede omitir la replicación de varios nucleótidos (creando una deleción) o insertar nucleótidos adicionales (creando una inserción). Cualquiera de los resultados puede conducir a una mutación de cambio de marco. Los productos de combustión como los hidrocarburos aromáticos policíclicos son agentes intercalantes particularmente peligrosos que pueden conducir a cánceres causados por mutaciones. Los agentes intercalantes bromuro de etidio y naranja de acridina se utilizan comúnmente en el laboratorio para teñir el ADN para su visualización y son mutágenos potenciales.
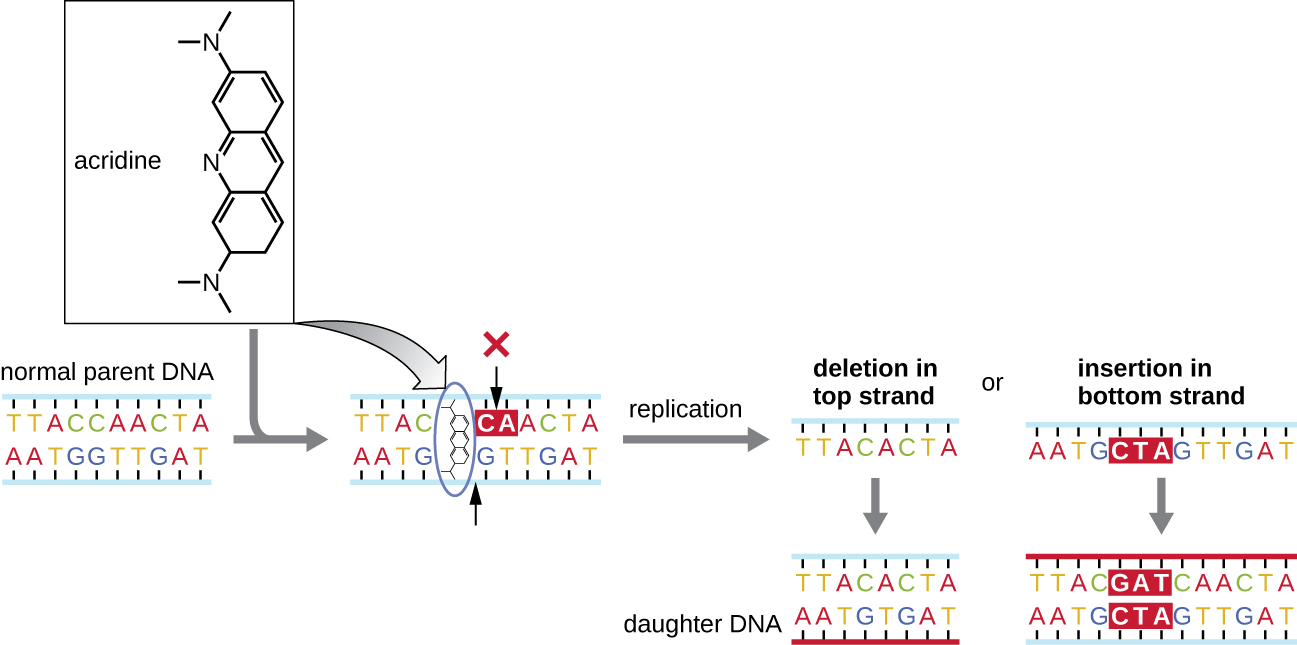
Radiación
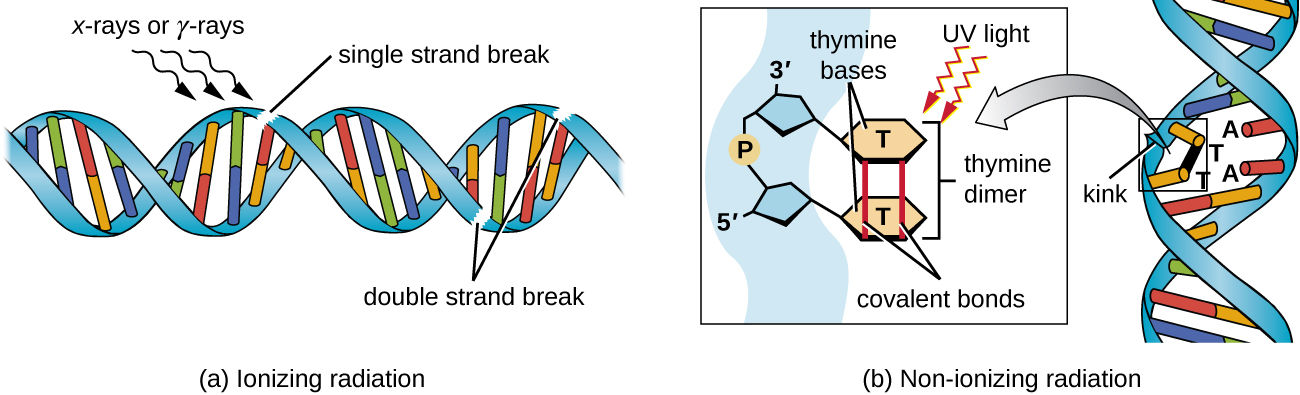
La exposición a radiación ionizante o no ionizante puede inducir mutaciones en el ADN, aunque por diferentes mecanismos. La fuerte radiación ionizante como los rayos X y los rayos gamma pueden causar roturas monocatenarias y bicatenarias en la cadena principal del ADN a través de la formación de radicales hidroxilo en la exposición a la radiación (Figura\(\PageIndex{5}\)). La radiación ionizante también puede modificar bases; por ejemplo, la desaminación de citosina a uracilo, análoga a la acción del ácido nitroso. 3 La exposición a la radiación ionizante se usa para matar microbios para esterilizar dispositivos médicos y alimentos, debido a su dramático efecto inespecífico en dañar el ADN, las proteínas y otros componentes celulares (ver Uso de métodos físicos para controlar microorganismos).
La radiación no ionizante, como la luz ultravioleta, no es lo suficientemente energética como para iniciar este tipo de cambios químicos. Sin embargo, la radiación no ionizante puede inducir la formación de dímeros entre dos bases de pirimidina adyacentes, comúnmente dos timinas, dentro de una cadena de nucleótidos. Durante la formación del dímero de timina, las dos timinas adyacentes se unen covalentemente y, si se dejan sin reparar, tanto la replicación como la transcripción del ADN se estancan en este punto. La ADN polimerasa puede proceder y replicar el dímero incorrectamente, lo que podría conducir a cambios de marco o mutaciones puntuales.
| Agentes Mutágenos | Modo de Acción | Efecto sobre el ADN | Tipo de mutación resultante |
|---|---|---|---|
| Análogos de nucleósidos | |||
| 2-aminopurina | Se inserta en lugar de A pero pares de bases con C | Convierte el par base AT a GC | Punto |
| 5-bromouracilo | Se inserta en lugar de T pero pares de bases con G | Convierte el par base AT a GC | Punto |
| Agente modificador de nucleótidos | |||
| Óxido nitroso | Desaminados C a U | Convierte el par base GC a AT | Punto |
| Agentes intercalantes | |||
| Naranja de acridina, bromuro de etidio, hidrocarburos aromáticos policíclicos | Distorsiona la doble hélice, crea un espaciado inusual entre nucleótidos | Introduce pequeñas eliminaciones e inserciones | Cambio de marco |
| Radiación ionizante | |||
| Rayos X, rayos-γ | Forma radicales hidroxilo | Causa roturas de ADN de cadena simple y doble | Los mecanismos de reparación pueden introducir mutaciones |
| Rayos X, rayos-γ | Modifica las bases (por ejemplo, desaminación de C a U) | Convierte el par base GC a AT | Punto |
| Radiación no ionizante | |||
| Ultraultravioleta | Forma dímeros de pirimidina (generalmente timina) | Causa errores de replicación de ADN | Cambio de marco o punto |
Ejercicio\(\PageIndex{3}\)
- ¿Cómo introduce una mutación un análogo de base?
- ¿Cómo introduce una mutación un agente intercalante?
- ¿Qué tipo de mutágeno causa los dímeros de timina?
Reparación de ADN
El proceso de replicación del ADN es altamente preciso, pero los errores pueden ocurrir espontáneamente o ser inducidos por mutágenos. Los errores no corregidos pueden llevar a graves consecuencias para el fenotipo. Las células han desarrollado varios mecanismos de reparación para minimizar el número de mutaciones que persisten.
Corrección
La mayoría de los errores introducidos durante la replicación del ADN son corregidos rápidamente por la mayoría de las ADN polimerasas a través de una función llamada corrección. En la revisión, la ADN polimerasa lee la base recién agregada, asegurando que es complementaria a la base correspondiente en la cadena molde antes de agregar la siguiente. Si se ha agregado una base incorrecta, la enzima realiza un corte para liberar el nucleótido equivocado y se agrega una nueva base.
Reparación de desajuste
Algunos errores introducidos durante la replicación se corrigen poco después de que la maquinaria de replicación se haya movido. Este mecanismo se llama reparación de desajustes. Las enzimas involucradas en este mecanismo reconocen el nucleótido incorrectamente agregado, lo extirpan y lo reemplazan con la base correcta. Un ejemplo es la reparación de desapareamientos dirigidos por metilo en E. coli. El ADN está hemimetilado. Esto significa que la hebra parental está metilada mientras que la cadena hija recién sintetizada no lo está. Se necesitan varios minutos antes de que la nueva hebra sea metilada. Las proteínas MuTs, MuTl y MuTH se unen al sitio hemimetilado donde se encuentra el nucleótido incorrecto. MuTH corta la hebra no metilada (la nueva hebra). Una exonucleasa elimina una porción de la cadena (incluyendo el nucleótido incorrecto). El hueco formado se rellena con ADN pol III y ligasa.
Reparación de Dímeros de Timina
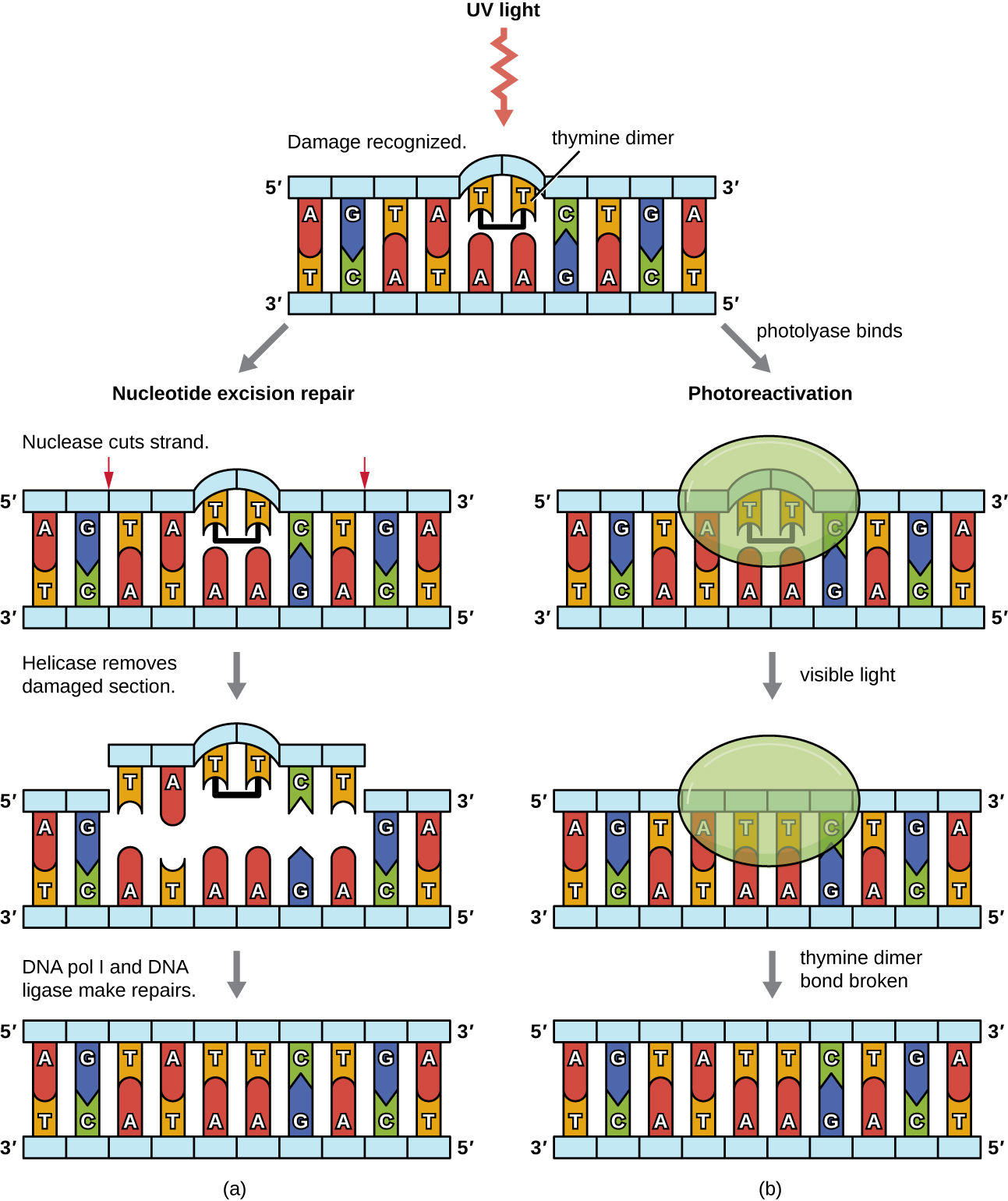
Debido a que la producción de dímeros de timina es común (muchos organismos no pueden evitar la luz ultravioleta), han evolucionado mecanismos para reparar estas lesiones. En la reparación por escisión de nucleótidos (también llamada reparación oscura), las enzimas eliminan el dímero de pirimidina y lo reemplazan con los nucleótidos correctos (Figura\(\PageIndex{6}\)). En E. coli, el ADN es escaneado por un complejo enzimático. Si se encuentra una distorsión en la doble hélice que fue introducida por el dímero de pirimidina, el complejo enzimático corta la cadena principal de azúcar-fosfato varias bases aguas arriba y aguas abajo del dímero, y el segmento de ADN entre estos dos cortes se elimina enzimáticamente. El ADN pol I reemplaza los nucleótidos faltantes por los correctos y la ADN ligasa sella la brecha en la cadena principal de azúcar-fosfato.
La reparación directa (también llamada reparación de luz) de los dímeros de timina se produce a través del proceso de fotorreactivación en presencia de luz visible. Una enzima llamada fotóliasa reconoce la distorsión en la hélice del ADN causada por el dímero de timina y se une al dímero. Luego, en presencia de luz visible, la enzima fotoliasa cambia de conformación y rompe el dímero de timina, permitiendo que las timinas vuelvan a emparejarse correctamente con las adeninas en la cadena complementaria. La fotorreactivación parece estar presente en todos los organismos, con la excepción de los mamíferos placentarios, incluidos los humanos. La fotorreactivación es particularmente importante para los organismos expuestos crónicamente a la radiación ultravioleta, como plantas, bacterias fotosintéticas, algas y corales, para evitar la acumulación de mutaciones causadas por la formación de dímeros de timina.
Ejercicio\(\PageIndex{4}\)
- Durante la reparación de desajustes, ¿cómo reconoce la enzima cuál es la nueva y cuál es la hebra vieja?
- ¿Qué tipo de mutación repara la fotoliasa?
Identificación de mutantes bacterianos
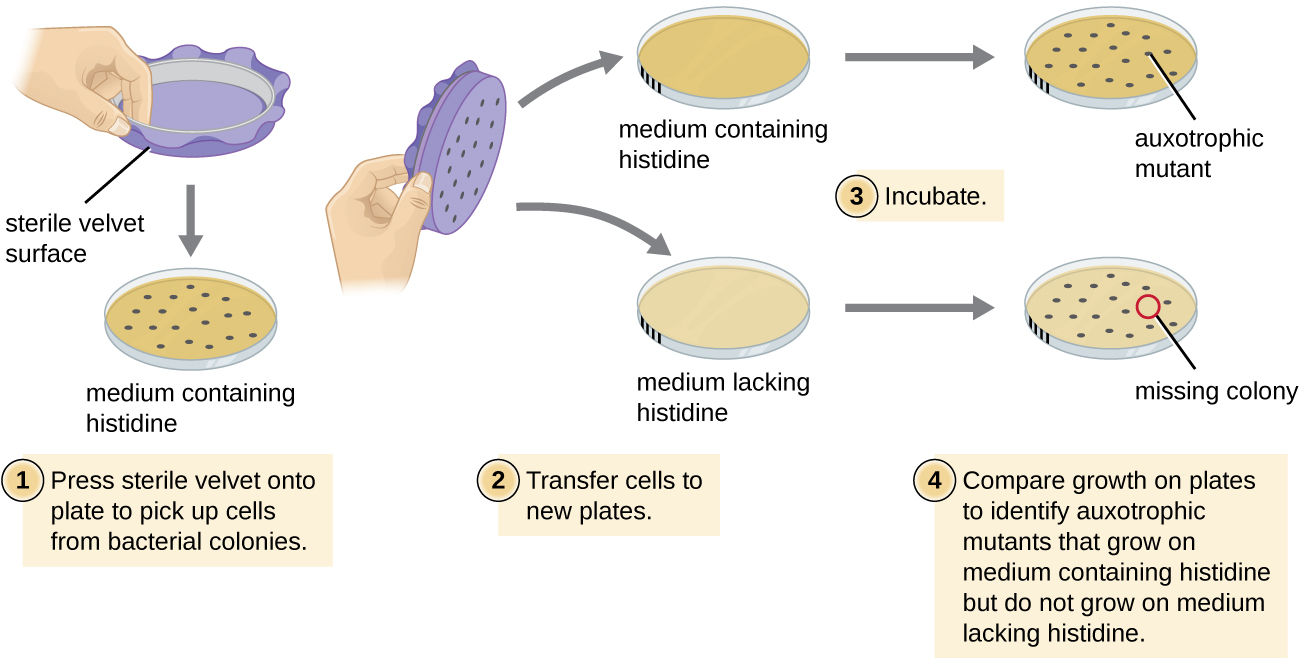
Una técnica común utilizada para identificar mutantes bacterianos se llama réplica de placas. Esta técnica se utiliza para detectar mutantes nutricionales, llamados auxótrofos, que tienen una mutación en un gen que codifica una enzima en la vía de biosíntesis de un nutriente específico, como un aminoácido. Como resultado, mientras que las células de tipo silvestre conservan la capacidad de crecer normalmente en un medio que carece del nutriente específico, los auxótrofos son incapaces de crecer en dicho medio. Durante la siembra de réplica (Figura\(\PageIndex{7}\)), una población de células bacterianas se mutageniza y luego se plaquean como células individuales en una placa compleja nutricionalmente completa y se deja crecer en colonias. Las células de estas colonias se retiran de esta placa maestra, a menudo usando terciopelo estéril. Este terciopelo, que contiene celdas, se presiona luego en la misma orientación sobre placas de diversos medios. Al menos una placa también debe estar nutricionalmente completa para asegurar que las células se transfieren adecuadamente entre las placas. Las otras placas carecen de nutrientes específicos, lo que permite al investigador descubrir diversos mutantes auxótrofos incapaces de producir nutrientes específicos. Las células de la colonia correspondiente en la placa nutricionalmente completa pueden ser utilizadas para recuperar el mutante para su posterior estudio.
Ejercicio\(\PageIndex{5}\)
¿Por qué las células se siembran en una placa nutricionalmente completa además de las placas deficientes en nutrientes cuando se busca un mutante?
La prueba de Ames
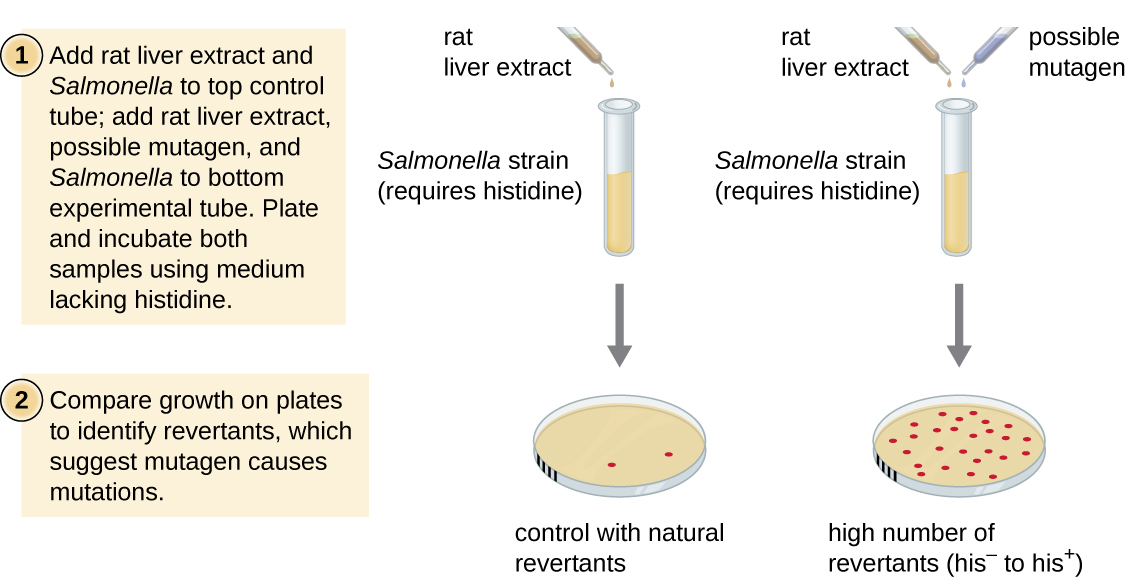
La prueba de Ames, desarrollada por Bruce Ames (1928—) en la década de 1970, es un método que utiliza bacterias para el cribado rápido y económico del potencial cancerígeno de nuevos compuestos químicos. La prueba mide la tasa de mutación asociada a la exposición al compuesto, la cual, si es elevada, puede indicar que la exposición a este compuesto está asociada con un mayor riesgo de cáncer. La prueba de Ames utiliza como organismo de prueba una cepa de Salmonella typhimurium que es un auxótrofo de histidina, incapaz de sintetizar su propia histidina debido a una mutación en un gen esencial requerido para su síntesis. Después de la exposición a un mutágeno potencial, estas bacterias se colocan en placas sobre un medio que carece de histidina, y se registra el número de mutantes que recuperan la capacidad de sintetizar histidina y se compara con el número de dichos mutantes que surgen en ausencia del mutágeno potencial (Figura\(\PageIndex{8}\)). Los químicos que son más mutagénicos traerán consigo más mutantes con síntesis de histidina restaurada en la prueba de Ames. Debido a que muchos productos químicos no son directamente mutagénicos sino que son metabolizados a formas mutagénicas por las enzimas hepáticas, el extracto de hígado de rata se incluye comúnmente al inicio de este experimento para imitar el metabolismo hepático. Después de realizar la prueba de Ames, los compuestos identificados como mutagénicos se prueban adicionalmente para determinar sus posibles propiedades cancerígenas mediante el uso de otros modelos, incluyendo modelos animales como ratones y ratas.
Ejercicio\(\PageIndex{6}\)
- ¿Qué mutación se utiliza como indicador de la tasa de mutación en la prueba de Ames?
- ¿Por qué la prueba de Ames puede funcionar como prueba de carcinogenicidad?
Conceptos clave y resumen
- Una mutación es un cambio heredable en el ADN. Una mutación puede conducir a un cambio en la secuencia de aminoácidos de una proteína, posiblemente afectando su función.
- Una mutación puntual afecta a un solo par de bases. Una mutación puntual puede causar una mutación silenciosa si el codón de ARNm codifica para el mismo aminoácido, una mutación sin sentido si el codón de ARNm codifica para un aminoácido diferente, o una mutación sin sentido si el codón de ARNm se convierte en un codón de terminación.
- Las mutaciones sin sentido pueden retener la función, dependiendo de la química del nuevo aminoácido y su ubicación en la proteína. Las mutaciones sin sentido producen proteínas truncadas y frecuentemente no funcionales.
- Una mutación de desplazamiento de marco resulta de una inserción o deleción de varios nucleótidos que no es un múltiplo de tres. El cambio en el marco de lectura altera cada aminoácido después del punto de la mutación y da como resultado una proteína no funcional.
- Las mutaciones espontáneas ocurren a través de errores de replicación del ADN, mientras que las mutaciones inducidas ocurren a través de la exposición a
- Los agentes mutagénicos son frecuentemente cancerígenos pero no siempre. Sin embargo, casi todos los carcinógenos son mutagénicos.
- Los mutágenos químicos incluyen análogos de bases y químicos que modifican las bases existentes. En ambos casos, las mutaciones se introducen después de varias rondas de replicación del ADN.
- La radiación ionizante, como los rayos X y los rayos γ, conduce a la rotura del esqueleto fosfodiéster del ADN y también puede modificar químicamente las bases para alterar sus reglas de emparejamiento de bases.
- La radiación no ionizante como la luz ultravioleta puede introducir dímeros de pirimidina (timina) que, durante la replicación y transcripción del ADN, pueden introducir cambios de marco o mutaciones puntuales.
- Las células tienen mecanismos para reparar mutaciones naturales. La ADN polimerasa tiene actividad correctora. La reparación de desapareamientos es un proceso para reparar bases incorporadas incorrectamente después de completar la replicación del ADN.
- También se pueden reparar dímeros de pirimidina. En la reparación por escisión de nucleótidos (reparación oscura), las enzimas reconocen la distorsión introducida por el dímero de pirimidina y reemplazan la cadena dañada con las bases correctas, utilizando como molde la cadena de ADN no dañada. Las bacterias y otros organismos también pueden utilizar la reparación directa, en la que la enzima fotoliasa, en presencia de luz visible, rompe las pirimidinas.
- A través de la comparación del crecimiento en la placa completa y la falta de crecimiento en medios que carecen de nutrientes específicos, se pueden identificar mutantes específicos de pérdida de función llamados auxótrofos.
- La prueba de Ames es un método económico que utiliza bacterias auxotróficas para medir la mutagenicidad de un compuesto químico. La mutagenicidad es un indicador de potencial cancerígeno.
Notas al pie
- 1 Organización Mundial de la Salud. “Datos del Observatorio Mundial de la Salud (GHO), VIH/SIDA”. http://www.who.int/gho/hiv/en/. Consultado el 5 de agosto de 2016.
- 2 Organización Mundial de la Salud. “Datos del Observatorio Mundial de la Salud (GHO), VIH/SIDA”. http://www.who.int/gho/hiv/en/. Consultado el 5 de agosto de 2016.
- 3 K.R. Tindall et al. “Cambios en la Secuencia de Bases de ADN Inducidos por Mutagénesis de Rayos Gamma de Fago Lambda y Prof Genética 118 núm. 4 (1988) :551—560.


