12.14: Más ejemplos de RMN
- Page ID
- 76640
Ejemplos adicionales de RMN
Para cada molécula, predecir el número de señales en los espectros 1H-NMR y 13 C-NMR (no contar picos divididos - por ejemplo, un cuarteto cuenta como una sola señal). Supongamos que los grupos diastereotópicos no son equivalentes.
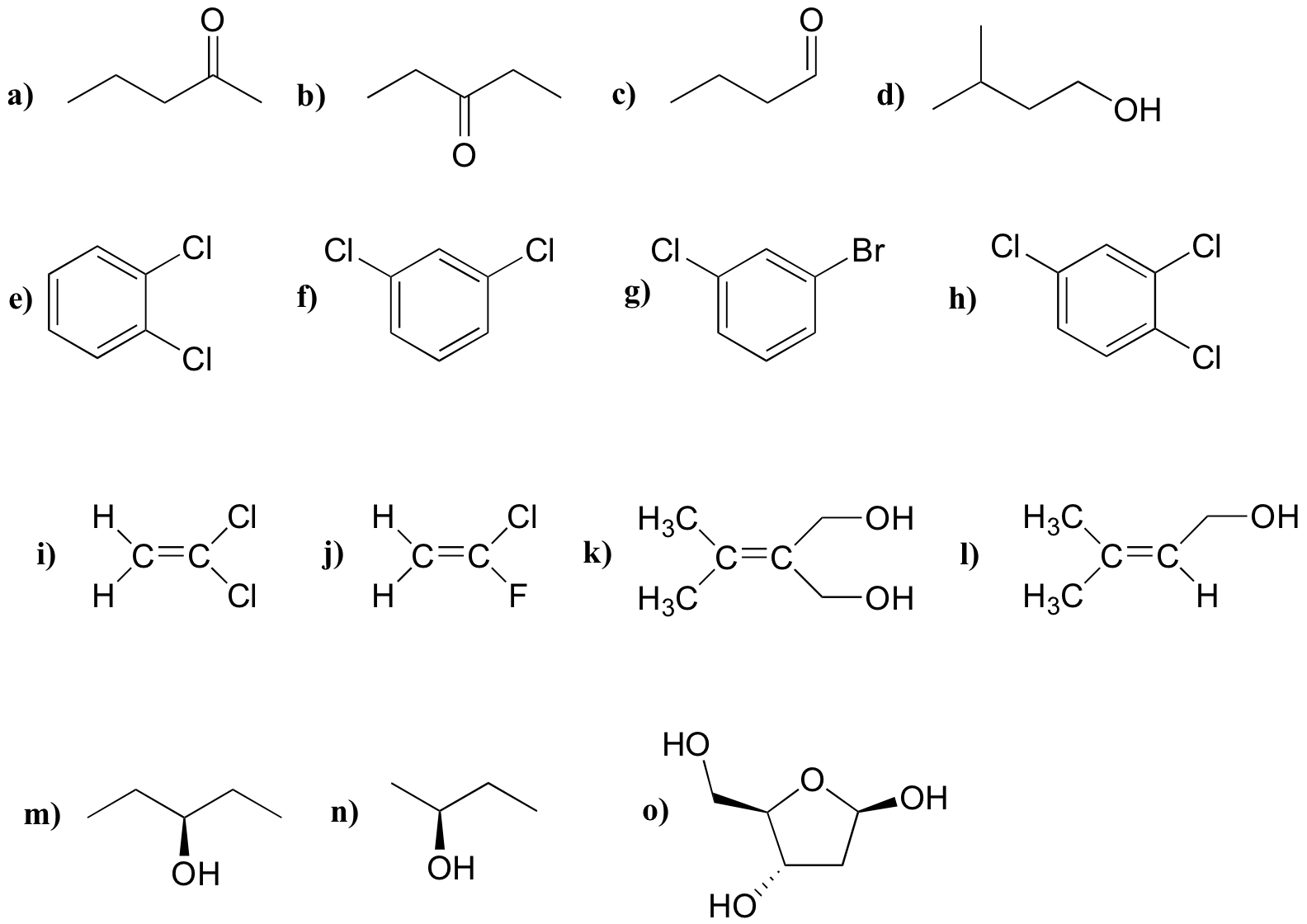
P5.2: Para cada uno de los 20 aminoácidos comunes, predecir el número de señales en el espectro de 13 C-NMR desacoplados de protones.
P5.3: Calcular el valor de desplazamiento químico (expresado en Hz, a un decimal) de cada subpico en la señal de doblete de 1H-RMN a continuación. Haz esto para:
a) un espectro obtenido en un instrumento de 300 MHz
b) un espectro obtenido en un instrumento de 100 MHz
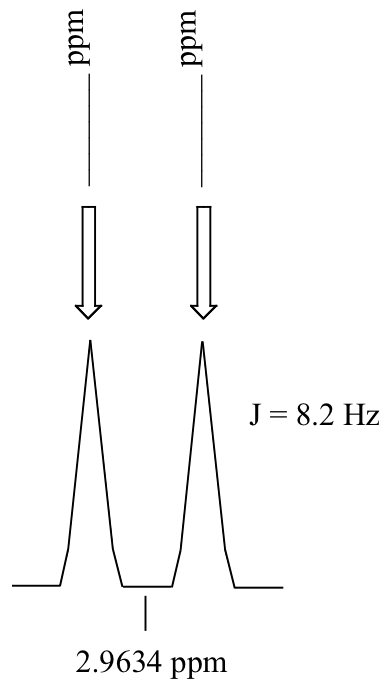
P5.4: Considerar una señal de cuarteto en un espectro de 1H-RMN obtenida en un instrumento de 300 MHz. El desplazamiento químico se registra como 1.7562 ppm, y la constante de acoplamiento es J = 7.6 Hz. ¿Cuál es el desplazamiento químico, expresado al 0.1 Hz más cercano, del subpico más lejano en el cuarteto? ¿Cuál es la frecuencia de resonancia (nuevamente expresada en Hz) de este subpico?)
P5.5: Un patrón de división fácilmente reconocible para las señales de protones aromáticos de estructuras de benceno disustituido es un par de dobletes. ¿Este patrón indica sustitución orto, meta o para?
P5.6: Coinciden los espectros de abajo con sus correspondientes estructuras A-F.
Estructuras:
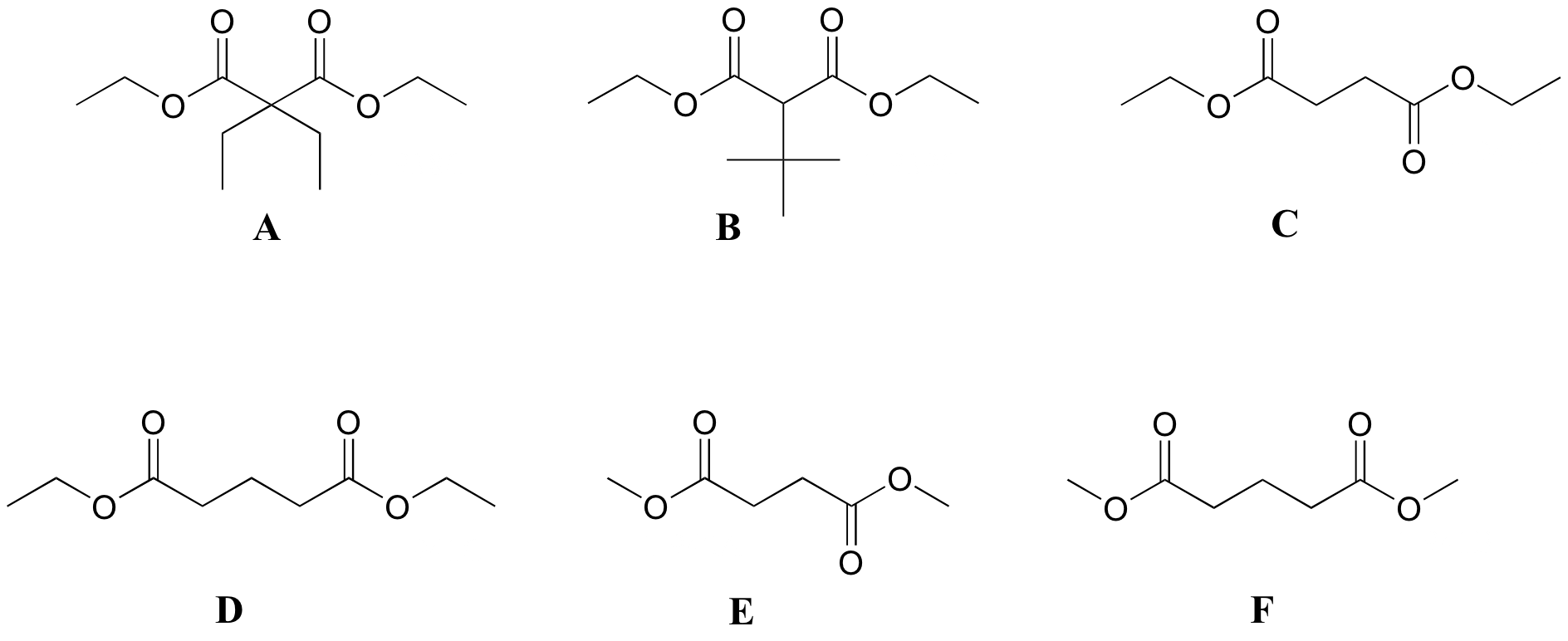
Espectro 1
|
δ |
dividiendo |
integración |
|
4.13 |
q |
2 |
|
2.45 |
t |
2 |
|
1.94 |
quinteto |
1 |
|
1.27 |
t |
3 |
Espectro 2
|
δ |
dividiendo |
integración |
|
3.68 |
s |
3 |
|
2.99 |
t |
2 |
|
1.95 |
quinteto |
1 |
Espectro 3
|
δ |
dividiendo |
integración |
|
4.14 |
q |
1 |
|
2.62 |
s |
1 |
|
1.26 |
t |
1.5 |
Espectro 4
|
δ |
dividiendo |
integración |
|
4.14 |
q |
4 |
|
3.22 |
s |
1 |
|
1.27 |
t |
6 |
|
1.13 |
s |
9 |
Espectro 5
|
δ |
dividiendo |
integración |
|
4.18 |
q |
1 |
|
1.92 |
q |
1 |
|
1.23 |
t |
1.5 |
|
0.81 |
t |
1.5 |
Espectro 6
|
δ |
dividiendo |
integración |
|
3.69 |
s |
1.5 |
|
2.63 |
s |
1 |
P5.7: Coinciden los espectros 7-12 abajo con sus correspondientes estructuras G-L.
Estructuras:

Espectro 7:
| δ |
dividiendo |
integración |
|
9.96 |
d |
1 |
|
5.88 |
d |
1 |
|
2.17 |
s |
3 |
|
1.98 |
s |
3 |
Espectro 8:
|
δ |
dividiendo |
integración |
|
9.36 |
s |
1 |
|
6.55 |
q |
1 |
|
2.26 |
q |
2 |
|
1.99 |
d |
3 |
|
0.96 |
t |
3 |
Espectro 9:
|
δ |
dividiendo |
integración |
|
9.57 |
s |
1 |
|
6.30 |
s |
1 |
|
6.00 |
s |
1 |
|
1.84 |
s |
3 |
Espectro 10:
|
δ |
dividiendo |
integración |
|
9.83 |
t |
1 |
|
2.27 |
d |
2 |
|
1.07 |
s |
9 |
Espectro 11:
|
δ |
dividiendo |
integración |
|
9.75 |
t |
1 |
|
2.30 |
dd |
2 |
|
2.21 |
m |
1 |
|
0.98 |
d |
6 |
Espectro 12:
|
δ |
dividiendo |
integración |
|
8.08 |
s |
1 |
|
4.13 |
t |
2 |
|
1.70 |
m |
2 |
|
0.96 |
t |
3 |
P5.8: Hacer coincidir los espectros de 1H-RMN 13-18 a continuación con sus correspondientes estructuras M-R.
Estructuras:
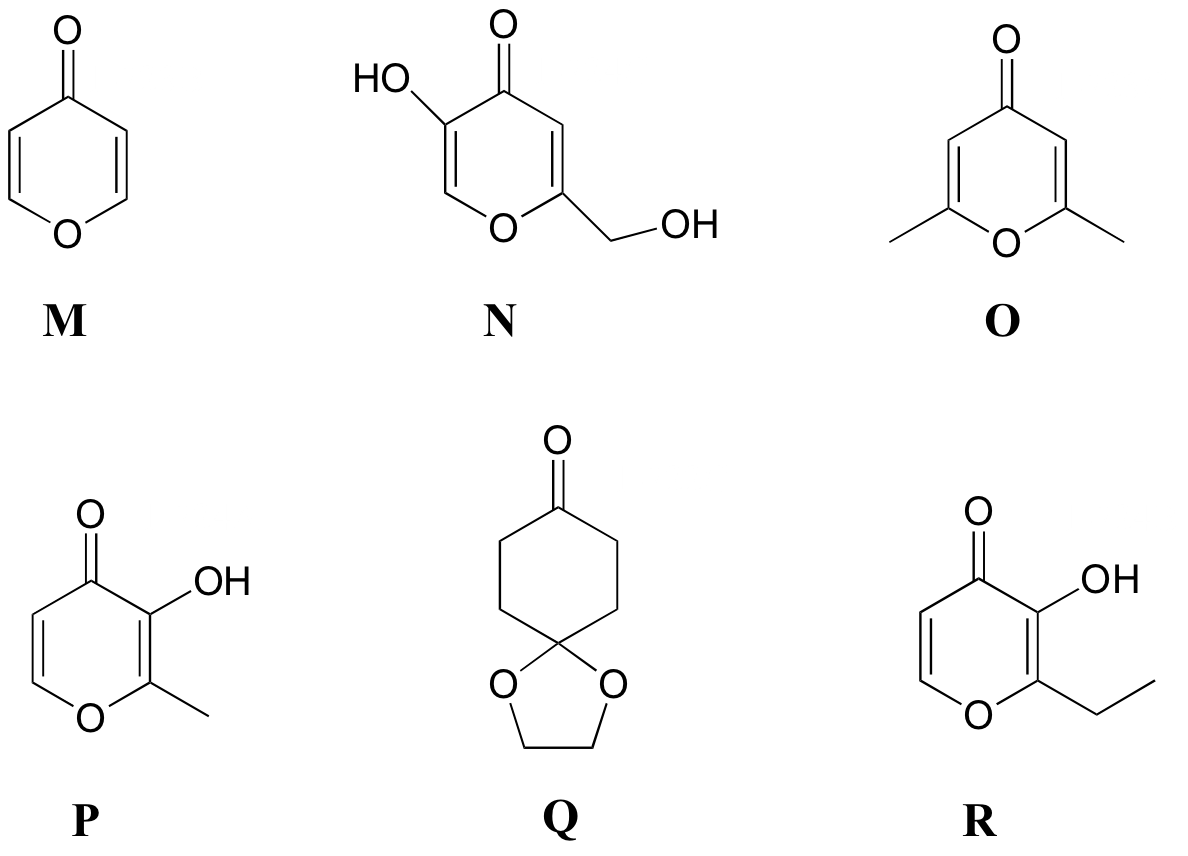
Espectro 13:
|
δ |
dividiendo |
integración |
|
8.15 |
d |
1 |
|
6.33 |
d |
1 |
Espectro 14: 1-723C (estructura O)
|
δ |
dividiendo |
integración |
|
6.05 |
s |
1 |
|
2.24 |
s |
3 |
Espectro 15:
|
δ |
dividiendo |
integración |
|
8.57 |
s (b) |
1 |
|
7.89 |
d |
1 |
|
6.30 |
d |
1 |
|
2.28 |
s |
3 |
Espectro 16:
|
δ |
dividiendo |
integración |
|
9.05 |
s (b) |
1 |
|
8.03 |
s |
1 |
|
6.34 |
s |
1 |
|
5.68 |
s (b) |
1 |
|
4.31 |
s |
2 |
Espectro 17:
|
δ |
dividiendo |
integración |
|
7.76 |
d |
1 |
|
7.57 |
s (b) |
1 |
|
6.44 |
d |
1 |
|
2.78 |
q |
2 |
|
1.25 |
t |
3 |
Espectro 18:
|
δ |
dividiendo |
integración |
|
4.03 |
s |
1 |
|
2.51 |
t |
1 |
|
2.02 |
t |
1 |
P5.9: Hacer coincidir los espectros de 1H-RMN 19-24 a continuación con sus correspondientes estructuras S-X.
Estructuras:

Espectro 19:
|
δ |
dividiendo |
integración |
|
9.94 |
s |
1 |
|
7.77 |
d |
2 |
|
7.31 |
d |
2 |
|
2.43 |
s |
3 |
Espectro 20:
|
δ |
dividiendo |
integración |
|
10.14 |
s |
2 |
|
8.38 |
s |
1 |
|
8.17 |
d |
2 |
|
7.75 |
t |
1 |
Espectro 21:
|
δ |
dividiendo |
integración |
|
9.98 |
s |
1 |
|
7.81 |
d |
2 |
|
7.50 |
d |
2 |
Espectro 22:
|
δ |
dividiendo |
integración |
|
7.15-7.29 |
m |
2.5 |
|
2.86 |
t |
1 |
|
2.73 |
t |
1 |
|
2.12 |
s |
1.5 |
Espectro 23:
|
δ |
dividiendo |
integración |
|
7.10 |
d |
1 |
|
6.86 |
d |
1 |
|
3.78 |
s |
1.5 |
|
3.61 |
s |
1 |
|
2.12 |
s |
1.5 |
Espectro 24:
|
δ |
dividiendo |
integración |
|
7.23-7.30 |
m |
1 |
|
3.53 |
s |
1 |
P5.10: Hacer coincidir los espectros de 1H-RMN 25-30 a continuación con sus estructuras correspondientes AA-FF.
Estructuras:
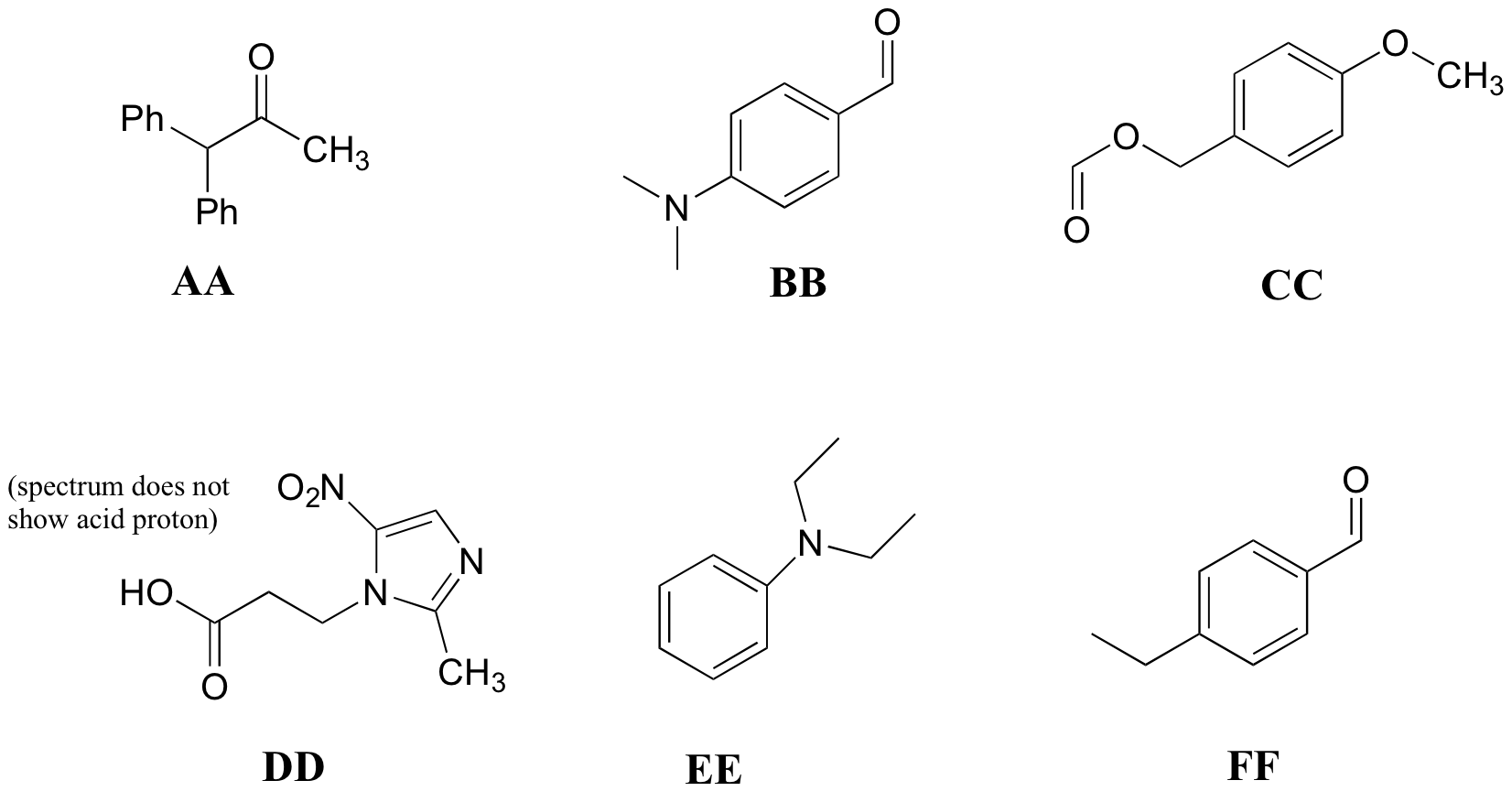
Espectro 25:
|
δ |
dividiendo |
integración |
|
9.96 |
s |
1 |
|
7.79 |
d |
2 |
|
7.33 |
d |
2 |
|
2.72 |
q |
2 |
|
1.24 |
t |
3 |
Espectro 26:
|
δ |
dividiendo |
integración |
|
9.73 |
s |
1 |
|
7.71 |
d |
2 |
|
6.68 |
d |
2 |
|
3.06 |
s |
6 |
Espectro 27:
|
δ |
dividiendo |
integración |
|
7.20-7.35 |
m |
10 |
|
5.12 |
s |
1 |
|
2.22 |
s |
3 |
Espectro 28:
|
δ |
dividiendo |
integración |
|
8.08 |
s |
1 |
|
7.29 |
d |
2 |
|
6.87 |
d |
2 |
|
5.11 |
s |
2 |
|
3.78 |
s |
3 |
Espectro 29:
|
δ |
dividiendo |
integración |
|
7.18 |
d |
1 |
|
6.65 |
m |
1.5 |
|
3.2 |
q |
2 |
|
1.13 |
t |
3 |
Espectro 30:
|
δ |
dividiendo |
integración |
|
8.32 |
s |
1 |
|
4.19 |
t |
2 |
|
2.83 |
t |
2 |
|
2.40 |
s |
3 |
P5.11: Hacer coincidir los espectros de 1H-RMN 31-36 a continuación con sus estructuras correspondientes GG-LL
Estructuras:
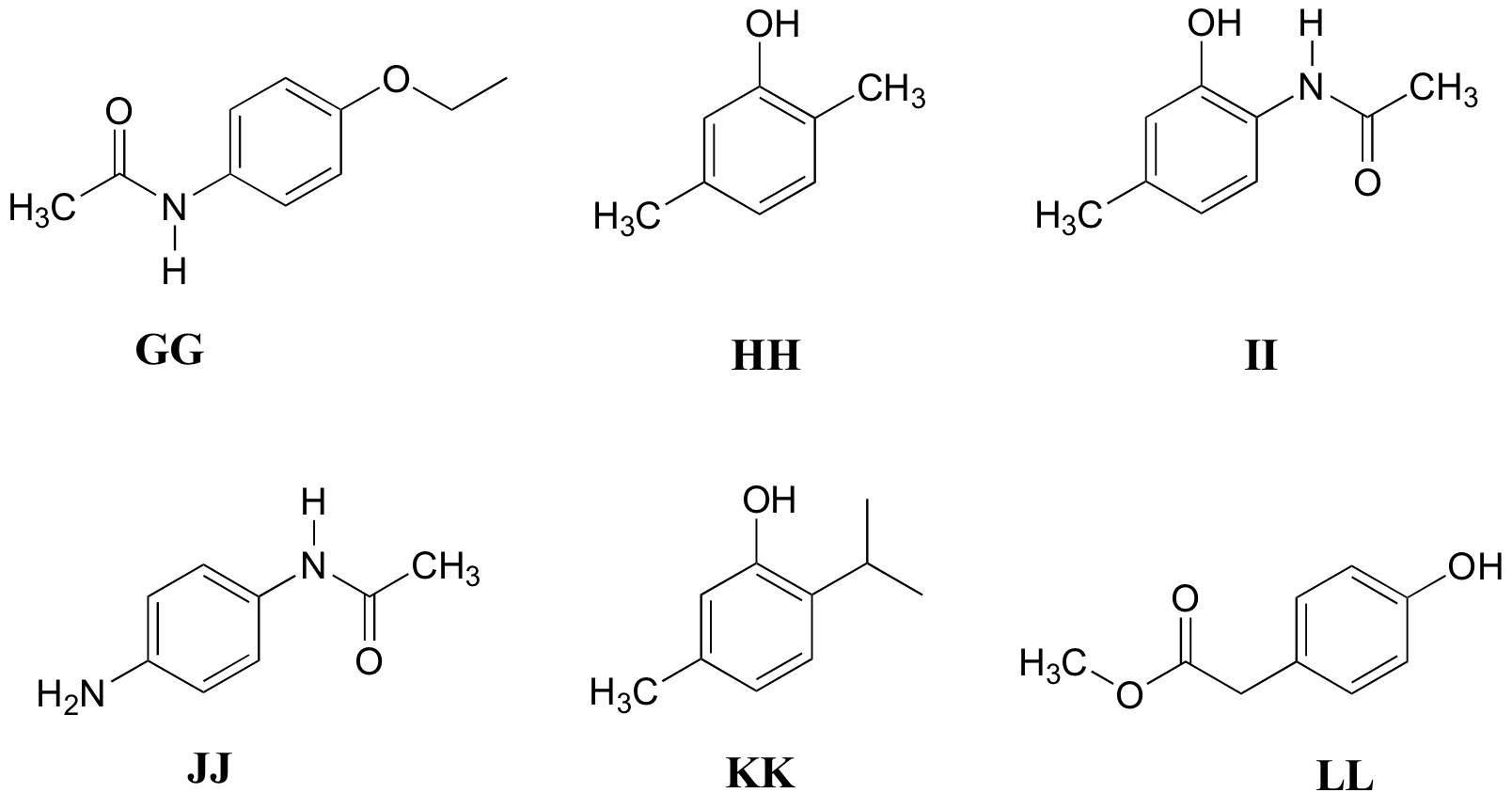
Espectro 31:
|
δ |
dividiendo |
integración |
|
6.98 |
d |
1 |
|
6.64 |
d |
1 |
|
6.54 |
s |
1 |
|
4.95 |
s |
1 |
|
2.23 |
s |
3 |
|
2.17 |
s |
3 |
Espectro 32:
|
δ |
dividiendo |
integración |
|
7.08 |
d |
1 |
|
6.72 |
d |
1 |
|
6.53 |
s |
1 |
|
4.81 |
s |
1 |
|
3.15 |
7-tet |
1 |
|
2.24 |
s |
3 |
|
1.22 |
d |
6 |
Espectro 33:
|
δ |
dividiendo |
integración |
|
7.08 |
d |
2 |
|
6.71 |
d |
2 |
|
6.54 |
s |
1 |
|
3.69 |
s |
3 |
|
3.54 |
s |
2 |
Espectro 34:
|
δ |
dividiendo |
integración |
|
9.63 |
s |
1 |
|
7.45 |
d |
2 |
|
6.77 |
d |
2 |
|
3.95 |
q |
2 |
|
2.05 |
s |
3 |
|
1.33 |
t |
3 |
Espectro 35:
|
δ |
dividiendo |
integración |
|
9.49 |
s |
1 |
|
7.20 |
d |
2 |
|
6.49 |
d |
2 |
|
4.82 |
s |
2 |
|
1.963 |
s |
3 |
Espectro 36:
|
δ |
dividiendo |
integración |
|
9.58 |
s (b) |
1 |
|
9.31 |
s |
1 |
|
7.36 |
d |
1 |
|
6.67 |
s |
1 |
|
6.55 |
d |
1 |
|
2.21 |
s |
3 |
|
2.11 |
s |
3 |
P5.12: Utilizar los datos de RMN dados para deducir estructuras.
a) Fórmula molecular: C 5 H 8 O
1 H-RMN:
|
δ |
dividiendo |
integración |
|
9.56 |
s |
1 |
|
6.25 |
d (J~1 Hz) |
1 |
|
5.99 |
d (J~1 Hz) |
1 |
|
2.27 |
q |
2 |
|
1.18 |
t |
3 |
13 C-RMN
|
δ |
DEPT |
|
194.60 |
CH |
|
151.77 |
C |
|
132.99 |
CH 2 |
|
20.91 |
CH 2 |
|
11.92 |
CH 3 |
b) Fórmula molecular: C 7 H 14 O 2
1 H-RMN:
|
δ |
dividiendo |
integración |
|
3.85 |
d |
2 |
|
2.32 |
q |
2 |
|
1.93 |
m |
1 |
|
1.14 |
t |
3 |
|
0.94 |
d |
6 |
13 C-RMN
|
δ |
DEPT |
|
174.47 |
C |
|
70.41 |
CH 2 |
|
27.77 |
CH |
|
27.64 |
CH 2 |
|
19.09 |
CH 3 |
|
9.21 |
CH 3 |
c) Fórmula molecular: C 5 H 12 O
1 H-RMN:
|
δ |
dividiendo |
integración |
|
3.38 |
s |
2H |
|
2.17 |
s |
1H |
|
0.91 |
s |
9H |
13 C-RMN
|
δ |
DEPT |
|
73.35 |
CH 2 |
|
32.61 |
C |
|
26.04 |
CH 3 |
d) Fórmula molecular: C 10 H 12 O
1 H-RMN:
|
δ |
dividiendo |
integración |
|
7.18-7.35 |
m |
2.5 |
|
3.66 |
s |
1 |
|
2.44 |
q |
1 |
|
1.01 |
t |
1.5 |
13 C-RMN
|
δ |
DEPT |
|
208.79 |
C |
|
134.43 |
C |
|
129.31 |
CH |
|
128.61 |
CH |
|
126.86 |
CH |
|
49.77 |
CH 2 |
|
35.16 |
CH 2 |
|
7.75 |
CH 3 |
P5.13:
13 Se dan datos de RMN C para las moléculas que se muestran a continuación. Completar la columna de asignación de picos de cada tabla de datos de RMN.
a)
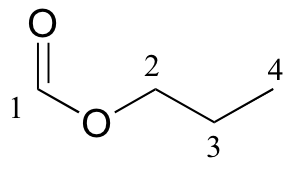
|
δ |
DEPT |
carbono # |
|
161.12 |
CH |
|
|
65.54 |
CH 2 |
|
|
21.98 |
CH 2 |
|
|
10.31 |
CH 3 |
b)
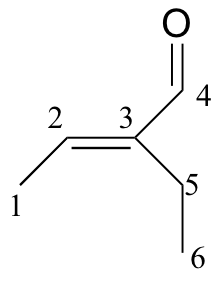
|
δ |
DEPT |
carbono # |
|
194.72 |
C |
|
|
149.10 |
C |
|
|
146.33 |
CH |
|
|
16.93 |
CH 2 |
|
|
14.47 |
CH 3 |
|
|
12.93 |
CH 3 |
c)
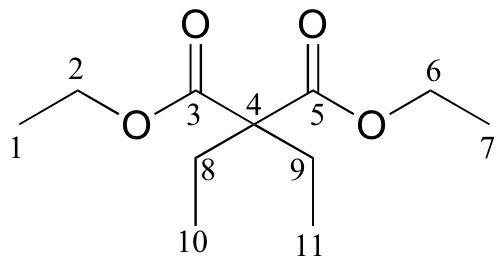
|
δ |
DEPT |
carbono # |
|
171.76 |
C |
|
|
60.87 |
CH 2 |
|
|
58.36 |
C |
|
|
24.66 |
CH 2 |
|
|
14.14 |
CH 3 |
|
|
8.35 |
CH 3 |
d)
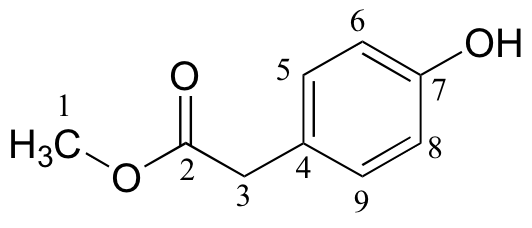
|
δ |
DEPT |
carbono # |
|
173.45 |
C |
|
|
155.01 |
C |
|
|
130.34 |
CH |
|
|
125.34 |
C |
|
|
115.56 |
CH |
|
|
52.27 |
CH 3 |
|
|
40.27 |
CH 2 |
e)
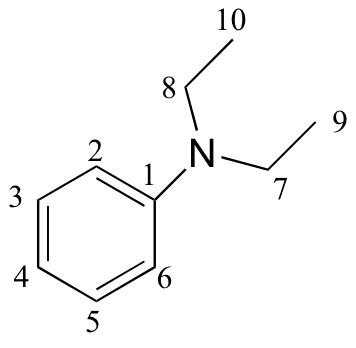
|
δ |
DEPT |
carbono # |
|
147.79 |
C |
|
|
129.18 |
CH |
|
|
115.36 |
CH |
|
|
111.89 |
CH |
|
|
44.29 |
CH 2 |
|
|
12.57 |
CH 3 |
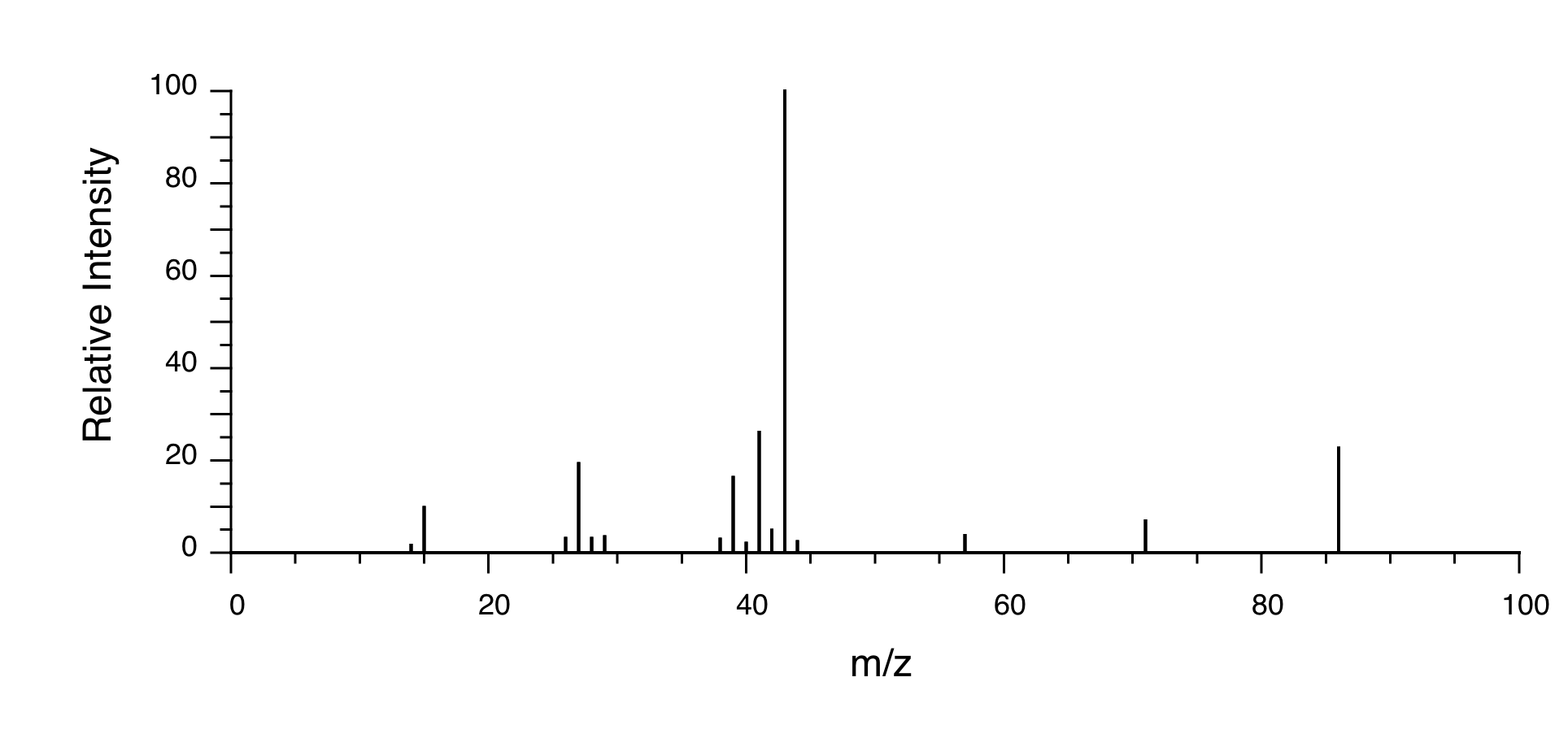
P5.14: Se obtienen los siguientes datos para una muestra desconocida. Deducir su estructura.
1 H-RMN:
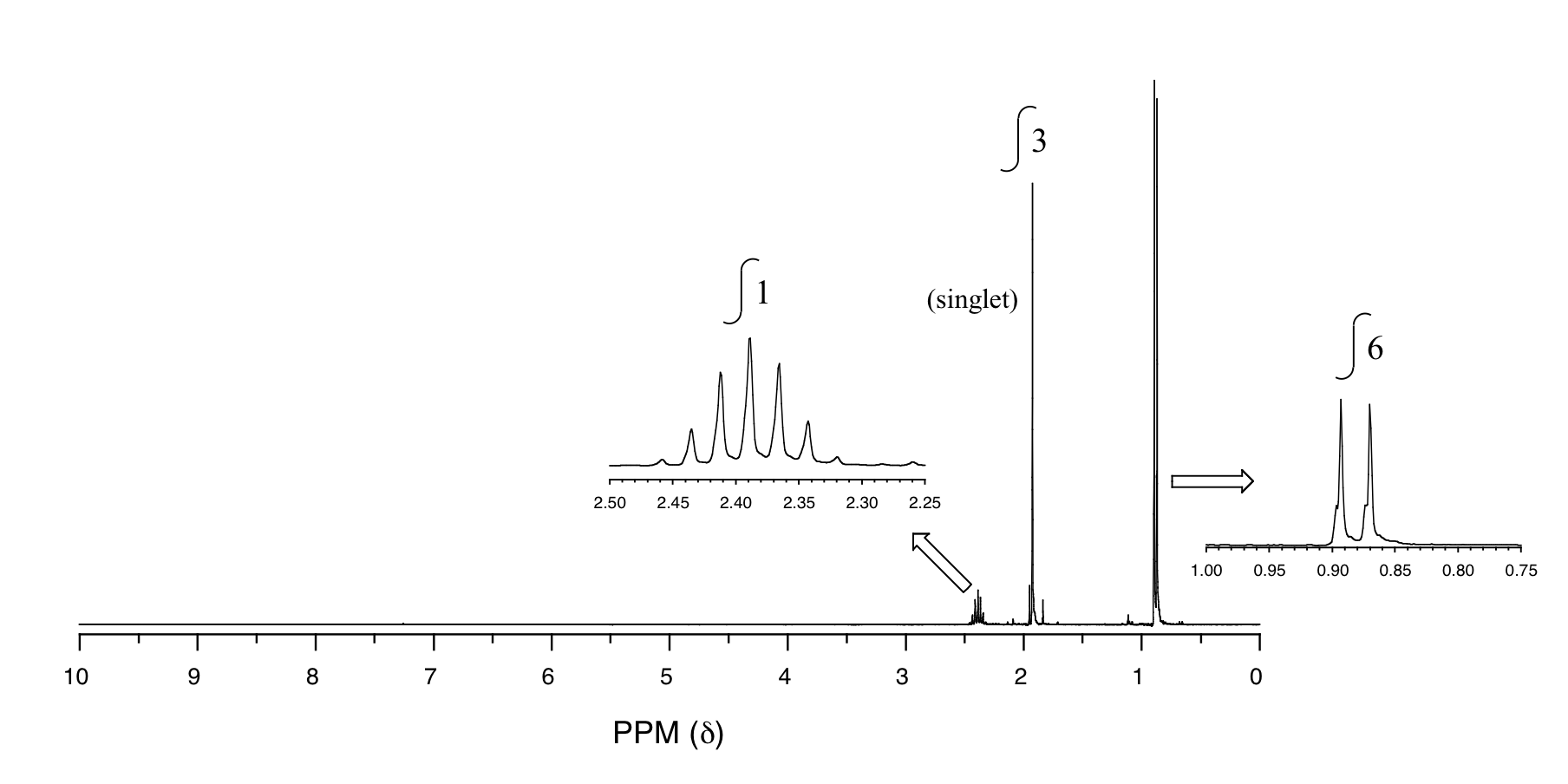
13 C-RMN:
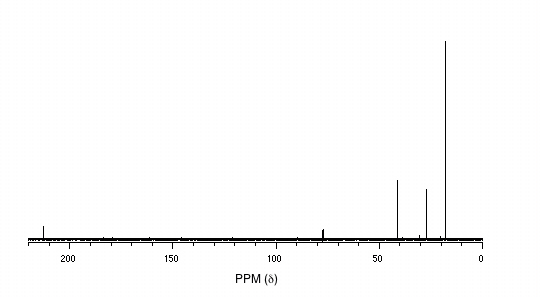
Espectrometría de Masas:
P5.15: Se toma un espectro de 1H-RMN de una muestra que proviene de una botella de 1-bromopropano. Sin embargo, sospechas que la botella podría estar contaminada con 2-bromopropano. El espectro de RMN muestra los siguientes picos:
|
δ |
dividiendo |
integración |
|
4.3 |
septeto |
0.0735 |
|
3.4 |
triplete |
0.661 |
|
1.9 |
sexteto |
0.665 |
|
1.7 |
doblete |
0.441 |
|
1.0 |
triplete |
1.00 |
¿Qué tan mal está contaminada la botella? Específicamente, ¿qué porcentaje de las moléculas en la botella son 2-bromopropano?
Problemas de desafío
C5.1: Todos los 13 espectros de RMN C mostrados en este capítulo incluyen una señal debida a CDCl 3, el disolvente utilizado en cada caso. Explique el patrón de división para esta señal.
C5.2: Los investigadores quisieron investigar una reacción que pueda ser catalizada por la enzima alcohol deshidrogenasa en levaduras. Trataron 4'-acilpiridina (1) con levadura viva y aislaron el producto (s) alcohólico (alguna combinación de 2A y 2B).
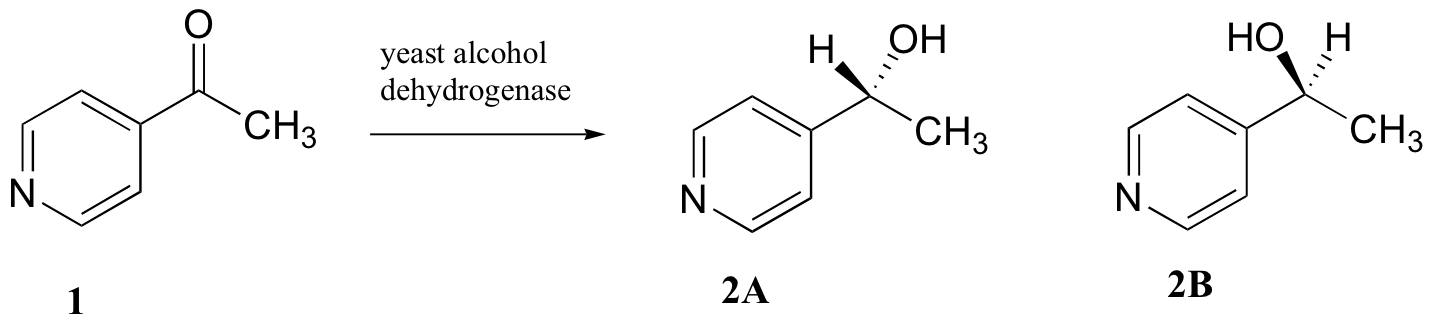
a) ¿Los productos 2A y 2B tendrán espectros 1H-NMR idénticos o diferentes? Explique.
b) Sugerir un experimento de 1H-RMN que podría usarse para determinar qué porcentaje de material de partida (1) se convirtió en producto (2A y 2B).
c) Con 2A/2B purificado, los investigadores llevaron a cabo la reacción posterior que se muestra a continuación para elaborar 3A y 3B, conocidos como 'Éster de Mosher'. ¿3A y 3B tienen espectros 1H-NMR idénticos o diferentes? Explique.
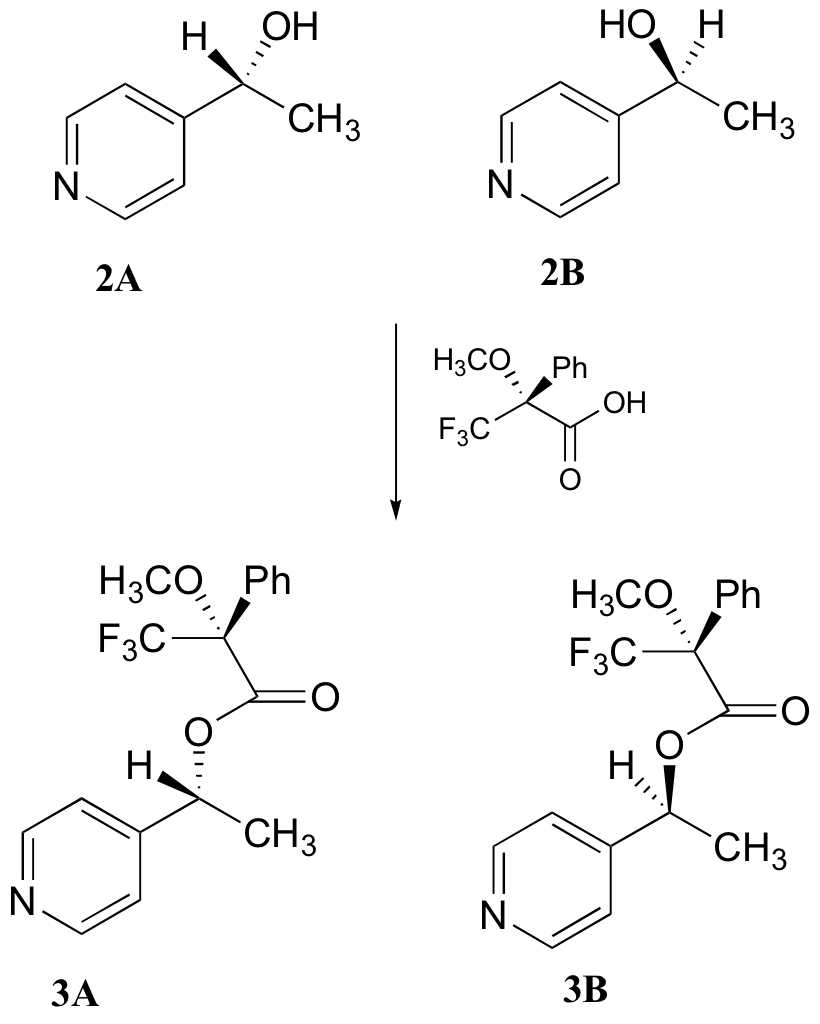
d) Explicar, muy específicamente, cómo los investigadores podrían utilizar 1H-NMR para determinar las cantidades relativas de 2A y 2B formadas en la reacción catalizada por la enzima de levadura.

