8.3: Estructura del ADN
- Page ID
- 54700
A. Pistas tempranas y conceptos erróneos continuos
Para 1878, se demostró que una sustancia en el pus de soldados heridos derivada de núcleos celulares (llamada nucleina) estaba compuesta por 5 bases (las familiares de ADN y ARN). Se pensó que las cuatro bases que se sabía que componían el ADN (como parte de los nucleótidos) estaban conectadas a través de los grupos fosfato en cadenas cortas de repetición de cuatro nucleótidos. Para la década de 1940, sabíamos que el ADN era un polímero largo. Sin embargo, todavía se consideró demasiado simple para dar cuenta de los genes (ver arriba). Después de los experimentos de Hershey y Chase, solo unos pocos holdouts no aceptarían el ADN como material genético. Entonces, la pregunta que quedaba era cómo una molécula tan “simple” podría dar cuenta de todos los genes, incluso en un organismo tan simple como bacteria. La respuesta a esta pregunta fue mentir al menos en parte en una comprensión de la estructura física del ADN, posible gracias al advenimiento de la Cristalografía de Rayos X.
Si una sustancia puede cristalizarse, el cristal difractará los rayos X en ángulos revelando estructuras regulares (repetitivas) del cristal. William Astbury demostró que el ADN de alto peso molecular tenía una estructura tan regular. Sus cristalografías sugerían que el ADN era un polímero lineal de bases apiladas (nucleótidos), cada nucleótido separado del siguiente por 0.34 nm. Astbury también es recordado por acuñar el término “biología molecular” para describir sus estudios. El término abarca ahora como todos los aspectos de la estructura biomolecular, así como las funciones moleculares (ej. replicación, transcripción, traducción, regulación génica...).
En una ironía de la historia, el biólogo ruso Nikolai Koltsov ya había intuido en 1927 que la base de la transferencia genética de rasgos sería una “molécula hereditaria gigante” compuesta por “dos hebras espejo que se replicarían de manera semiconservadora utilizando cada hebra como plantilla”. ¡Una inferencia bastante fantástica si lo piensas ya que se propuso mucho antes de que Watson y Crick y sus colegas trabajaran la estructura de la doble hélice de ADN!
Wilkins, Franklin, Watson y Crick
Maurice Wilkins, un bioquímico inglés, fue el primero en aislar ADN altamente puro y de alto peso molecular. Trabajando en el laboratorio de Wilkins, Rosalind Franklin pudo cristalizar este ADN y producir imágenes de difracción de rayos X de muy alta resolución de los cristales de ADN. La cristalografía más famosa (y definitiva) de Franklin fue “Foto 51”, que se muestra a continuación.
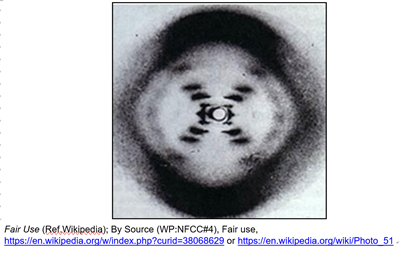
Esta imagen confirmó la dimensión de repetición de 0.34 nm de Astbury y reveló dos números más, 3.4 nm y 2 nm, reflejando estructuras de repetición adicionales en el cristal de ADN. Cuando James Watson y Francis Crick se apoderaron de estos números, los usaron junto con otros datos para construir modelos de ADN a partir de tuercas, pernos y plomería. Sus modelos finalmente revelaron que el ADN era un par de antiparalelos complementarios de polímeros de ácidos nucleicos..., ¡sombras de macromoléculas de imagen especular de Koltsov! Cada cadena es una cadena de nucleótidos unidos por enlaces fosfodiéster, las dos cadenas se mantienen juntas en una doble hélice mediante interacciones complementarias de enlace H.
Veamos la evidencia de estas conclusiones y como lo hacemos, nos referimos a las dos ilustraciones de la doble hélice a continuación.
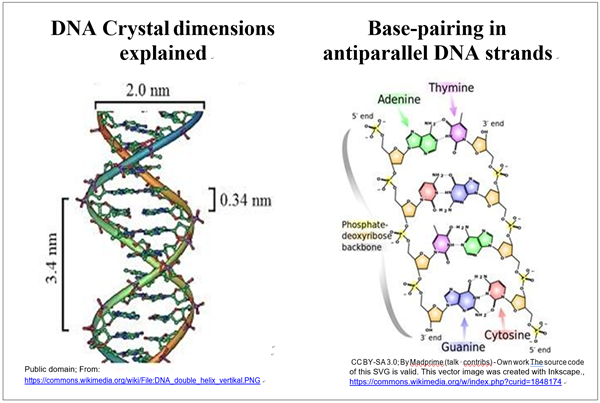
Recordando que la dimensión de 0.34 nm de Astbury era la distancia entre nucleótidos sucesivos en una cadena de ADN, Watson y Crick supusieron que la repetición de 3.4 nm era un múltiplo estructuralmente significativo de 10 veces del número de Astbury. Cuando comenzaron a construir sus modelos de ADN, se dieron cuenta desde los ángulos de enlace que conectaban los nucleótidos que la hebra estaba formando una hélice, de lo que concluyeron que la repetición de 3.4 nm era el paso de la hélice, es decir, la distancia de una vuelta completa de la hélice. Esto significó que había 10 bases por giro de la hélice. Además, razonaron que el número de 2.0 nm podría reflejar el diámetro de la hélice. Cuando su modelo a escala de una hélice de ADN monocatenario predijo un diámetro helicoidal mucho menor a 2.0 nm, pudieron modelar una hélice doble que casi cumplió con el requisito de 2.0 nm de diámetro. Al construir su doble hélice, Watson y Crick se dieron cuenta de que las bases en hebras opuestas se unirían para formar enlaces H, manteniendo unida la hélice. Sin embargo, para que su doble hélice tenga un diámetro constante de 2.0 nm, también se dieron cuenta de que las bases de pirimidina más pequeñas, Timina (T) y Citosina (C), tendrían que unirse H a la purina más grande bases, Adenina (A) y Guanosina (G).
Ahora a la pregunta de cómo una molécula de ADN “simple” podría tener la diversidad estructural necesaria para codificar miles de polipéptidos y proteínas diferentes. En estudios tempranos, el ADN purificado de E. coli se hidrolizó químicamente hasta monómeros nucleotídicos. Los productos de hidrólisis contenían cantidades casi iguales de cada base, reforzando la noción de que el ADN era esa molécula simple que no podía codificar genes. Pero Watson y Crick tuvieron acceso privado a revelar datos de Erwin Chargaff. Chargaff había determinado la composición base del ADN aislado de diferentes especies, incluyendo E. coli. Encontró que la composición base del ADN de diferentes especies no siempre fue equimolar, lo que significa que para algunas especies, el ADN no estaba compuesto por cantidades iguales de cada una de las cuatro bases (ver algunos de estos datos a continuación).
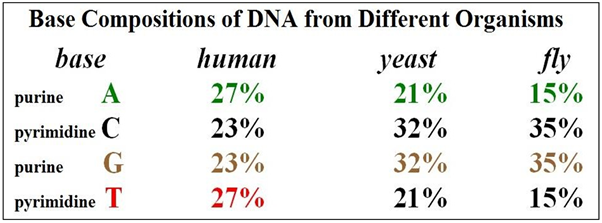
El mero hecho de que el ADN de algunas especies pudiera tener composiciones de base que se desviaran de la equimolaridad puso fin al argumento de que el ADN tenía que ser una secuencia muy simple. Por último, era seguro aceptarlo para aceptar lo obvio, es decir, que el ADN era efectivamente la “materia de genes”.
Los datos de Chargaff también mostraron un patrón único de razones de base. Aunque las composiciones de base pueden variar entre especies, la relación A/T y G/C siempre fue una, para cada especie. De igual manera la relación de (A+C)/(G+T) y (A+G)/(C+T). A partir de esta información, Watson y Crick dedujeron que A (una purina) se uniría H con T (una pirimidina), y G (una purina) se uniría H con C (una pirimidina) en la doble hélice. Al construir su modelo con esta nueva información, también encontraron que la unión H entre las bases complementarias sería máxima solo si las dos cadenas de ADN fueran antiparalelas, conduciendo a la estructura más estable de la doble hélice.
Watson y Crick publicaron sus conclusiones sobre la estructura del ADN en 1953 (Pulse aquí para leer su artículo seminal: Estructura molecular de los ácidos nucleicos: una estructura para el ácido nucleico de desoxirribosa. Su artículo también es famoso por predecir un mecanismo semiconservador de replicación, algo que Koltsov había predicho 26 años antes, aunque basado en la intuición... ¡y mucho menos evidencia!
Watson, Crick y Wilkins compartieron un Premio Nobel en 1962 por su trabajo sobre la estructura del ADN. Desafortunadamente, Franklin murió en 1958 y los premios Nobel no fueron otorgados póstumamente. Todavía hay controversia sobre por qué Franklin no obtuvo el crédito apropiado por su papel en la obra. Pero ha estado recibiendo un reconocimiento bien merecido y largamente retrasado, ¡incluida una universidad en Chicago nombrada en su honor!
169 Desentrañar la estructura del ADN
La confirmación de la sugerencia de Watson & Crick de replicación semiconservativa vino del experimento muy elegante de Meselson y Stahl, que probó los tres posibles modelos de replicación que se muestran a continuación.

En su experimento, las células de E. coli se cultivaron en medio que contenía 15N, un isótopo de nitrógeno 'pesado'. Después de muchas generaciones, todo el ADN de las células se había marcado con el isótopo pesado. En ese punto, las células marcadas con 15N se colocaron de nuevo en un medio que contenía el isótopo 14N “ligero” más común y se les permitió crecer durante exactamente una generación.
Las predicciones de Meselson y Stahl y su diseño experimental se muestran a continuación.
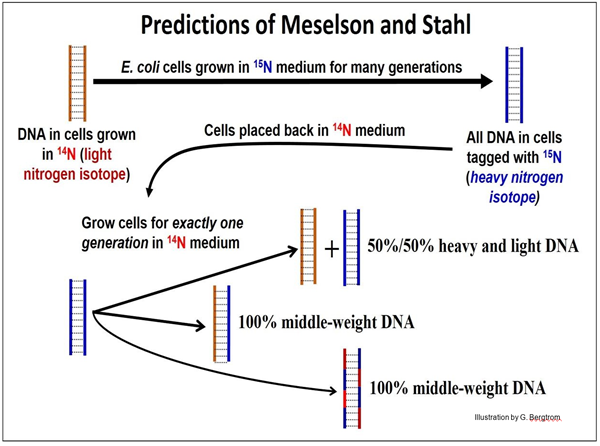
Meselson y Stahl sabían que el ADN marcado con 14N y marcado con 15N formaría bandas separadas después de la centrifugación en gradientes de densidad de cloruro de CsCl. Probaron sus predicciones purificando y centrifugando el ADN de las células marcadas con 15N cultivadas en medio 14N para una generación. Encontraron que este ADN formaba una sola banda con una densidad entre la del ADN marcado con 15N y el ADN marcado con 14N. Este resultado eliminó un modelo conservador de replicación del ADN (como también predijeron Watson y Crick. Eso dejó dos posibilidades: la replicación era semiconservativa o dispersiva. El modelo dispersivo se eliminó cuando se demostró que el ADN aislado de células cultivadas para una 2da generación en 14N contenía dos bandas de ADN en los gradientes de densidad de CsCl.
Cromosomas
Entendimos desde principios del siglo XX que los cromosomas contenían genes. Por lo tanto, se hace necesario comprender la relación entre cromosomas, cromatina, ADN y genes. Como se señaló anteriormente, los cromosomas son una versión condensada especializada de la cromatina, con características estructurales clave que se muestran a continuación.
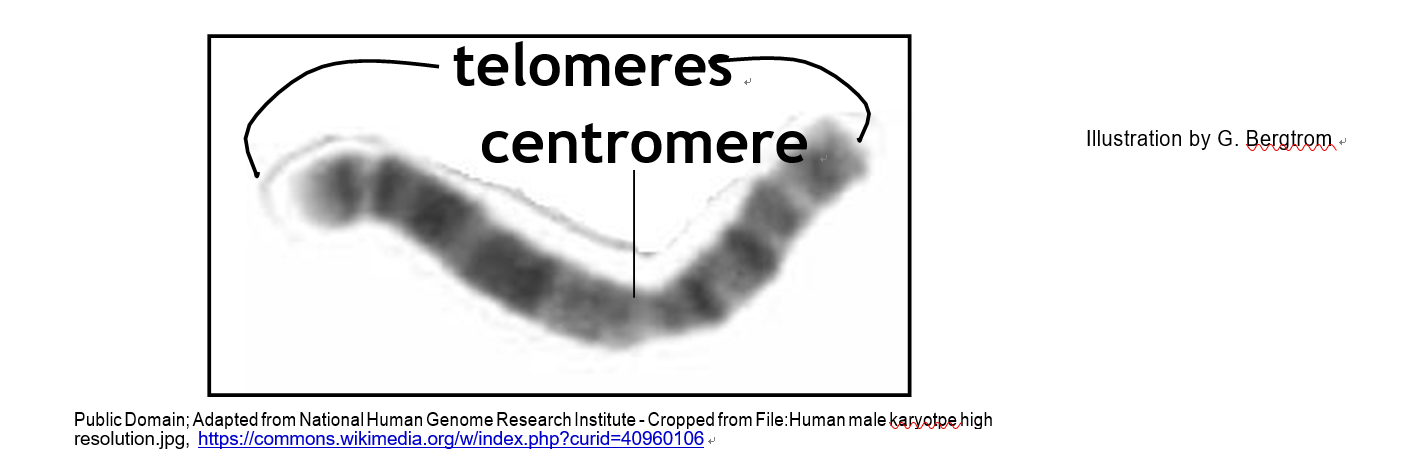
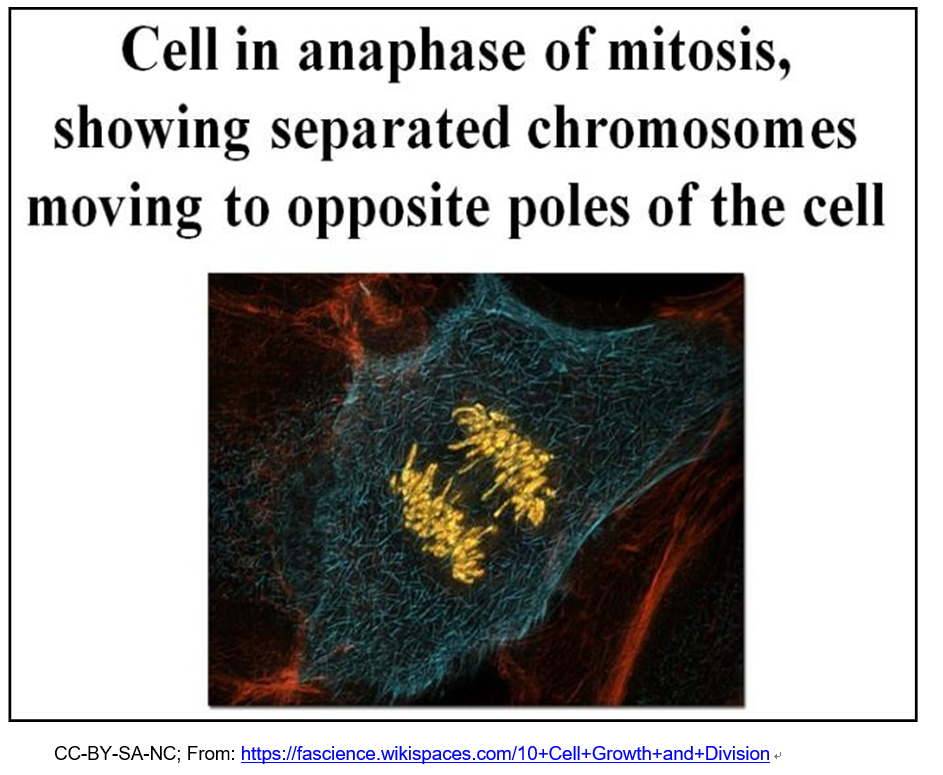
Ahora sabemos que la estructura compacta de un cromosoma previene el daño al ADN durante la división celular. Este daño puede ocurrir cuando las fuerzas sobre los centrómeros generadas por las fibras mitóticas o meióticas del huso separan a las cromátidas. A medida que el núcleo se descompone durante la mitosis o meiosis, los microscopistas de finales del siglo XIX vieron que los cromosomas se condensaban a partir del fondo citoplásmico disperso. Estos cromosomas permanecieron visibles a medida que se separaban, moviéndose a polos opuestos de la célula durante la división celular. Tales observaciones del comportamiento cromosómico durante la división celular señalaron su papel en la herencia. A continuación se muestra una célula coloreada por computadora en la mitosis.
Es posible distinguir un cromosoma de otro por cariotipo. Cuando las células en metafase de mitosis se ponen bajo presión, estallan y los cromosomas se separan. Tal diseminación cromosómica se muestra a continuación.
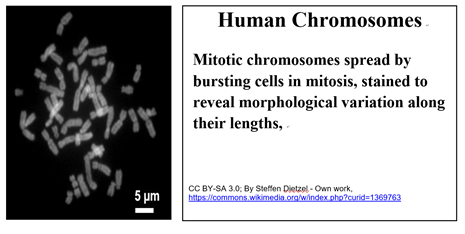
A principios del siglo XX, se demostró que el número, tamaños y formas de los cromosomas eran específicos de especie. Además, una mirada cercana a los diferenciales cromosómicos reveló que los cromosomas venían en pares morfológicamente emparejados. Esto fue tan reminiscente de los factores hereditarios pareados de Gregor Mendel que los cromosomas fueron entonces ampliamente aceptados como el asiento estructural de la herencia genética. Cortar micrografías como la anterior y emparejar los cromosomas por su morfología genera un cariotipo. Los homólogos humanos emparejados se identifican fácilmente en la micrografía coloreada a continuación.
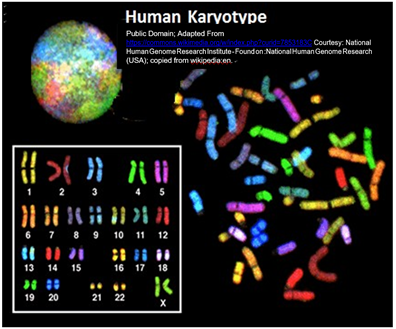
Capturadas en la mitosis, todas las células humanas en división contienen 23 pares de cromosomas homólogos. El cariotipo es de una hembra; nótese el par de cromosomas de sexo homólogo (“X”) (parte inferior derecha del recuadro). Los cromosomas X e Y en los machos no son verdaderamente homólogos. Los cromosomas en la diseminación original y en el cariotipo alineado teñidos con anticuerpos fluorescentes a secuencias de ADN específicas de cromosomas, 'iluminan' los diferentes cromosomas.


