3.4: Regulación de la Actividad Enzimática
- Page ID
- 53391
La Figura\(\PageIndex{7}\) (y 9) también ilustra los efectos de dos tipos diferentes de inhibición sobre los diferentes componentes de la cinética enzimática. Las enzimas pueden ralentizarse o incluso prevenirse de catalizar reacciones de muchas maneras, incluyendo evitar que el sustrato entre en el sitio activo o evitar que la enzima altere la conformación para catalizar la reacción. Los inhibidores que hacen esto pueden hacerlo de manera reversible o irreversible. Los inhibidores irreversibles también se llaman inactivadores, y o bien se unen a la enzima con una afinidad tan alta como para ser virtualmente irreversibles, o en realidad forman enlaces covalentes con la enzima. Los inhibidores reversibles generalmente se agrupan en dos tipos básicos: competitivos y no competitivos.
La finasterida (los nombres comerciales incluyen Propecia y Procar) es un inhibidor irreversible que se une muy fuertemente a la enzima 5-a-re- ductasa, utilizada en la conversión de la testosterona en dihidrotestosterona. Se utiliza en el tratamiento de la calvicie de patrón masculino, la hiperplasia proestática benigna y el cáncer de próstata.
La aspirina es un ejemplo de un inhibidor irreversible que en realidad forma un enlace covalente con la enzima. La aspirina (ácido acetilsalicílico) transfiere su grupo acetilo a un residuo de serina en la ciclooxigenasa-2 (COX-2). Esto detiene la producción de prostaglandinas productoras de amationes y tromboxanos por COX-2.
El metotrexato es un inhibidor competitivo de la dihidrofolato reductasa (DHFR), una enzima que sintetiza tetrahidrofolato, que es un precursor para la síntesis de purinas, y por lo tanto para ADN y ARN. Tiene una estructura molecular muy similar para el ácido fólico, el sustrato natural de DHFR. El metotrexato se usa como medicamento anticanceroso porque afecta a las células que se reproducen rápidamente (que necesitan producir ADN antes que otras células) más que las células no cancerosas.
La inhibición competitiva es quizás la más sencilla de entender. La molécula inhibidora compite directamente con el sustrato por el sitio activo de una enzima no unida. Si un inhibidor se une al sitio activo, el sustrato es incapaz de hacerlo hasta que el inhibidor haya desocupado el sitio. Por lo tanto, uno podría potencialmente abrumar la inhibición competitiva con concentraciones suficientemente mayores de sustrato para que la probabilidad de que la enzima choque en un sustrato para unirse sea mayor en comparación con la probabilidad de chocar con un inhibidor. Luego se logra V max normal, desinhibido a pesar de la presencia del inhibidor competitivo, el cual solo ha afectado al K m, es decir, la concentración de sustrato necesaria para alcanzar V max /2. Esta es la firma cinética de los inhibidores competitivos: con concentraciones crecientes de inhibidores, K M aumenta pero V max no se ve afectado.
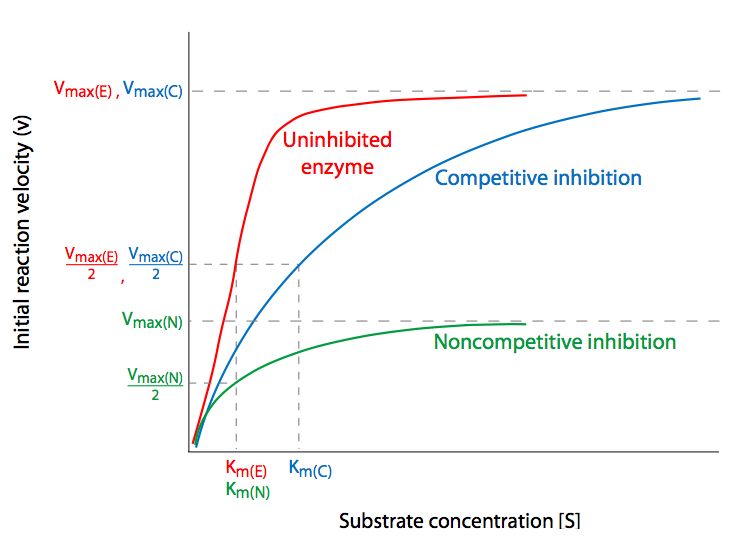
La inhibición no competitiva implica inhibir la enzima alterando su capacidad para completar la reacción catalizada a través de la unión de la enzima en una posición que no es el sitio activo. Cuando el inhibidor se une a la enzima, provoca un cambio, generalmente conformacional, que puede impedir que la enzima se una al sustrato, o impedir que la enzima actúe sobre un sustrato unido. En cualquier caso, aumentar la disponibilidad de sustrato no superará en última instancia el efecto del inhibidor. Así,\(V_{max}\) se reduce no porque alguna proporción de las enzimas ya no sean utilizables, sino porque las enzimas que están disponibles tienen el mismo acceso al sustrato que lo haría sin inhibidor (es decir, no está en competencia con un inhibidor), la no\(K_m\) se ve afectada.
Allostery
La regulación no competitiva es un ejemplo de regulación alostérica de enzimas. Las interacciones alostéricas ocurren cuando la unión de un ligando (no necesariamente un sustrato) a una proteína influye en la unión de otro ligando a la proteína en un sitio de unión separado. Este tipo de interacciones pueden ser positivas (activadoras) o negativas (inhibidoras), y bien homotrópicas (ambos ligandos son idénticos) o heterotrópicas (los ligandos son diferentes). Curiosamente, a veces el ligando regulador puede ser realmente un producto de la reacción catalizada. En este tipo de mecanismo de retroalimentación, el progreso de una reacción es autorregulado.
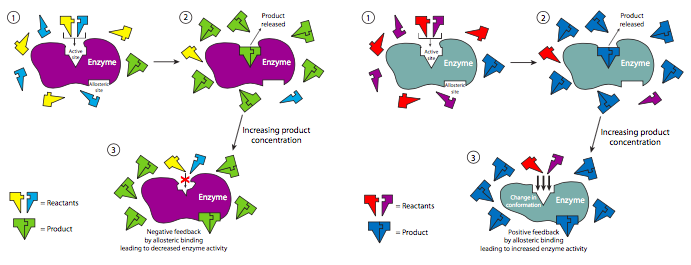
Muchas de las enzimas en las vías metabólicas (capítulos 5 y 6) están reguladas por un inhibidor no competitivo de origen natural. Un ejemplo es la fosfofructoquinasa (PFK), la cual está involucrada en la glucólisis, la cual produce ATP para la célula. Sin embargo, si hay niveles suficientemente altos de ATP en la célula que otros procesos celulares no están usando, entonces puede unirse a la fosfofructoquinasa fuera de su sitio activo (se une a la fructosa-6-fosfato) y apagarla. Esto bloquea la glucólisis y la producción de exceso de ATP cuando la célula no lo necesita. A medida que se agota el ATP, hay menos disponible para inhibir PFK y la glucólisis comienza de nuevo.
Modelado del comportamiento alostérico
Existen dos modelos para tales interacciones alostéricas.
- El modelo de simetría, también conocido como modelo concertado, o modelo MWC (Monod, Wyman y Changeux, 1965), propone que la enzima alostérica es un oligómero de varias subunidades, cada una de las cuales está simétricamente relacionada, y puede estar en un estado “tensado” o “relajado”, pero todas las subunidades están en el mismo estado y en equilibrio. Cuando un ligando se une, cambia el estado de la subunidad o subunidades a las que se une, y para mantener el equilibrio, que a su vez hace que el estado de las otras subunidades coincida, alterando así las propiedades de unión para un ligando posterior.
- El modelo secuencial, o el modelo KNF (Koshland, Nemethy y Filmer, 1966), propone algo bastante diferente: aunque también supone subunidades en estados tensos o relajados, no requiere que todas las subunidades estén conectadas de tal manera que manden que todas las subunidades estén en el mismo estado , y por lo tanto los cambios conformacionales en una subunidad no necesitan propagarse a todos. En cambio, usando un modelo de ajuste inducido de unión al ligando en lugar del mecanismo básico más rígido de bloqueo y llave, sugiere que cuando un ligando se une a la enzima, induce un ligero cambio conformacional en el sitio activo que aumenta su afinidad por el ligando. El cambio conformacional puede alterar ligeramente la conformación de otras subunidades de la enzima, pero puede no constituir un cambio de estado entre relajado y tenso dependiendo de cuán estrechamente interaccionen las subunidades. Sin embargo, el cambio es suficiente para aumentar la afinidad del sustrato en subunidades adyacentes.
Avanzado: Inhibición no competitiva y mixta
La mayoría de los cursos celulares/moleculares detienen la discusión de inhibidores enzimáticos a nivel competitivo vs no competitivo basado en sus perfiles cinéticos. No obstante, cabe señalar que si tomas un curso de bioquímica, es posible que encuentres los términos inhibidor no competitivo e inhibidor mixto. Estos términos se definen no solo por la cinética enzimática, sino por el mecanismo de interacción:
- Los inhibidores no competitivos se unen solo al complejo enzima-sustrato (ES), y no a la enzima antes de que haya encontrado sustrato. Esto conduce a una disminución de V max y disminución de K m.
- La inhibición mixta significa que el inhibidor puede unirse a la enzima sola o al complejo enzima-sustrato. Debido a que las afinidades del inhibidor por las dos formas de la enzima son diferentes, y porque parte de ella depende de la concentración del sustrato mientras que el otro tipo de unión no lo hace, generalmente, V max disminuye y K m aumenta. La inhibición no competitiva es un caso especial de inhibición mixta en el que se disminuye o se suprime la actividad catalítica de la enzima, pero la capacidad de unirse al sustrato es inalterada.
Otros Mecanismos de Inhibición
Si bien es importante, especialmente en el ámbito farmacéutico, el uso de inhibidores enzimáticos no es la única forma de regular las enzimas. Existen numerosos ejemplos de un tipo de enzima que activa o inhibe a otro. El ejemplo general más común son las proteínas quinasas. Estas enzimas fosforilan (transfieren el grupo fosfato a) otras enzimas y con ello las activan. Las quinasas son generalmente rápidas y muy específicas, y este es un método eficiente para activar rápidamente grandes cantidades de enzimas particulares. Por el contrario, las proteínas fosfatasas son enzimas (también bastante rápidas, pero mucho menos específicas que las quinasas) que eliminan los grupos fosfato de las proteínas fosforiladas, desactivando así esas enzimas. Hay que tener en cuenta que esto es una generalización, y que no todas las fosforilaciones se están activando. Además de la inhibición enzimática de las enzimas, también hay inhibición por unión y secuestro de los sustratos. De hecho, el antibiótico vancomicina funciona justamente de esta manera, uniéndose al péptido sustrato para la transpeptidasa y evitando que la enzima la reconozca. La transpeptidasa normalmente ayuda a estabilizar la pared celular de ciertas bacterias alterando algunas de las proteínas, y sin su actividad, la protección de la pared celular se ve comprometida y las bacterias pueden ser destruidas más fácilmente.
Más de la mitad de las enzimas descubiertas hasta ahora no actúan en el mecanismo simplista de Michaelis-Menten uno-sustrato-uno-producto, sino que operan con dos sustratos y dos productos, generalmente con la transferencia de un grupo activo. Este tipo de reacciones a veces se conocen como reacciones Bi Bi. Existen dos clases principales de estas reacciones: las reacciones secuenciales, en las que todos los sustratos se unen con la enzima antes de que continúe la reacción, y las reacciones de ping pong, en las que se crean y liberan uno o más productos antes de que todos los sustratos hayan sido encuadernado. De hecho, a diferencia de las reacciones secuenciales, los dos sustratos no interactúan entre sí mientras se unen a la enzima.
Condiciones Óptimas
La actividad de las enzimas está muy influenciada tanto por el pH como por la temperatura, como se esperaba de la discusión sobre la estructura proteica en el capítulo anterior. Los perfiles de actividad de la mayoría de las enzimas muestran un pico de actividad que se reduce a ambos lados, ya sea pH o temperatura. Esta es una característica innata de la enzima. Por ejemplo, la pepsina, una enzima digestiva secretada en el estómago (pH 2) no funciona cuando el pH > 5. Por otro lado, otra enzima digestiva, la tripsina, que se secreta al duodeno (intestino delgado proximal) donde el pH es ~8, no funciona en ambientes ácidos. Los cambios en el pH pueden cambiar la ionización de las cadenas laterales de aminoácidos que pueden alterar la interacción con el sustrato, o conducir a cambios en la estructura terciaria.
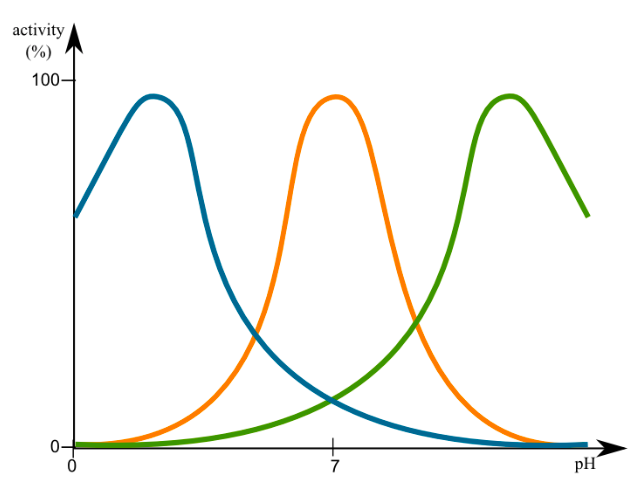
De igual manera, a temperaturas subóptimas, la probabilidad de interacción proteína-sustrato es baja pero por encima de la temperatura óptima, el aumento de la energía puede conducir a la ruptura de los enlaces de hidrógeno dentro de la estructura de la enzima, dando como resultado cambios que activan la capacidad catalítica de la enzima o impiden que sustrato de unión con suficiente afinidad. La temperatura óptima de la mayoría de las enzimas es muy cercana a su ambiente típico. Por lo tanto, una enzima humana operaría de manera óptima alrededor de 37 °C, mientras que una enzima de bacterias que viven en respiraderos volcánicos de aguas profundas (por ejemplo, Thermophilus aquaticus) podría tener un óptimo de temperatura superior a 90 °C. Esta es una de las razones por las que la refrigeración puede ralentizar el crecimiento de microorganismos (lo que obviamente no tienen capacidad para regular su temperatura), y por qué la mayoría de los microorganismos son destruidos (enzimas desnaturalizadas permanentemente) cuando se ponen en ambientes sostenidos de alta temperatura. Curiosamente, la ADN polimerasa de la bacteria T. aquaticus, también comúnmente llamada polimerasa Taq, se utiliza en una técnica de laboratorio de amplificación rápida de ADN conocida como PCR (reacción en cadena de la polimerasa, ver capítulo Métodos) en la que las muestras se calientan repetidamente a altas temperaturas para separar las cadenas de ADN en preparación para hacer copias de los mismos. Las ADN polimerasas de la mayoría de las especies procariotas o eucariotas serían desnaturalizadas e in- activadas por el alto calor, pero Taq ha evolucionado (con respecto a su estructura terciaria) para una estabilidad estructural extraordinaria incluso en extremos térmicos.
Finalmente, muchas enzimas requieren un compañero molecular que no tiene actividad catalítica propia, pero como un catalizador, no se ve alterado permanentemente por la reacción química. Estas moléculas son cofactores. Algunos son simples: elementales, de hecho, incluyendo iones metálicos como Zn 2+ o Ca 2+. Otros son un poco más complejos: los cofactores orgánicos pequeños se llaman coenzimas, y logran lo mismo, actuando como un socio requerido de la enzima en la catalización de una reacción. La interacción con la propia enzima varía y puede ser solo transitoria, como en NAD + /NADH que son coenzimas utilizadas en reacciones redox, o unidas permanentemente a la enzima por enlace covalente como el grupo hemo de la hemoglobina. A menudo la función de la coenzima es proporcionar un grupo activo para facilitar la reacción catalizada. La coenzima A, en diversas vías metabólicas como la glucólisis o el ciclo del ácido tricarboxílico, puede unirse a un sustrato para formar un producto estable que luego actúa como intermedio. El Co-A se libera de la molécula a medida que experimenta el siguiente paso en una serie de reacciones en la vía metabólica (ver Capítulo 5).
Desde el punto de vista de la salud humana, es interesante señalar que muchas coenzimas son vitaminas, o derivadas de vitaminas. Estas son las vitaminas B biotina (B 7), cobalamina (B 12), ácido fólico (B 3), niacina/nicotinamida (B 9), ácido pantoténico (B 5), piridoxina (B 6), riboflavina (B 2) y tiamina (B 1). Las vitaminas son pequeños compuestos orgánicos que no son sintetizados por un organismo y por lo tanto deben ser ingeridos. Generalmente son necesarios solo en pequeñas cantidades, pero necesarios no obstante. Naturalmente, las vitaminas con las que estamos familiarizados son las que requieren los humanos. Los roles específicos de estas vitaminas y las secuencias de no tener suficientes de ellas se discuten más adelante en este libro de texto, ya que se introducen en detalle las enzimas con las que trabajan.


