10.6: Traducción procariota
- Page ID
- 53374
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Tan pronto como el ARN ha emergido del RNAP y hay suficiente espacio para acomodar un ribosoma, la traducción puede comenzar en procariotas. De hecho, para genes altamente expresados, ¡no sería inusual ver múltiples ARN polimerasas transcribiendo el ADN y múltiples ribosomas en cada uno de los transcritos traduciendo el ARNm en proteína! El proceso comienza con la subunidad ribosómica pequeña (y solo la subunidad pequeña, si está unida a la subunidad grande, es incapaz de unirse al ARNm), que se une al ARNm de manera suelta y comienza a escanearlo en busca de una secuencia de reconocimiento llamada secuencia Shine-Dalgarno, después de sus descubridores. Una vez que esto es reconocido por el ARNr de subunidad ribosómica pequeña, la subunidad pequeña se posiciona alrededor del codón de inicio (AUG). Este proceso se ve facilitado por factores de iniciación de la siguiente manera.
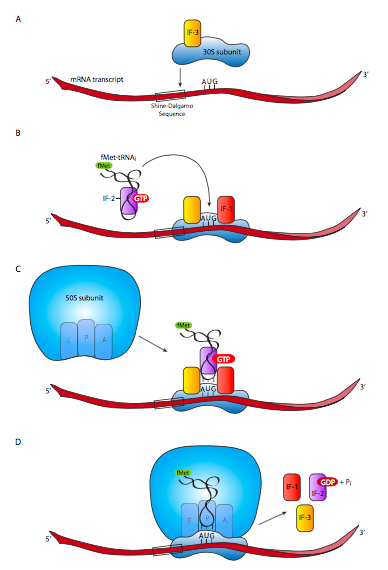
La subunidad ribosómica 30S se disocia de la subunidad ribosómica 50S si se asoció con una, y se une a los factores de iniciación IF-1 e IF-3. IF-1 se une al sitio A, donde evita que nuevas moléculas de aminoacil-ARNt entren antes de ensamblar el ribosoma completo. También facilita el montaje y estabilización del complejo de iniciación. IF-3 es necesario para permitir que la subunidad 30S se una al ARNm. Una vez ocurrido esto, IF-2-GTP llega a escena, llevando consigo el iniciador Aminoacil-ARNt. Esto se asienta en el sitio P, que se posiciona de manera que el anticodón del ARNt se asiente sobre el codón de inicio AUG del ARNm. La hidrólisis del GTP unido a IF-2 y la liberación de todos los factores de iniciación es necesaria para permitir que la subunidad 50S se una a la subunidad 30S para formar el ribosoma completo y completamente funcional. Debido a que se requirió la hidrólisis de GTP, la unión de las subunidades es irreversible espontáneamente y requiere un gasto de energía al finalizar la traducción. Una vez que la subunidad 50S se une a la subunidad 30S, el sitio A está listo para aceptar el siguiente aminoacil-ARNt.
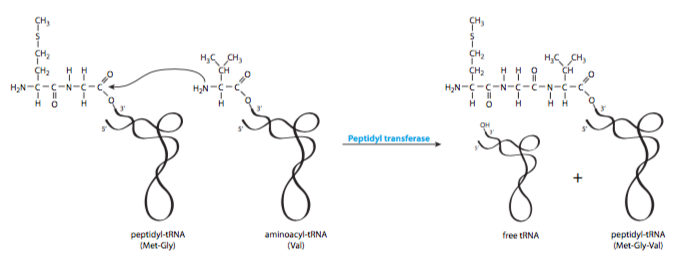
Un concepto erróneo común y comprensible es que el nuevo aminoácido traído al ribosoma se agrega a la cadena polipeptídica en crecimiento. De hecho, el mecanismo es exactamente lo contrario: el polipéptido se agrega al nuevo aminoácido (Figura\(\PageIndex{4}\)). Esto comienza a partir del segundo aminoácido para ser agregado a una nueva proteína (Figura\(\PageIndex{5}\)). El primer aminoácido, una metionina, debe recordar, vino junto con IF-2 y el ARNt iniciador. El nuevo aminoacil-ARNt es escoltado por EF-Tu, un factor de elongación que porta un GTP. Una vez que el AA-ARNt está en su lugar, EF-Tu hidroliza el GTP y se disocia del aminoacil-ARNt y el ribosoma.
Durante mucho tiempo, hubo un poco de misterio en torno al acoplamiento simultáneo de dos moléculas de ARNt en codones de ARNm inmediatamente adyacentes. En condiciones normales, no debería haber suficiente espacio, ya que los ARNt son bastante voluminosos y uno debe impedir que el otro alcance el ARNm para hacer una coincidencia codon-anticodón. La materia finalmente se aclaró en 2001 con exámenes cristalográficos de rayos X que mostraron una curva en el ARNm entre el codón en la ranura P y el codón en la ranura A. La curva coloca los dos ARNt asociados en ángulos ligeramente diferentes y, por lo tanto, crea suficiente espacio para que ambos mantengan enlaces de hidrógeno de formación de bases con el ARNm. Ver Yusupov et al, Science 292 (5518): 883-896, 2001.
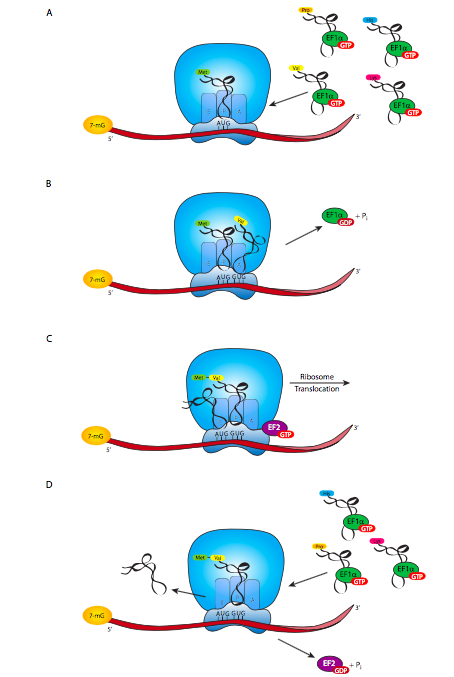
Cuando un nuevo aminoacil-ARNt cae en la ranura A del ribosoma, el anticodón se alinea con el codón del ARNm. Si no hay complementariedad, el aminoacil-ARNt pronto flota de nuevo fuera de la ranura para ser reemplazado por otro candidato. Sin embargo, si hay complementariedad (o algo lo suficientemente cercano, recordando la idea de bamboleo) entonces se forman enlaces H entre el codón y el anti-codón, el ARNt cambia de conformación, lo que desplaza la conformación de EF-Tu, provocando la hidrólisis de GTP a GDP + P i, y liberación del aA-ARNt. La interacción codon-anticodón es estable el tiempo suficiente para que la actividad catalítica del ribosoma hidroliza el enlace entre fMet y el ARNt en la ranura P, y unir el fMet al nuevo aminoácido con un enlace peptídico en la ranura A. El nuevo aminoácido todavía está unido a su ARNt, y a medida que ocurre este proceso, el ribosoma cambia de posición con respecto al ARNm y ARNt. Esto pone el ARNt ahora vacío (sin aminoácidos unidos) en la ranura E, el ARNtAA en la ranura P, unido a ese aa que está unido a Met, y la ranura A está nuevamente abierta para que entre un nuevo ARNt. El factor de elongación EF-G se une cerca de la ranura A tan pronto como EF-Tu sale, y es requerido para la translocación ribosómica, proporcionando energía para el proceso hidrolizando un GTP que lleva consigo al ribosoma. A partir de las experiencias de mis alumnos, la mejor manera de aprender esto parece ser estudiar los diagramas y ver los movimientos de las moléculas, rellenando los detalles mecanicistas en tu mente. Este proceso continúa hasta que el ribosoma alinea la ranura A con un codón de parada.
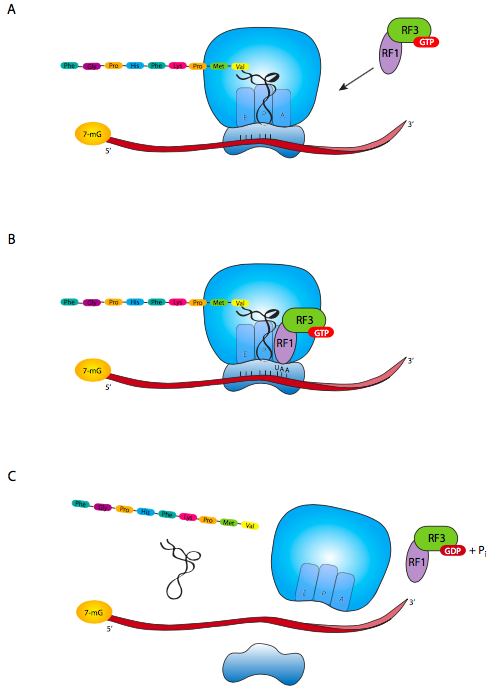
No hay ARNt con un anticodón para el codón de terminación. En cambio, hay un conjunto de factores de liberación que t en el sitio A del ribosoma, se unen al codón de terminación y activan el ribosoma para cortar el enlace entre la cadena polipeptídica y el último ARNt (Figura\(\PageIndex{6}\)). Dependiendo de qué codón de parada esté presente, ya sea RF1 (reconoce UAA o UAG) o RF2 (para UAA o UGA) primero entra en la ranura A. El RF1 o RF2 está complejado con RF3, que está involucrado en la liberación posterior del complejo de RF de la ranura A. Esto es necesario porque una vez que el polipéptido ha sido liberado del ribosoma, se debe liberar el ARNm. El factor liberador de ribosomas (RRF) también se une en la ranura A, lo que provoca un cambio conformacional en el ribosoma liberando el ARNt anterior y ahora vacío. Finalmente, EF-G se une a RRF, y con una hidrólisis acompañante de GTP, provoca la dissociación del ribosoma en subunidades grandes y pequeñas separadas. Nótese que es la combinación de EF-G/RRF la que causa disociación; EF-G solo juega un papel diferente en el movimiento de los ribosomas cuando no está en el codón de parada.


