11.1: Escisión proteolítica
- Page ID
- 53066
La modificación más común es la escisión proteolítica. Algunos de los polipéptidos de pre-escisión se escinden inmediatamente, mientras que otros se almacenan como precursores inactivos para formar un conjunto de enzimas (u otro tipo de proteínas) que se pueden activar muy rápidamente, en una escala de tiempo de segundos a minutos, en comparación con tener que pasar por transcripción y traducción, o incluso solo traducción. Curiosamente, aunque la metionina (Met) es universalmente el primer aminoácido de un polipéptido recién sintetizado, muchas proteínas tienen que la metionina se escinde (también es cierto para algunos F-met procariotas).
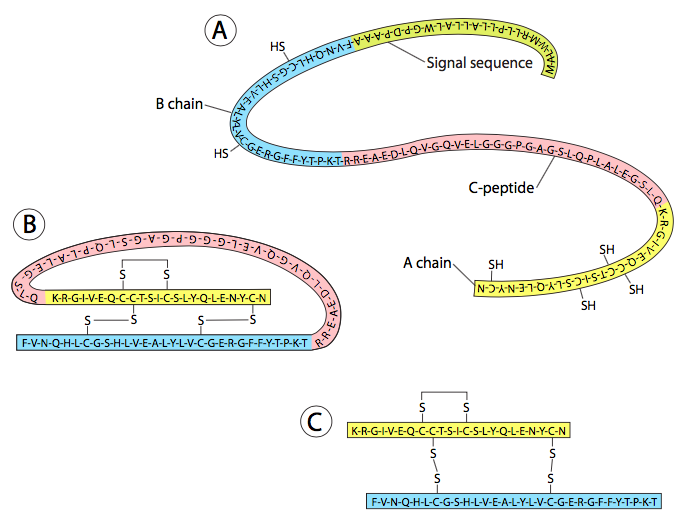
La activación de proteínas por escisión de precursores es un tema común: la proteína precursora se denomina proproteína, y el péptido que se escinde de ella para activar la proteína se llama propéptido. Entre los ejemplos más conocidos de proteínas que se derivan de proproteínas se encuentran la hormona insulina, la familia de caspasas de proteínas de muerte celular y la proteína neural asociada a Alzheimer β-amiloide. La insulina es un ejemplo interesante (Figura\(\PageIndex{1}\)) en mamíferos: la preproinsulina (inactiva como hormona) se traduce primero a partir del ARNm de insulina. Después de una escisión que elimina una secuencia N-terminal, se genera proinsulina (aún inactiva). La proinsulina forma algunos enlaces disulfuro internos, y cuando se produce la acción proteolítica final, se extrae una porción sustancial (llamada péptido C) del centro de la proinsulina. Sin embargo, dado que la proteína estaba unida internamente por disulfuro, las dos piezas terminales permanecen conectadas para convertirse en la hormona insulina activa.
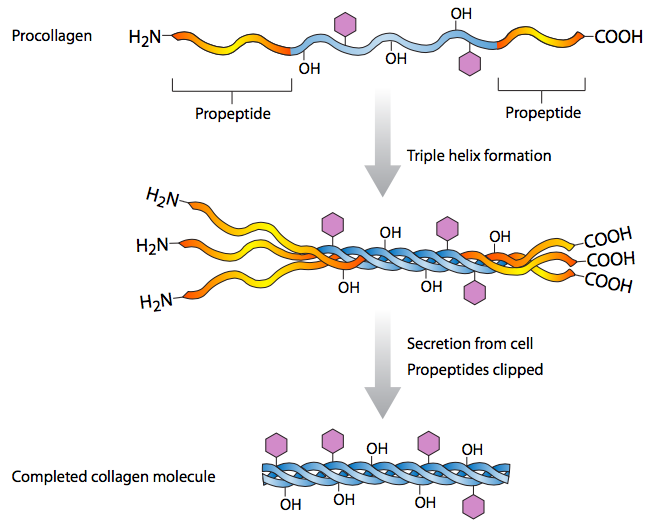
Otro ejemplo interesante de procesamiento de proteínas es el del ensamblaje de colágeno (Figura\(\PageIndex{2}\)). Como leerás en el capítulo 13, el colágeno es una proteína secretada muy grande que proporciona estructura y absorbancia de choque para la matriz extracelular en animales. La puedes encontrar en piel, pezuñas, cartílagos y diversos tejidos conectivos. Una proteína de colágeno individual es en realidad una triple hélice retorcida de tres subunidades. Las subunidades de colágeno se hacen como procolágeno, y los propéptidos se eliminan de ambos extremos N y C para generar la proteína final. Sin embargo, no se escinden hasta después de que las tres subunidades se ensamblen una alrededor de la otra. De hecho, las subunidades de colágeno que ya han sido procesadas no se ensamblan en proteínas de triple hélice. Las secuencias propeptídicas son claramente necesarias para el ensamblaje eficiente del complejo proteico final.