11.2: Tráfico de Proteínas
- Page ID
- 53084
La idea de que las secuencias de propéptidos tienen funciones importantes en la maduración de proteínas más allá de solo evitar que sean activas no es exclusiva del ensamblaje. Una clase principal de secuencias peptídicas escindidas son los péptidos señal. Los péptidos señal dirigen la proteína desde el citoplasma hacia un compartimento celular particular. En el caso de los procariotas, esto significa esencialmente la membrana celular, pero para los eucariotas, existen péptidos señal específicos que pueden dirigir la proteína al núcleo, a las mitocondrias, al retículo endoplásmico, y otros orgánulos intracelulares. Los péptidos son específicamente reconocidos por receptores en las membranas de compartimentos particulares, que luego ayudan a guiar la inserción de la proteína dentro o a través de la membrana. Casi toda la síntesis de proteínas en eucariotas se lleva a cabo en el citoplasma (con la excepción de algunas proteínas en los cloroplastos y mitocondrias), por lo que las proteínas que se encuentran en cualquier otro compartimento o incrustadas en cualquier membrana deben haber sido dirigidas y transportadas a ese compartimento por su secuencia señal.
Aunque esto se considera principalmente un proceso eucariota dado que hay tantas dianas potenciales, los procariotas sí tienen proteínas de membrana (de hecho, unas 800 diferentes en E. coli que comprenden ~ 20% de la proteína total), y se posicionan allí con la ayuda de enzimas insertasa como YidC y complejos como la translocasa Sec. La translocasa Sec utiliza una partícula de reconocimiento de señal (SRP) muy parecida a la de los eucariotas, y se discutirá más adelante en este capítulo cuando se introduzca la SRP. YidC, que tiene homólogos eucariotas (por ejemplo Oxa1 en mitocondrias), es una proteína transmembrana de 61 kDa que se coloca en la membrana a través de un mecanismo de translocasa SRP-sec. Una vez allí, YidC interactúa con polipéptidos nacientes (una vez que alcanzan ~70 aminoácidos de largo) que han comenzado a interactuar con los lípidos de la membrana celular, y empuja la proteína hacia/a través de la membrana.
El núcleo es uno de esos compartimentos, y los ejemplos de las proteínas que se encuentran dentro incluyen ADN y ARN polimerasas, factores de transcripción e histonas. Estas y otras proteínas nucleares tienen una secuencia señal N-terminal conocida como NLS, o señal de localización nuclear. Esta es una vía bien estudiada que involucra un conjunto de proteínas adaptadoras de importina y el complejo de poro nuclear (Figura\(\PageIndex{3}\)). El transporte al núcleo es particularmente desafiante porque tiene una doble membrana (recuerde que es contigua a la membrana del retículo endoplásmico. Si bien existen otros mecanismos para elaborar proteínas que están incrustadas en la membrana nuclear, el mecanismo principal para la importación y exportación de moléculas grandes dentro y fuera del núcleo mismo es el complejo de poros nucleares. El complejo es muy grande y puede estar compuesto por más de 50 proteínas diferentes (nucleoporinas, a veces llamadas nups). Las nucleoporinas se ensamblan en un gran poro octagonal abierto a través de las membranas nucleares. Como\(\PageIndex{3}\) indica la Figura, hay fibrillas similares a antenas en la cara citoplásmica, y estas ayudan a guiar las proteínas desde su origen en el citoplasma hasta el poro nuclear, y en el lado nuclear hay una estructura de canasta. Por supuesto, no todas las proteínas están permitidas en el núcleo, y el mecanismo para distinguir dianas apropiadas es sencillo. La proteína debe llevar una señal de localización nuclear (NLS). Mientras que en el citoplasma, una proteína importina-α se une a la NLS de una proteína nuclear, y también se une a una importina-β. La importina-β es reconocida y unida por el complejo de poro nuclear. Los detalles del mecanismo de transporte son turbios, pero se cree que las repeticiones de fenilalanina-glicina en las subunidades de nucleoporina (Fg-NUPs) están involucradas.
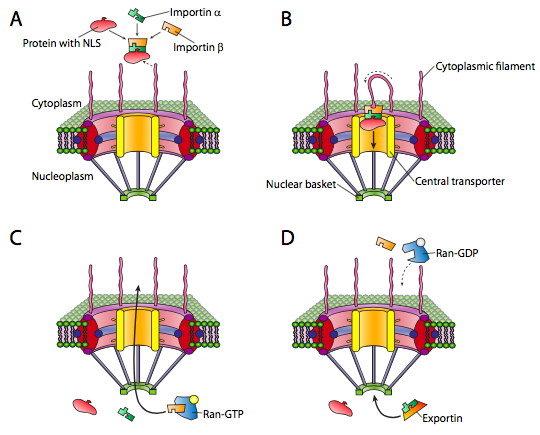
Una vez que el agregado de nucleoproteína-importina se mueve hacia el núcleo, RAN-GTP, una GTPasa pequeña, hace que el agregado se disocie (Figura\(\PageIndex{3}\) c). La proteína importada se libera en el núcleo. Las importinas también se liberan en el núcleo, pero se exportan de nuevo hacia afuera para ser reutilizadas con otra proteína dirigida al núcleo.
Los mecanismos de activación de GTPasa pequeña de otros procesos serán discutidos nuevamente con más detalle en capítulos posteriores (cytoskel-eton, señalización). La clave para entender el mecanismo es recordar que la GTPasa hidroliza el GTP al PIB, pero aún se mantiene en el PIB. Aunque la GTPasa hidrolizará GTP espontáneamente, la proteína activadora de GTPasa, GAP (o RAN-GAP en este caso) acelera enormemente la velocidad de hidrólisis. Para volver a ciclar el sistema a GTP, el PIB no se vuelve a fosforilar: se intercambia por un nuevo GTP. El intercambio se ve facilitado en gran medida por la acción de una proteína accesoria, el factor de intercambio de nucleótidos guanina (GEF), en este caso particular, un RAN-GEF.
La exportación del núcleo al citoplasma también ocurre a través del poro nuclear. El ran-GTP también forma parte del complejo de exportación (Figura\(\PageIndex{3}\) d), y en conjunto con una proteína exportina y lo que se va a exportar, se mueve fuera del núcleo a través del poro nuclear. Una vez en el citoplasma, la hidrólisis de GTP a GDP por Ran (activado por RAN-GAP, una proteína citoplásmica) proporciona la energía para disociar la carga (por ejemplo, ARNm) de las moléculas transportadoras exportadoras. El Ran-PIB luego se une a las importinas, vuelve a ingresar al núcleo y el PIB se intercambia por GTP.
El poro nuclear es el único complejo de transporte que abarca capas de membrana dual, aunque existen pares coordinados de complejos de transporte en orgánulos de doble membrana como las mitocondrias. Las proteínas de transporte en la membrana mitocondrial externa se unen con las proteínas de transporte en la membrana mitocondrial interna para mover las proteínas unidas a la matriz (por ejemplo, las involucradas en el ciclo de TCA) desde el citoplasma. Los complejos que mueven las proteínas a través de la membrana externa están formados por la familia de proteínas Tom (translocador de membrana externa). Algunas de las proteínas permanecerán incrustadas en la membrana externa: son procesadas por un complejo SAM (maquinaria de clasificación y ensamblaje) también incrustado en la membrana externa). Mientras tanto, otros continúan con las proteínas Tim (translocador de membrana interna) que las mueven a través de la membrana interna. Al igual que con las proteínas nucleares, existe una secuencia señal consenso sobre las proteínas mitocondriales que está unida por chaperonas citosólicas que las llevan a los transportadores Tom. Como se muestra en la siguiente tabla, existen secuencias/propéptidos señal que dirigen proteínas a varios otros compartimentos.
De particular importancia para el resto de este capítulo, es la secuencia que dirige las proteínas al retículo endoplásmico, y por extensión, cualquier proteína destinada al RE, el aparato de Golgi, la membrana celular, vesículas y compartimentos derivados vesicularmente, y secreción fuera de la célula. Aquí, además de una secuencia señal N-terminal, la posición de las secuencias de señal internas secundarias (a veces llamadas parches de señal) ayuda a determinar la disposición de la proteína a medida que ingresa al ER.
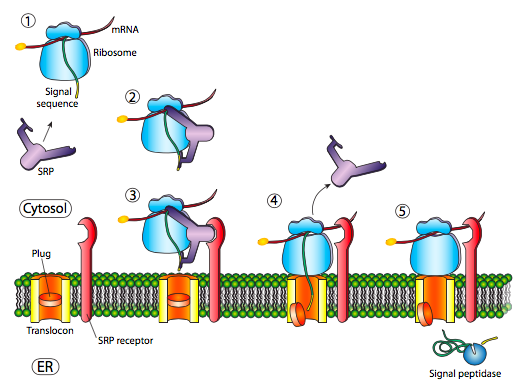
La inserción inicial requiere el reconocimiento de la secuencia señal por SRP, la proteína de reconocimiento de señal. La SRP es una proteína G e intercambia su GDP unido por un GTP al unirse a la secuencia señal de una proteína. La SRP con su proteína unida luego se acopla a un receptor (llamado receptor SRP, asombrosamente) incrustado en la membrana del ER y que se extiende hacia el citoplasma. La SRP generalmente se une tan pronto como la secuencia señal está disponible, y cuando lo hace, detiene la traducción hasta que se acopla a la membrana del ER. Por cierto, este es el origen del retículo endoplásmico “rugoso”: los ribosomas que se encuentran en el RE están unidos a la superficie citoplásmica del RE por el polipéptido naciente que está produciendo y una SRP. El receptor SRP puede existir por sí mismo o en asociación con un translocon, que es un canal de translocación bipartito. El receptor SRP (SR) también es una GTPasa, y suele portar una molécula GDP cuando no está asociado. Sin embargo, al asociarse con el translocon intercambia su PIB por un GTP. Estas GTP son importantes porque cuando la SRP se une a la SR, ambas actividades de GTPasa se activan y la liberación resultante de energía se disocia tanto del translocón como del polipéptido naciente. Esto alivia el bloqueo de la traducción impuesto por la SRP, y la nueva proteína es empujada a través del translocon a medida que se sintetiza. Una vez que la secuencia señal ha entrado completamente en el lumen del ER, revela un sitio de reconocimiento para la peptidasa señal, una enzima hidrolítica que reside en el lumen del ER y cuyo propósito es cortar el péptido señal.
Los procariotas también usan un homólogo SRP. En E. coli, la SRP es simple, compuesta por una subunidad proteica (Ffh) y un ARN pequeño de 4.5S. En comparación, algunos eucariotas superiores tienen una SRP compuesta por seis subunidades de proteínas diferentes y un ARN 7S. De manera similar, existe un homólogo procariota simple para el receptor SRP, ftsY. Una diferencia interesante es que FtsY generalmente no interactúa con las proteínas exportadas, y parece ser necesario solo para las proteínas incrustadas en la membrana. De lo contrario, existen muchas similitudes en el mecanismo para la inserción basada en SRP de proteínas de membrana en especies eucariotas y procariotas, incluyendo la dependencia de GTP, y la finalización del mecanismo por una translocasa (SecyEG en E. coli).
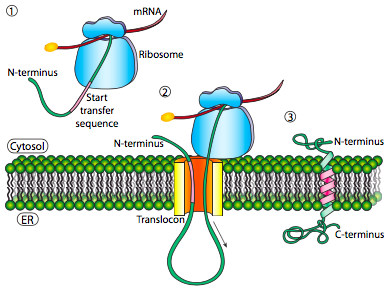
Si esa era la única secuencia señal en la proteína, el resto de la proteína se sintetiza y se empuja a través del translocon y se deposita una proteína soluble en el lumen del RE, como se muestra en la Figura\(\PageIndex{4}\). ¿Qué pasa con las proteínas que están incrustadas en una membrana? Las proteínas transmembrana tienen secuencias de señal internas (a veces llamadas parches de señal). Dependiendo de sus localizaciones relativas, pueden considerarse secuencia de inicio-transferencia o parada de transferencia, donde “transferencia” se refiere a la translocación del péptido a través del translocón. Esto es más fácil de entender haciendo referencia a la Figura\(\PageIndex{5}\).
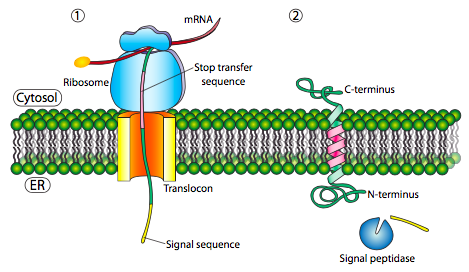
Si hay un tramo significativo de residuos hidrófobos en su mayoría ininterrumpidos, se consideraría una señal de detención-transferencia, ya que esa parte de la proteína puede quedar atrapada en el translocón (y posteriormente en la membrana del RE) obligando al resto de la proteína a permanecer fuera del RE. Esto generaría una proteína que se inserta en la membrana una vez, con su extremo N en la luz del RE y el extremo C en el citoplasma. En una proteína transmembrana multipaso, podría haber varios parches de señal hidrofóbica de inicio y parada de transferencia.
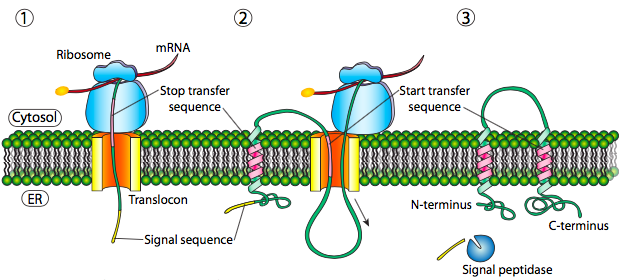
Basándose en el ejemplo de un solo paso, si hubiera otro parche de señal después de la secuencia de detención-transferencia, actuaría como una secuencia de inicio-transferencia, uniéndose a un translocon y permitiendo que el resto de la proteína se trasladara al ER. Esto da como resultado una proteína con ambos extremos N- y C- en el lumen del ER, pasando a través de la membrana del ER dos veces, y con un asa citoplasmática sobresaliendo. Por supuesto, el N-terminal podría estar del otro lado. Para un N-terminal citoplásmico, la proteína no puede tener una secuencia señal N-terminal (Figura\(\PageIndex{7}\)). En su lugar, tiene un parche de señal interna. Desempeña esencialmente el mismo papel, pero la orientación del parche significa que el N-terminal permanece citoplásmico. El polipéptido traducido después del parche se alimenta al ER. Y así como en el último ejemplo, múltiples secuencias de parada e inicio pueden reinsertar la proteína en la membrana y cambiar el revestimiento de la siguiente porción.