16.4: Oncogenes
- Page ID
- 53214
Los oncogenes son generalmente mutaciones dominantes de ganancia de función de genes celulares normales llamados protooncogenes. Estos protooncogenes son en sí mismos reguladores positivos del ciclo celular, pero están regulados por otros factores, ya sean señales extracelulares o mecanismos intracelulares. Las mutaciones que los convierten en oncogenes eliminan específicamente toda o parte de esta regulación. De este modo, se vuelven hiperactivos e intentan impulsar el ciclo celular hacia adelante, lo que lleva a una mayor proliferación. Estas mutaciones también pueden clasificarse en algunas categorías mecanicistas generales.
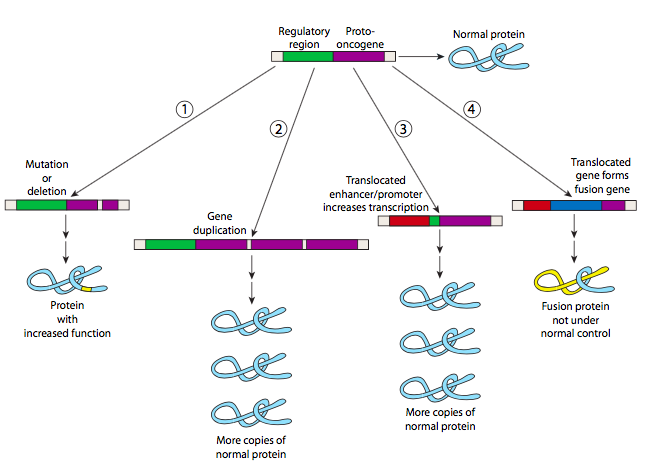
Estas (Figura\(\PageIndex{12}\)) son mutaciones a la región codificadora que incrementan la actividad fisiológica, duplicaciones génicas dando como resultado más copias del gen a nivel de ADN y por lo tanto más a nivel de proteína, mutaciones a la región reguladora del gen o que alteran la regulación de la expresión génica, así aumentar el número de copias de la proteína, y finalmente, translocaciones que reemplazan parte de la región codificante, dando como resultado una proteína quimérica cuya actividad puede estar bajo un esquema de control diferente al normal.
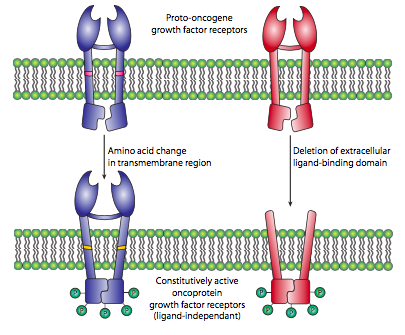
Ejemplos de dos tipos de mutaciones se ilustran en la Figura\(\PageIndex{13}\) con un receptor mitógeno como protooncogén. En el primer caso, la porción transmembrana del receptor ha sido mutada, provocando un cambio de aminoácidos que altera la conformación no solo de la región transmembrana, sino del dominio cinasa citoplásmico, que se vuelve constitutivamente activo, independientemente de que un ligando se haya unido fuera o no. En el segundo caso, se ha eliminado todo el dominio extracelular debido a una mutación de un codón de aminoácido en un codón de terminación o translocación, y el receptor resultante siempre está activo, también independiente de la unión al ligando.
Algunos tipos de infección retroviral pueden lograr la conversión de un protooncogén en un oncogén insertando ADN viral cerca de la región promotora del protooncogén. Debido a que los promotores virales tienden a ser muy fuertes, pueden inducir la sobreexpresión del producto protooncogénico. En especies aviares, se sabe que el virus de la leucosis aviar causa tumores por inserción cerca del oncogén c-myc, mientras que en humanos, otro retrovirus, el HTLV (virus linfotrópico T humano), puede causar enfermedad aguda (paraparesia espástica tropical), pero también puede causar leucemia de células T y linfoma.
¿Qué funciones son características de los protooncogenes? Receptores mitógenos, como ya se describió anteriormente, y ejemplificados por el receptor tirosina quinasas EGFR (receptor del factor de crecimiento epidérmico), VEGFR (receptor del factor de crecimiento endotelial vascular), RON (receptor de origen nantais, un receptor de proteína estimulante de macrófagos) y ErbB2 (también HER2/neu, otro EGF humano receptor). Los factores de crecimiento en sí mismos también pueden ser protooncogenes, como FGF-5, uno de varios oncogenes en la familia de factores de crecimiento de broblastos, o c-sis, una forma oncogénica de PDGF (factor de crecimiento derivado de plaquetas). Las proteínas de cascada de señal, a menudo tirosina o serina/treonina quinasas u otras enzimas reguladoras, son un gran grupo de protooncogenes (por ejemplo, tirosina quinasas de la familia Src, tirosina quinasas de la familia BTK, Ser/Thr quinasas dependientes de ciclina, GTPasas pequeñas de la familia Ras). Finalmente, diversos factores de transcripción (por ejemplo, familias Ets, Myc, E2F), pueden mutarse eficazmente en oncogenes.


