10.8: Temas avanzados, Resumen y puntos clave, Lectura adicional, Bibliografía
- Page ID
- 54954
Aún quedan muchos otros problemas que deben resolverse estudiando la estructura del ARN. En esta sección se perfilarán algunos de ellos.
Otros problemas
Observe algunos de los problemas que se muestran gráficamente a continuación:
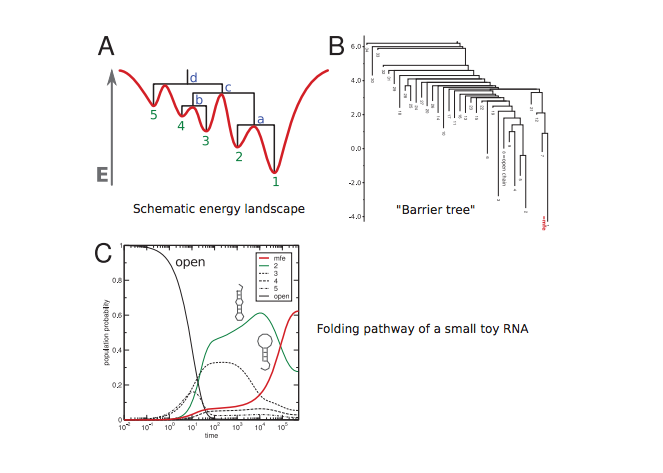
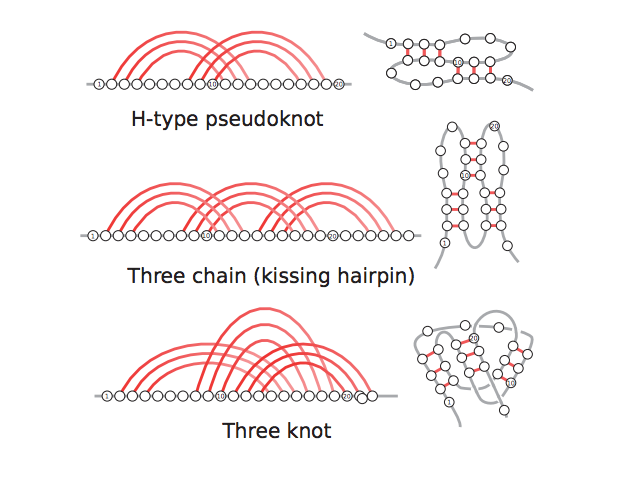
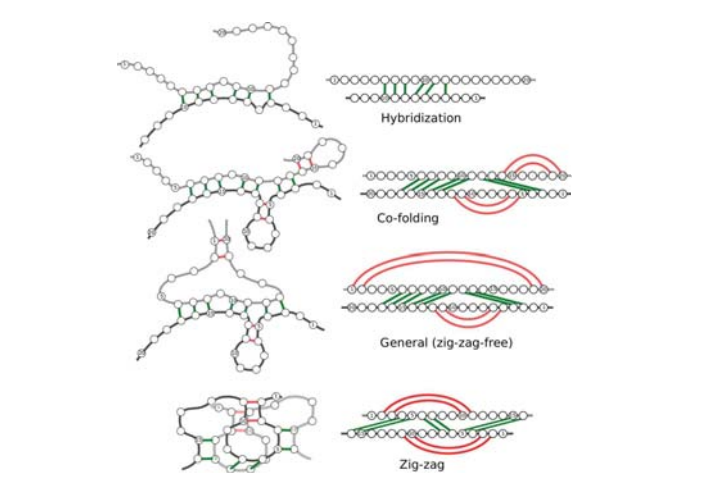
Relevancia
Hay muchos ARN dentro de la célula aparte de los mRNAs, ARNt y ARNr. La pregunta es ¿cuál es la relevancia de todo este ARN no codificante? Algunos creen que es ruido resultado a través del experimento, algunos piensan que es solo ruido biológico que no tiene sentido en el organismo vivo. Por otro lado algunos creen que el ARN basura en realidad podría tener un papel importante como señales dentro de la célula y todo es realmente funcional, la verdad probablemente se encuentra en algún punto intermedio.
Investigación actual
Hay regiones conservadas en el genoma que no codifican ninguna proteína, y ahora Stefan et al. las están investigando para ver si tienen estructuras lo suficientemente estables para formar ARN funcionales. Resulta que alrededor del 6% de estas regiones tienen señas de identidad de buena estructura de ARN, que sigue siendo 30000 elementos estructurales. El grupo ha anotado algunos de estos elementos, pero aún queda un largo camino por recorrer. Se han encontrado muchos miARN, snoWRNAs y por supuesto muchos falsos positivos. ¡Pero hay resultados emocionantes que surgen en este tema! así que la nota final es, ¡es una muy buena zona para trabajar!
Resumen y puntos clave
1. El espectro funcional de los ARN es prácticamente ilimitado
(a) Los ARN similares a las Ribozimas y Riboswitches contemporáneos podrían haber existido en un mundo de ARN. Algunos de ellos aún existen como fósiles vivos en las células actuales.
(b) Los ARN evolutivamente más jóvenes, incluidos los miARN y muchos ARNc largos, forman una capa reguladora no basada en proteínas.
- La estructura del ARN es crítica para su función y se puede predecir computacionalmente
(a) Nussinov/Zuker: Estructura mínima de energía libre (b) McCaskill: Función de partición y probabilidades de pares
(c) CYK/Interide-Outside: solución probabilística al problema usando SCFG
- La información filogenética puede mejorar la predicción de estructuras
- La biología computacional de los ARN es un campo activo de investigación con muchos problemas algorítmicos duros aún abiertos
10.10 Lectura adicional
• Visión general
— Washietl S, Will S. et al. Análisis computacional de ARN no codificantes. Wiley Interdiscip Rev RNA. 2012, 10.1002/wrna.1134
• Función RNA: artículos de revisión de John Mattick
• Plegamiento de ARN de secuencia única
— Nussinov R, Jacobson AB, algoritmo rápido para predecir la estructura secundaria del ARN monocatenario.Proc Natl Acad Sci U S A. 1980 Nov; 77: (11) 6309-13
— Zuker M, Stiegler P Plegamiento óptimo por computadora de grandes secuencias de ARN utilizando termodinámica e información auxiliar. Nucleic Acids Res. 1981 Ene; 9: (1) 133-48
— McCaskill JS La función de partición de equilibrio y las probabilidades de unión de pares de bases para la estructura secundaria de ARN. Biopolímeros. 1990; 29: (6-7) 1105-19
— Dowell RD, Eddy SR, Evaluación de varias gramáticas estocásticas ligeras sin contexto para la predicción de estructura secundaria de ARN. BMC Bioinformática. 2004 jun; 5:71
— Do CB, Woods DA, Batzoglou S, ContraFold: predicción de estructura secundaria de ARN sin modelos basados en la física. Bioinformática. 2006 Jul; 22: (14) e90-8
• Plegamiento de ARN consenso
— Hofaker IL, Fekete M, Stadler PF, Predicción de estructura secundaria para secuencias de ARN alineadas. J Mol Biol. 2002 jun; 319: (5) 1059-66
— Knudsen B, Hein J, predicción de estructura secundaria de ARN usando gramáticas estocásticas libres de contexto e historia evolutiva. Bioinformática. 1999 Jun; 15: (6) 446-54
• Búsqueda de genes de ARN
— Pedersen JS, Bejerano G, Siepel A, Rosenbloom K, Lindblad-Toh K, Lander ES, Kent J, Miller W, Haussler D Identificación y clasificación de estructuras secundarias de ARN conservadas en el genoma humano. PLoS Comput Biol. 2006 Abr; 2: (4) e33
— Washietl S, Hofaker IL, Stadler PF, Predicción rápida y confiable de ARN no codificantes. Proc Natl Acad Sci U S A. 2005 Feb; 102: (7) 2454-9
Bibliografía
- [1] R Durbin. Análisis de Secuencia Biológica.
- [2] W. Gilbert.” origen de la vida: El mundo del rna”. Naturaleza., 319 (6055) :618, 1986.
- [3] Rachel Sealfon, 2012. Información extra extraída de la recitación 5 diapositivas.
- [4] Z. Wang, M. Gestein y M. Snyder. RNA-seq: una herramienta revolucionaria para la transcriptómica. Nat Rev Genet., 10 (1) :57—63, 2009.
- [5] Stefan Washietl, 2012. Todas las imágenes/fórmulas cortesía de las diapositivas de Stefan.
- [6] R. Tejedor. Biología Molecular. 3ª edición.


