13.2: Interferencia de ARN
- Page ID
- 54521
La interferencia de ARN ha sido uno de los descubrimientos más significativos y emocionantes de la historia reciente. El impacto de este descubrimiento es enorme con aplicaciones que van desde estudios de derribo y pérdida de función hasta la generación de mejores modelos animales con caída condicional de gen (s) deseado (s) a gran escala, cribas basadas en ARNi para ayudar al descubrimiento de fármacos.
Historia del descubrimiento
El descubrimiento del fenómeno de silenciamiento génico se remonta a la década de 1990 con Napoli y Jorgensen demostrando la regulación a la baja de la chalcona sintasa tras la introducción del transgén exógeno en plantas [17]. Posteriormente se observó una supresión similar en otros sistemas [10, 22]. En otro conjunto de trabajos no relacionados en ese momento, Lee et al. identificaron en una pantalla genética que lin-4 endógena expresaba un producto no codificante de proteínas que es complementario al gen lin-14 y controlaban el momento del desarrollo larval (del primer al segundo estado larval) en C. elegans [15]. Ahora lo conocemos como el primer miARN que se descubre. En 2000, se descubrió otro miARN, let-7, en el mismo organismo y se encontró que estaba involucrado en la promoción de la transición larval tardía a adulta [21]. El trabajo seminal de Mello y Fire en 1998 (por el cual fue galardonado con el Premio Nobel en 2006) demostró que la introducción del ARNbc exógeno en C. elegans silenció específicamente genes mediante interferencia de ARN, explicando el fenómeno de supresión previa observado en plantas [7]. Estudios posteriores encontraron la conversión de ARNbc en ARNip en la vía de ARNi. En 2001, el término miARN y el vínculo entre miARN y ARNi se describió en tres artículos en Science [23]. Con esto, nos hemos dado cuenta de que la maquinaria reguladora génica estaba compuesta predominantemente por dos clases de ARN pequeños, con miARN involucrado en la regulación de genes endógenos y ARNip involucrado en defensa en respuesta a ácidos nucleicos virales, transposones y transgenes [5]. Trabajos posteriores revelaron efectores aguas abajo: Dicers (para la escisión de especies precursoras) y proteínas Argonaute (parte del complejo silenciador inducido por ARN para realizar los efectos silenciadores reales), completando nuestra comprensión actual de las vías de silenciamiento del ARN. Los detalles del mecanismo y las diferencias entre las especies se discuten más adelante.
Vías de biogénesis
Hay un tema común involucrado tanto para el silenciamiento mediado por ARNip como para el miARN. En la biogénesis tanto de ARNip como de miARN, los precursores bicatenarios se escinden por una RNasa en fragmentos cortos de ∼22 nt. Una de las cadenas (la cadena guía) se carga en una proteína Argonaute, un componente central del complejo de ribonucleoprotien más grande RISC que facilita el reconocimiento y silenciamiento del ARN diana. El mecanismo de silenciamiento es la escisión del ARNm diana o la represión de la traducción.
Aparte de este tema común, las proteínas involucradas en estos procesos difieren entre especies y existen etapas adicionales en el procesamiento del miARN previo a su maduración e incorporación al RISC (Figura 13.1). Para la biogénesis del ARNip, los precursores son los dsRNAs, muchas veces de fuentes exógenas como virus o transposones. Sin embargo, estudios recientes también han encontrado ARNip endógenos [9]. Independientemente de la fuente, estos ARNbc son procesados por la endonucleasa RNasa III, Dicer, en ARNsi de ∼22 nt. Esta escisión catalizada por RNasa III deja los 5'fosfatos característicos y los voladizos 3' de 2 nt [2]. Cabe destacar que diferentes especies han evolucionado con diferente número de parálogos. Esto se vuelve importante ya que, para ser discutido más adelante, la vía de biogénesis de miARN también utiliza Dicer para el procesamiento de precursores de miARN (más específicamente pre-miARN). Para especies como D. melanogaster, hay dos proteínas Dicer distintas y como resultado normalmente hay un procesamiento preferencial de los precursores (por ejemplo, Dicer-1 para escisión de miARN y Dicer-2 para escisión de ARNip) [5]. En contraste, los mamíferos y nematodos solo tienen una única proteína Dicer y como tal ambas vías de biogénesis convergen a la misma etapa de procesamiento [5]. En etapas posteriores de la ruta de biogénesis de ARNip, una de las cadenas en el dúplex de ARNip se carga en RISC para silenciar los ARN diana (Figura 13.1C).
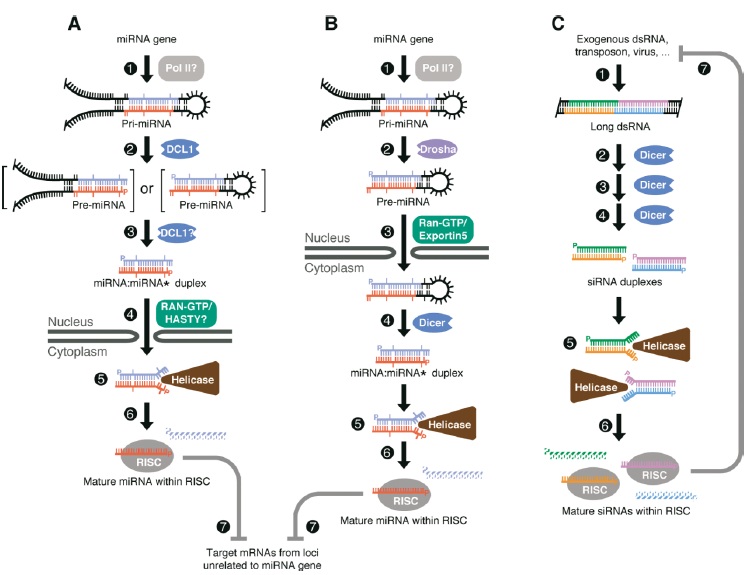
Figura 13.1: Vías de biogénesis de ARNip y miARN. (A) Biogénesis de miARN vegetal (B) Biogénesis de miARN animal (C) Biogénesis de ARNip animal. Adoptada de Bartel, 2004 (ref [2]). Copyright © 2004 Cell Press.
En la vía de biogénesis de miARN, la mayoría de los precursores son transcritos pol II de las regiones intrónicas, algunas de las cuales codifican múltiples miARN en grupos. Estos precursores, en forma de estructura tallo-bucle, se llaman pri-miARN. Los pri-miRNAs se escinden primero en el núcleo por una endonucleasa RNasa III (Drosha en animales y Dcl1 en plantas) en intermedios de tallo bucle de ∼60-70 nt, denominados pre-miRNAs [2]. En los animales, el pre-miARN es luego exportado al citoplasma por Exportin-5. Esto es seguido por la escisión del intermedio pre-miARN por Dicer para eliminar el tallo-asa. Una de las cadenas en el dúplex de miARN maduro resultante se carga en RISC, similar a la descrita para la biogénesis de ARNip Figura 13.1B. Curiosamente, en las plantas, el pri-miARN se procesa en miARN maduro a través de dos escisiones por la misma enzima, Dcl1, en el núcleo antes de su exportación al citoplasma para su carga (Figura 13.1A).
Funciones y mecanismo de silenciamiento
La visión clásica de la función de miARN basada en los primeros descubrimientos de miARN ha sido análoga a un cambio binario mediante el cual miARN reprime la traducción de algunas dianas de ARNm clave para iniciar una transición de desarrollo. Sin embargo, estudios posteriores han ampliado enormemente esta definición. En las plantas, la mayoría de los miARN se unen a la región codificante del ARNm con complementariedad casi perfecta. Por otro lado, los miARN animales se unen con complementariedad parcial (excepto por una región semilla, residuos 2-8) a las regiones 3' UTR del ARNm. Como tal, hay potencialmente cientos de dianas por un solo miARN en animales en lugar de solo unos pocos [1]. Además, en los mamíferos, solo una parte de las dianas predichas están involucradas en el desarrollo, y el resto se predice que cubrirá una amplia gama de procesos moleculares y biológicos [2]. Por último, el silenciamiento de miARN actúa a través de la represión de la traducción y la escisión del ARNm (y también la desestabilización como se analiza a continuación) (como se muestra por ejemplo por Bartel y sus compañeros de trabajo en la escisión dirigida por miR-96 de HOXB6 [26]). En conjunto, la visión moderna de la función de miARN ha sido que el miARN amortigua la expresión de muchas dianas de ARNm para optimizar la expresión, reforzar la identidad celular y agudizar las transiciones.
El mecanismo para el cual miARN media el silenciamiento del ARNm diana sigue siendo un área de investigación activa. Como se discutió anteriormente, el silenciamiento del ARN puede tomar la forma de escisión, desestabilización (que conduce a la posterior degradación del ARNm) o represión de la traducción. En las plantas, se ha encontrado que el modo predominante de silenciamiento del ARN es a través de la escisión catalizada por Argonautas. Sin embargo, la contribución de estos diferentes modos de silenciamiento ha sido menos clara en los animales. Análisis globales recientes del grupo Bartel en colaboración con Gygi e Ingolia y Weissman arrojan luz sobre esta cuestión. En un estudio de 2008, los grupos Bartel y Gygi examinaron los cambios globales en el nivel de proteínas mediante espectrometría de masas después de la introducción o deleción de miARN [1]. Sus resultados revelaron la represión de cientos de genes por miARN individuales y, lo que es más importante, la desestabilización del ARNm representa la mayoría de las dianas altamente reprimidas (Figura 13.2).
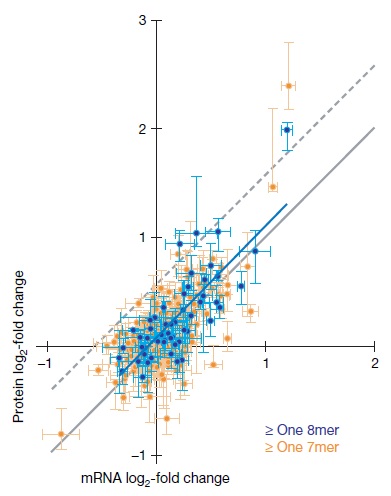
Figura 13.2: Cambios en proteínas y ARNm tras la pérdida de miR-223, a partir de mensajes con al menos un sitio 3'UTR de 8 meros (azul) o al menos un 7-mero (naranja). Adoptada de Baek et al., 2008 (ref [1]). Copyright © 2008 Macmillan Publishers Limited.
Esto está respaldado además por un estudio posterior utilizando tanto RNA-seq como un nuevo perfil de ribosomas demostrado por primera vez por Inoglia y Weissman 2009 que permite interrogar las actividades de traducción global con resolución de subcodones [14]. Los resultados mostraron que la desestabilización del ARNm diana es el mecanismo predominante a través del cual el miARN reduce la producción de proteínas.


