25.2: Direcciones actuales de investigación
- Page ID
- 54437
This page is a draft and is under active development.
La funcionalidad de codificación en el ADN es una forma en que los biólogos sintéticos programan células A medida que el precio de la secuenciación y síntesis del ADN sigue disminuyendo, la codificación de cadenas de ADN se ha vuelto más factible. De hecho, el número de pares de bases que se pueden sintetizar por US$ ha aumentado exponencialmente, similar a la Ley de Moore.
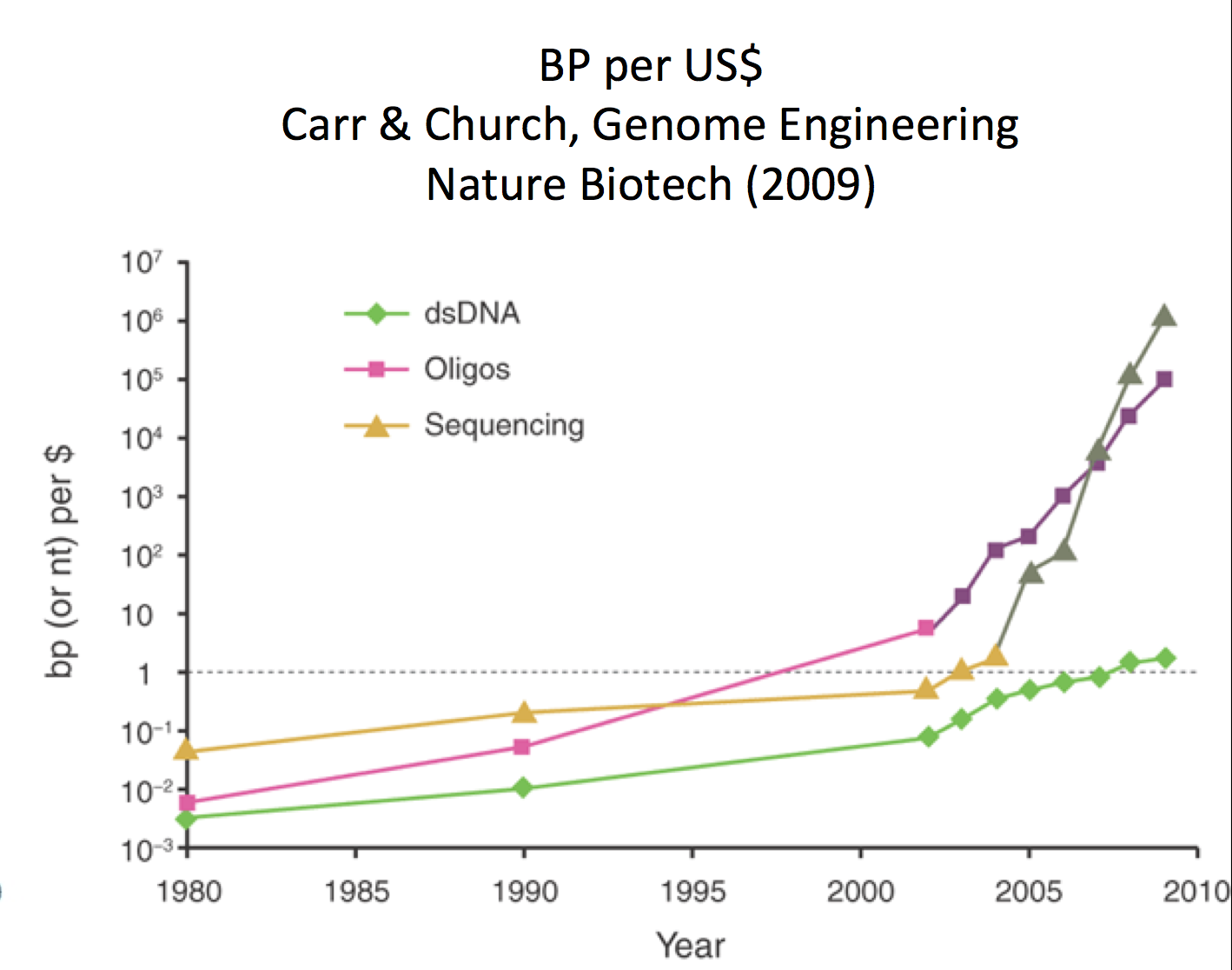
Fuente: Carr, Peter A. y George M. Church. “Ingeniería del Genoma”.
Nature Biotechnology 27, núm. 12 (2009): 1151-62
Figura 26.4: Costo de sintetizar un par base versus dólar estadounidense
Esto ha hecho que el proceso de diseñar, construir y probar circuitos biológicos sea mucho más rápido y económico. Una de las principales áreas de investigación en biología sintética es la creación de síntesis rápida y automatizada de moléculas de ADN y la creación de células con la secuencia de ADN deseada. El objetivo de crear un sistema de este tipo es acelerar el diseño y depuración de hacer un sistema biológico para que los sistemas biológicos sintéticos puedan ser prototipados y probados en un proceso rápido e iterativo.
La biología sintética también tiene como objetivo desarrollar componentes biológicos abstractos que tengan un comportamiento estándar y bien definido como una pieza que un ingeniero eléctrico podría ordenar de un catálogo. Para ello, en 2003 se creó el Registro de Partes Biológicas Estándar (partsregistry.org) [4] y actualmente contiene más de 7000 piezas disponibles para los usuarios. La parte de investigación de la creación de dicho registro incluye la clasificación y descripción de las partes biológicas. El objetivo es encontrar piezas que tengan características deseables como:
Los reguladores de ortogonalidad no deben interferir entre sí. Deben ser independientes.
Composabilidad Los reguladores se pueden fusionar para dar función compuesta.
Los reguladores de conectividad se pueden encadenar para permitir cascadas y retroalimentación.
Homogeneidad Los reguladores deben obedecer una física muy similar. Esto permite previsibilidad y eficiencia.
La biología sintética aún se está desarrollando, y la investigación aún puede ser realizada por personas con pocos antecedentes en el campo. La Fundación Internacional de Máquinas Genéticamente Modificadas (iGEM) (igem.org) [3] creó el concurso iGEM donde estudiantes de pregrado y preparatoria compiten para diseñar y construir sistemas biológicos que operan dentro de células vivas. A los equipos estudiantiles se les entrega un kit de partes biológicas a principios del verano y trabajan en sus propias instituciones para crear un sistema biológico. Algunos proyectos interesantes incluyen:
Biodetector de arsénico El objetivo fue desarrollar un biosensor bacteriano que responda a un rango de concentraciones de arsénico y produzca un cambio en el pH que pueda calibrarse en relación con la concentración de arsénico. El objetivo del equipo era ayudar a muchos países subdesarrollados, en particular Bangladesh, a detectar la contaminación por arsénico en el agua. El dispositivo propuesto se pretendía ser más económico, portátil y más fácil de usar en comparación con otros detectores.
BactoBlood El equipo de UC Berkeley trabajó para desarrollar un sustituto de glóbulos rojos rentable construido a partir de bacterias E. coli diseñadas. El sistema está diseñado para transportar oxígeno de manera segura en el torrente sanguíneo sin inducir sepsis, y para ser almacenado por periodos prolongados en estado liofilizado.
E. Chromi El proyecto del equipo Cambridge se esforzó por facilitar el diseño y construcción de biosensores. Diseñaron y caracterizaron dos tipos de piezas - Afinadores de Sensibilidad y Generadores de Color - E. coli diseñados para producir diferentes pigmentos en respuesta a diferentes concentraciones de un inductor. La disponibilidad de estas piezas revolucionó el camino del futuro diseño de biosensores.

