7.3: Inhibición lateral en el patrón del sistema nervioso
- Page ID
- 53081
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
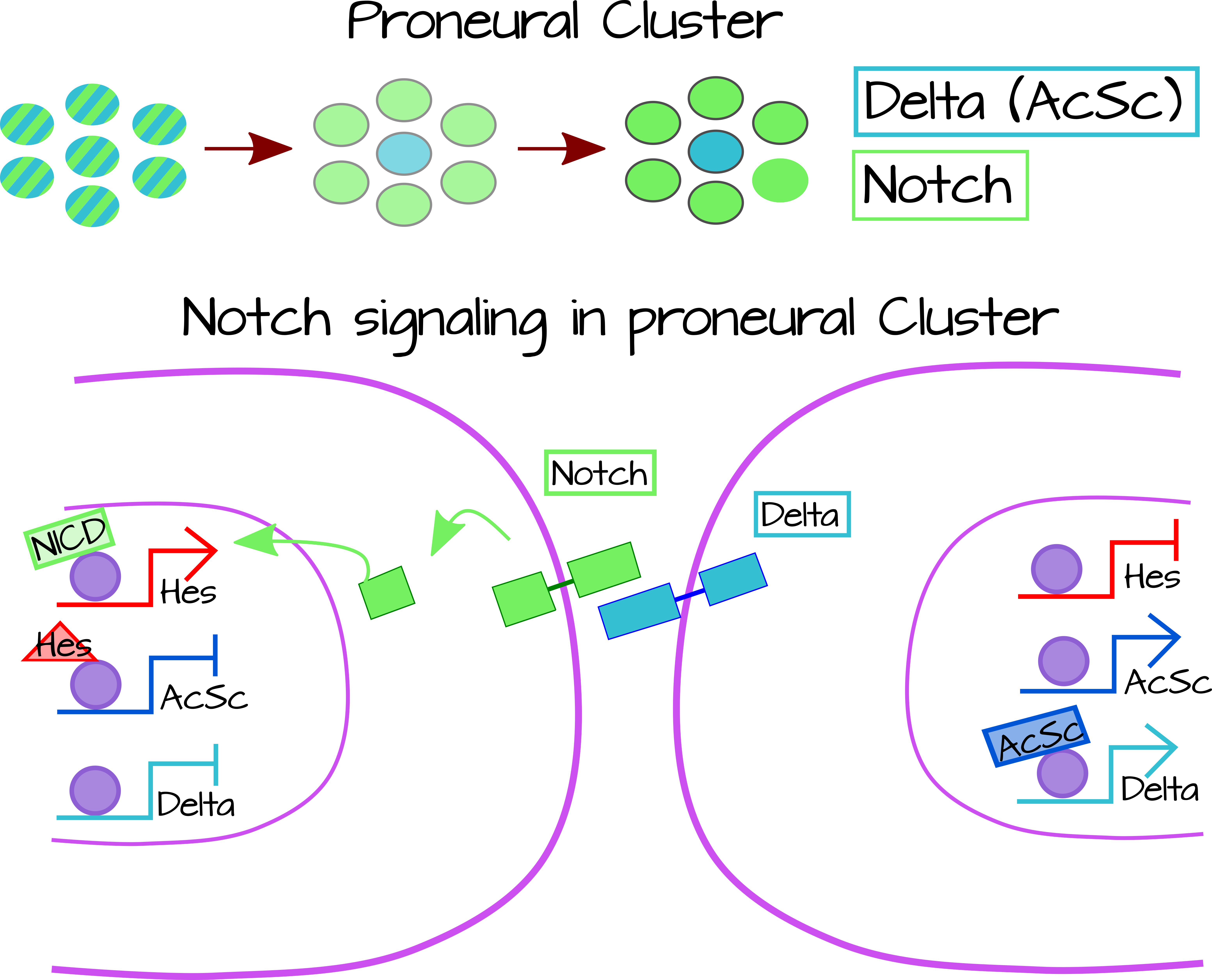
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)No todas las células en un sistema nervioso pueden ser neuronas. De hecho, en nuestro propio sistema nervioso central alrededor de la mitad a 3/4 de las células son células gliales 3. En Drosophila, las células progenitoras neurales (neuroblastos) son solo una porción de todas las células de neurectodermo. Estos neuroblastos expresan factores de transcripción proneural bHLH incluyendo genes del complejo Achaete-Scute. El complejo Achaete-Scute (Ac-Sc) es un grupo de 4 genes bHLH cuyos productos proteicos forman homo- y heterodímeros para regular la transcripción de genes neuronales. Las células que expresan Ac-Sc se deslaminan de la lámina de neurectodermo y finalmente dan lugar a neuronas 4. Antes de resumir el mecanismo similar a un patrón de Turing para la especificación de neuroblastos, primero quiero mencionar que la formación de neuroblastos de Drosophila difiere en una manera muy importante de un patrón tradicional de Turing: cada neuroblasto surge aislado de otros neuroblastos. Este patrón no es sobre un tejido entero sino que es súper local, ocurriendo solo sobre un grupo de 6-7 células con solo una célula convirtiéndose en una neurona (Figura 5) 5.
Una versión svg editable de esta figura se puede descargar en https://scholarlycommons.pacific.edu/open-images/37/
Después de la especificación del clúster proneural, ocurren oscilaciones aleatorias de los componentes de la vía de señalización de Notch hasta que una célula expresa aleatoriamente niveles más altos de Delta, el ligando Esto activa la señalización de Notch en las células vecinas. La activación de la vía de señalización Notch provoca la expresión del gen Hes Enhancer-of-Split, que es un represor del complejo aCSC. AcSC normalmente aumenta la expresión de Delta, pero en estas células Notch positivas donde acSC está fuera de Delta los niveles comienzan a bajar. En la celda Delta positiva vecina, no hay señalización Notch para desactivar aCSC (debido a la falta de Delta en todas las celdas vecinas) y los niveles de Delta permanecen altos. De esta manera, una célula en el cúmulo proneural es positiva para delta/ACSC y las células vecinas son Notch/Hes positivas 6 (Figura 3). Este sistema crea una célula positiva Delta en un campo a través de la inhibición lateral. En el modelo Turing, Delta sería el activador local y Notch sería el represor. Delta activa su propia expresión al no apagar aCSC y activa Notch en células vecinas. A diferencia del modelo de Turing, Notch y Delta no necesitan difundirse para que esto funcione, sino que la señal en sí es propagada por la señalización célula-célula.
Los morfógenos graduados, la reacción-difusión y la inhibición lateral son formas de generar patrones que se repiten (como manchas y rayas) o polarizan un tejido. Si alguna vez has estado en la playa o has tomado un curso de física sabes que otra forma de generar un patrón repetitivo es con un oscilador que emite una función de onda. Si no has tomado física (o estás oxidado) la forma más fácil de pensar en ondas y osciladores es imaginarte sosteniendo una cuerda que está atada a un poste. A medida que mueve el brazo hacia arriba y hacia abajo, actúa como oscilador, moviéndose de la posición alta a la posición baja. El rango de movimiento de tu brazo determina la altura de la ola. Puedes hacer que las ondas se muevan más rápido (con mayor frecuencia) moviendo tu brazo más rápido. La velocidad del oscilador determina la velocidad a la que se mueven las olas. Si tu brazo se mueve demasiado despacio, la ola se desintegra rápidamente y nunca llega a la pared.
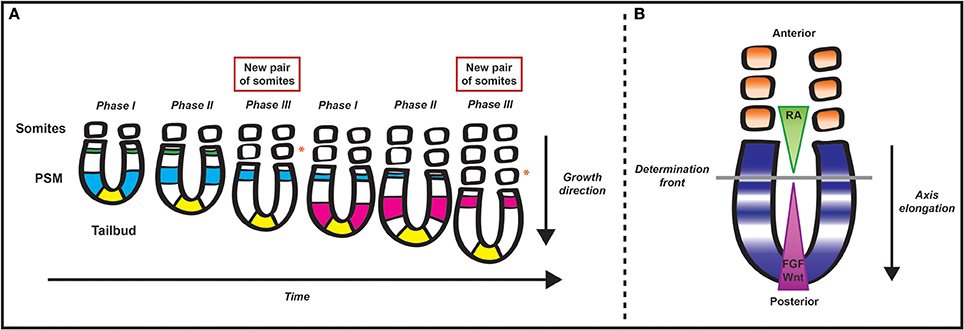
Las ondas y los osciladores también ocurren en sistemas biológicos. Uno de ellos es el oscilador de segmentación en vertebrados. En este caso la expresión génica de los genes de la vía Notch actúa como oscilador. Se ha propuesto un modelo de “reloj y frente de onda” para describir patrones segmentarios en vertebrados. La segmentación del mesodermo que rodea a la notocorda (los somitas) ocurre de manera anterior a posterior, con somitas brotando de una zona posterior de mesodermo no segmentado. A medida que brotan nuevos somitas, los viejos son empujados anteriormente, de tal manera que los somitas más anteriores brotan primero. El “reloj de segmentación” es el oscilador en el mesodermo pre-somítico en la parte posterior del animal. Una “ola” de expresión génica de clase hes viaja a través del mesodermo presomítico (PSM). Cuando alcanza el punto más anterior del mesodermo presomítico se detiene y el tejido brota del PSM. El marcador que indica a los tejidos cuando están lo suficientemente anteriores son gradientes opuestos de ácido retinoico (AR) en la parte anterior y FGF en la posterior. Cuando una onda de expresión de hes alcanza niveles bajos/moderados de cada molécula de gradiente, esto indica que el tejido se somitogénesis (gemación de somitas) 7.
En el video de arriba, la expresión del gen Hes en el PSM es en azul y en los somitas es roja. Entonces, la pregunta que podrías estar haciendo ahora es “¿Cómo actúa Hes como oscilador pero también viaja en una ola?” La ola que estás viendo en el video de arriba es una ola de expresión génica. Las propias células se mueven muy poco. Imagínese un estadio lleno de gente haciendo “la ola”. Si la ola tarda 1 minuto en moverse por el estadio, puede ser propugnada por un público coordinado si cada persona levanta la mano 1 segundo cada minuto.
La gente misma no está corriendo por el estadio, sino que están “oscilando” entre los brazos arriba y abajo. De igual manera, en el PSM cada celda oscila entre Hes on y Hes off. Al igual que una ola de estadio, las células están probando su entorno local para coordinarse con las celdas cercanas. Cuando la ola llega a niveles bajos de FGF y niveles más altos de RA se detiene, al igual que una ola golpeando una pared, y las células con patrones de expresión coincidentes brotan del PSM como somita (Figura 6).
¿Cómo actúa Hes como oscilador y cómo se coordinan las celdas entre sí? No sabemos al 100% la respuesta a esto, pero hay mucha evidencia de que Hes es un oscilador intrínseco celular y la ruta Notch (que controla la expresión de Hes) ayuda a coordinar las células vecinas. Los genes Hes son factores de transcripción y algunos genes Hes inhiben su propia expresión. Cuando los niveles de proteína de Hes son lo suficientemente altos, los niveles de transcripción bajan. Estos ARNm y proteínas de Hes también tienen una vida media corta, por lo que cuando los niveles de transcripción bajan, no permanecen bajos por mucho tiempo. A medida que el ARNm y las proteínas se degradan, se libera la represión de la expresión de Hes y se vuelve a hacer ARNm Por supuesto, esto eleva el nivel de proteína Hes, que reprime la expresión de Hes. De esta manera la expresión de Hes actúa como oscilador, siempre y cuando mantenga al menos un bajo nivel de expresión y se degrada rápidamente. En ratones, la degradación de la proteína ocurre dentro de los 20 minutos. Los mutantes estables que duran 30 minutos han amortiguado el comportamiento oscilatorio, al igual que subir y bajar el brazo demasiado lentamente al hacer ondas de cuerda 7. La oscilación es una propiedad intrínseca de las células, puede ocurrir en cultivo celular después del tratamiento con suero, pero esta oscilación está descoordinada entre las células - después de unos ciclos donde todas las células muestran la misma periodicidad de oscilación, comienzan a perder coordinación y oscilan en su propia longitud de onda 8 ,9.
Como puedes ver en el video anterior, las células en el mesodermo presomítico tienden a tener el mismo estado de oscilación de Hes que sus vecinos. Esto es probablemente coordinado a través de la vía de señalización de Notch. Como ya hemos visto, la vía Notch es una vía de señalización yuxtacrina con células contiguas comunicándose a través de receptores de membrana celular. Una vez activado, Notch sufre escisión intramembrana y la mitad citoplásmica viaja al núcleo donde libera represión sobre factores de transcripción de clase Su (H) para activar la expresión génica de efectores como los genes Hes (piloso/potenciador de genes divididos también llamados genes Her). Mediante la señalización entre sí, las células PSM pueden ejercer retroalimentación positiva sobre sus vecinos de tal manera que las células positivas para Notch inducen la señalización de Notch en las celdas de al lado 10.


