6.2: Transformar la matriz de varianza-covarianza evolutiva
- Page ID
- 54192
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)En 1999, Mark Pagel introdujo tres modelos estadísticos que permiten probar si los datos se desvían de un proceso de tasa constante que evoluciona en un árbol filogenético (Pagel 1999a, b) 1. Cada uno de estos tres modelos es una transformación estadística de los elementos de la matriz filogenética varianza-covarianza, C, que encontramos por primera vez en el Capítulo 3. Los tres también pueden considerarse como una transformación de las longitudes de las ramas del árbol, lo que agrega una comprensión más intuitiva de las propiedades estadísticas de las transformaciones del árbol (Figura 6.1). Podemos transformar el árbol y luego simular personajes bajo un modelo de movimiento browniano en el árbol transformado, generando patrones muy diferentes que si hubieran sido simulados en el árbol inicial.
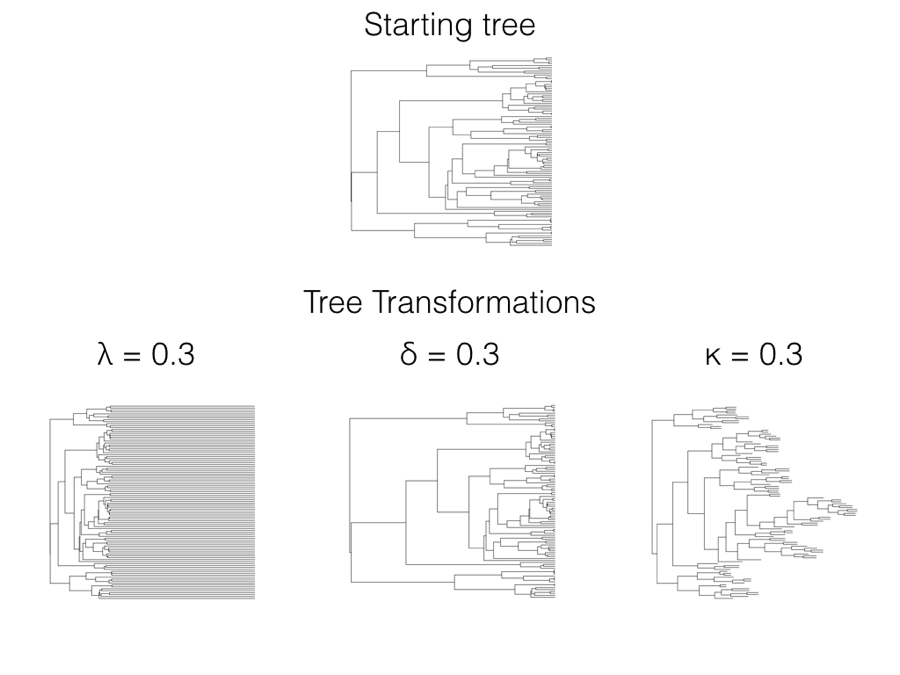
Hay tres transformaciones de árboles Pagel (lambda: λ, delta: δ y kappa: κ). Describiré cada uno de ellos junto con métodos comunes para ajustar modelos Pagel bajo marcos ML, AIC y Bayesianos. Las tres transformaciones de Pagel también pueden estar relacionadas con procesos evolutivos, aunque esas relaciones son a veces vagas en comparación con enfoques basados en modelos evolutivos explícitos más que en transformaciones de árboles (ver más abajo para más comentarios sobre esta distinción).
Quizás la transformación del árbol Pagel más utilizada es λ. Cuando se usa λ, se multiplica todos los elementos fuera de la diagonal en la matriz de varianza-covarianza filogenética por el valor de λ, restringido a valores de 0 ≤ λ ≤ 1. Los elementos diagonales permanecen inalterados. Entonces, si la matriz original para r especies es:
\[ \mathbf{C_o} = \begin{bmatrix} \sigma_1^2 & \sigma_{12} & \dots & \sigma_{1r}\\ \sigma_{21} & \sigma_2^2 & \dots & \sigma_{2r}\\ \vdots & \vdots & \ddots & \vdots\\ \sigma_{r1} & \sigma_{r2} & \dots & \sigma_{r}^2\\ \end{bmatrix} \label{6.1}\]
Entonces la matriz transformada será:
\[ \mathbf{C_\lambda} = \begin{bmatrix} \sigma_1^2 & \lambda \cdot \sigma_{12} & \dots & \lambda \cdot \sigma_{1r}\\ \lambda \cdot \sigma_{21} & \sigma_2^2 & \dots & \lambda \cdot \sigma_{2r}\\ \vdots & \vdots & \ddots & \vdots\\ \lambda \cdot \sigma_{r1} & \lambda \cdot \sigma_{r2} & \dots & \sigma_{r}^2\\ \end{bmatrix} \label{6.2}\]
En términos de transformaciones de longitud de rama, λ comprime las ramas internas mientras deja las ramas de punta del árbol inafectadas (Figura 6.1). λ puede variar de 1 (sin transformación) a 0 (lo que da como resultado una filogenia estelar completa, con todas las ramas de punta iguales en longitud y todas las ramas internas de longitud 0). En principio se pueden usar algunos valores de λ mayores que uno en la mayoría de las matrices de varianza-covarianza, aunque muchos valores de λ > 1 dan como resultado matrices que no son matrices válidas de varianza-covarianza y/o no se corresponden con ninguna transformación filogenética de árboles. Por esta razón recomiendo que λ se limite a valores entre 0 y 1.
λ se utiliza a menudo para medir la “señal filogenética” en datos comparativos. Esto tiene sentido intuitivo, ya que λ escala el árbol entre un modelo de tasas constantes (λ = 1) a uno donde cada especie es estadísticamente independiente de todas las demás especies del árbol (λ = 0). Estadísticamente, esta puede ser información muy útil. Sin embargo, existe cierto peligro en atribuir un resultado estadístico —ya sea señal filogenética o no— a algún proceso biológico en particular. Por ejemplo, la señal filogenética a veces se llama una “restricción filogenética”. Pero una forma de obtener una señal filogenética alta (λ cerca de 1) es evolucionar rasgos bajo un modelo de movimiento browniano, que implica una evolución de carácter completamente sin restricciones. De igual manera, la falta de señal filogenética —que podría llamarse “restricción filogenética baja ”— resulta de un modelo de OU con un parámetro α alto (ver más abajo), que es un modelo donde la evolución de rasgos lejos del valor óptimo es, de hecho, altamente restringida. Revell y col. (2008) muestran una amplia gama de circunstancias que pueden conducir a patrones de señal filogenética alta o baja, y precaución contra la sobreinterpretación de los resultados de los análisis de la señal filogenética, como la λ de Pagel. También vale la pena señalar que las estimaciones estadísticas de λ bajo un modelo ML tienden a agruparse cerca de 0 y 1 independientemente del valor verdadero, y la selección del modelo AIC puede tender a preferir modelos con λ ≠ 0 incluso cuando los datos se simulan bajo movimiento browniano (Boettiger et al. 2012).
La δ de Pagel está diseñada para capturar la variación en las tasas de evolución a través del tiempo. Bajo la transformación δ, todos los elementos de la matriz filogenética varianza-covarianza se elevan a la potencia δ, que se supone que es positiva. Entonces, si nuestra matriz C original se da arriba (ecuación 6.1), entonces la versión transformada δ será:
\[ \mathbf{C_\delta} = \begin{bmatrix} (\sigma_1^2)^\delta & (\sigma_{12})^\delta & \dots & (\sigma_{1r})^\delta\\ (\sigma_{21})^\delta & (\sigma_2^2)^\delta & \dots & (\sigma_{2r})^\delta\\ \vdots & \vdots & \ddots & \vdots\\ (\sigma_{r1})^\delta & (\sigma_{r2})^\delta & \dots & (\sigma_{r}^2)^\delta\\ \end{bmatrix} \label{6.3} \]
Dado que estos elementos representan las alturas de los nodos en el árbol filogenético, entonces δ también puede verse como una transformación de las alturas de los nodos filogenéticos. Cuando δ es uno, el árbol no se modifica y uno todavía tiene un proceso de movimiento browniano de velocidad constante; cuando δ es menor que 1, las alturas de los nodos se reducen, pero las ramas más profundas en el árbol se reducen menos que las ramas menos profundas (Figura 6.1). Esto representa efectivamente un modelo donde la tasa de evolución se ralentiza a través del tiempo. Por el contrario, δ > 1 estira las ramas menos profundas del árbol más que las ramas profundas, imitando un modelo donde la tasa de evolución se acelera a través del tiempo. Existe una estrecha conexión entre el modelo δ, el modelo ACDC (Blomberg et al. 2003) y el modelo de estallido temprano de Harmon et al. (2010) [ver también Uyeda y Harmon (2014), especialmente el apéndice).
Finalmente, la transformación de κ se utiliza a veces para capturar patrones de cambio “especiacional” en árboles. En el modelo κ, uno eleva todas las longitudes de rama en el árbol por la potencia κ (requerimos que κ ≥ 0). Esto tiene un efecto complicado en la matriz filogenética varianza-covarianza, ya que el efecto que esta transformación tiene en cada elemento de covarianza depende tanto del valor de κ como del número de ramas que se extienden desde la raíz del árbol hasta el ancestro común más reciente de cada par de especies. Entonces, si nuestra matriz C original viene dada por Ecuación\ ref {6.1}, la versión transformada será:
\[ \begin{array}{l} \mathbf{C_o} = \\ \left(\begin{smallmatrix} b_{1,1}^k + b_{1,2}^k \dots + b_{1,d_1}^k & b_{1-2,1}^k + b_{1-2,2}^k \dots + b_{1-2,d_{1-2}}^k & \dots & b_{1-r,1}^k + b_{1-r,2}^k \dots + b_{1-r,d_{1-r}}^k \\ b_{2-1,1}^k + b_{2-1,2}^k \dots + b_{2-1,d_{1-2}}^k & b_{2,1}^k + b_{2,2}^k \dots + b_{2,d_2}^k & \dots & b_{2-r,1}^k + b_{2-r,2}^k \dots + b_{2-r,d_{2-r}}^k \\ \vdots & \vdots & \ddots & \vdots\\ b_{r-1,1}^k + b_{r-1,2}^k \dots + b_{r-1,d_{1-r}}^k & b_{r-2,1}^k + b_{r-2,2}^k \dots + b_{r-2,d_{1-2}}^k & \dots & b_{r,1}^k + b_{r,2}^k \dots + b_{r,d_{r}}^k \\ \end{smallmatrix}\right) \\ \end{array} \label{6.4} \]
donde b x, y es la longitud de rama de la rama que es el ancestro común más reciente de los taxones x e y, mientras que d x, y es el número total de ramas que uno encuentra atravesando el camino desde la raíz hasta el ancestro común más reciente del par de especies especificado por x, y (o hasta la punta x si solo se especifica un taxón). No hace falta decir que esta transformación es más fácil de entender como una transformación de las propias ramas de los árboles que de la matriz de varianza-covarianza asociada.
Cuando el parámetro κ es uno, el árbol no se modifica y uno todavía tiene un proceso de movimiento browniano de velocidad constante; cuando κ= 0, todas las longitudes de rama son una. Los valores de κ entre estos dos extremos representan intermedios (Figura 6.1). κ a menudo se interpreta en términos de un modelo donde el cambio de carácter se concentra más o menos en eventos de especiación. Para que esta interpretación sea válida, tenemos que suponer que el árbol filogenético, según se da, incluye todos (o incluso la mayoría) de los eventos de especiación en la historia del clado. El problema con esta suposición es que es casi seguro que faltan eventos de especiación debido al muestreo: quizás algunas especies vivas del clado no han sido muestreadas, o especies que forman parte del clado se han extinguido antes del día de hoy y por lo tanto no se muestrean. Hay formas mucho mejores de estimar modelos especiacionales que pueden dar cuenta de estos problemas en el muestreo (por ejemplo, Bokma 2008; Goldberg e Igić 2012); estos métodos más nuevos deberían preferirse sobre el κ de Pagel para probar un patrón especiacional en datos de rasgos.
Hay dos formas principales de evaluar el ajuste de los tres modelos estilo página a los datos. Primero, se puede usar ML para estimar parámetros y pruebas de relación de verosimilitud (o puntuaciones A I C c) para comparar el ajuste de varios modelos. Cada uno representa un modelo de tres parámetros: un parámetro adicional agregado a los dos parámetros ya necesarios para describir el movimiento browniano de tasa única. Como se mencionó anteriormente, los estudios de simulación sugieren que esto a veces puede conducir a un exceso de confianza, al menos para el modelo λ. En ocasiones, los investigadores compararán el ajuste de un modelo particular (por ejemplo λ) con modelos donde ese parámetro se fija en sus dos valores extremos (0 o 1; esto no es posible con δ). Segundo, se pueden usar métodos bayesianos para estimar distribuciones posteriores de valores de parámetros, luego inspeccionar esas distribuciones para ver si se superponen con valores de interés (digamos, 0 o 1).
Podemos aplicar estos tres modelos Pagel a los datos del tamaño corporal de los mamíferos discutidos en el capítulo 5, comparando las puntuaciones A I C c para el movimiento browniano con las de las tres transformaciones. Obtenemos los siguientes resultados:
| Modelo | Estimaciones de parámetros | LnL | A I C c |
|---|---|---|---|
| Movimiento browniano | σ 2 = 0.088, θ = 4.64 | -78.0 | 160.4 |
| lambda | σ 2 = 0.085, θ = 4.64, λ = 1.0 | -78.0 | 162.6 |
| delta | σ 2 = 0.063, θ = 4.60, δ = 1.5 | -77.7 | 162.0 |
| kappa | σ 2 = 0.170, θ = 4.64, κ = 0.66 | -77.3 | 161.1 |
Tenga en cuenta que el movimiento browniano es el modelo preferido con la puntuación A I C c más baja, pero también que las cuatro puntuaciones A I C c están dentro de las 3 unidades, lo que significa que no podemos fácilmente distinguir entre ellos usando nuestros datos de mamíferos.


