1.4: Enlace no covalente
- Page ID
- 57071
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Enlace no covalente
La unión no covalente no implica compartir electrones. En su lugar:
- mantiene juntas las dos hebras de la doble hélice de ADN (enlaces de hidrógeno)
- pliega los polipéptidos en estructuras secundarias tales como la hélice alfa y la conformación beta
- permite que las enzimas se unan a su sustrato
- permite que los anticuerpos se unan a su antígeno
- permite que los factores de transcripción se unan entre sí
- permite que los factores de transcripción se unan al ADN
- permite que las proteínas (por ejemplo, algunas hormonas) se unan a su receptor
- permite el montaje de maquinaria macromolecular como
- ribosomas
- filamentos de actina
- microtúbulos
- y muchos más
Hay tres tipos principales de fuerzas no covalentes:
- interacciones iónicas
- interacciones hidrofóbicas
- enlaces de hidrógeno
Interacciones Iónicas
A cualquier pH dado, las proteínas tienen grupos cargados que pueden participar en su unión entre sí o con otros tipos de moléculas. Por ejemplo, como muestra la figura, los grupos carboxilo cargados negativamente en los residuos de ácido aspártico (Asp) y ácido glutámico (Glu) pueden ser atraídos por los grupos amino libres cargados positivamente en los residuos de lisina (Lys) y arginina (Arg).
Las interacciones iónicas son altamente sensibles a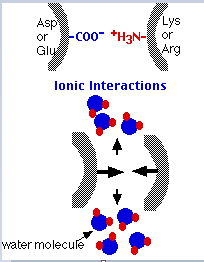
- cambios en el pH.
A medida que el pH desciende,
- H + se unen a los grupos carboxilo (COO -) de ácido aspártico (Asp) y ácido glutámico (Glu), neutralizando su carga negativa, y
- H + se unen al par desocupado de electrones en el átomo de N de los grupos amino (NH 2) de lisina (Lys) y arginina (Arg) dándoles una carga positiva
El resultado: No solo cambia la carga neta sobre la molécula (se vuelve más positiva) sino que se alteran muchas de las oportunidades que tienen sus grupos R para interacciones iónicas (electrostáticas) con otras moléculas e iones.
A medida que aumenta el pH,
- H + se eliminan de los grupos COOH de Asp y Glu, dándoles una carga negativa (COO −), y
- H + se eliminan de los grupos NH 3 + de Lys y Arg eliminando su carga positiva
El resultado: Nuevamente la carga neta sobre la molécula cambia (se vuelve más negativa) y, nuevamente, se alteran muchas de las oportunidades que tienen sus grupos R para interacciones electrostáticas con otras moléculas o iones.
- concentración de sal
El aumento de la concentración de sal reduce la fuerza de unión iónica al proporcionar iones competidores para los residuos cargados.
Interacciones hidrófobas
 Las cadenas laterales (grupos R) de aminoácidos tales como la fenilalanina y la leucina son no polares y por lo tanto interactúan mal con moléculas polares como el agua. Por esta razón, la mayoría de los residuos no polares en las proteínas globulares se dirigen hacia el interior de la molécula mientras que grupos polares como el ácido aspártico y la lisina están en la superficie expuestos al disolvente. Cuando los residuos no polares se exponen en la superficie de dos moléculas diferentes, es energéticamente más favorable que sus dos superficies no polares “aceitosas” se acerquen entre sí desplazando de cerca las moléculas polares de agua entre ellas.
Las cadenas laterales (grupos R) de aminoácidos tales como la fenilalanina y la leucina son no polares y por lo tanto interactúan mal con moléculas polares como el agua. Por esta razón, la mayoría de los residuos no polares en las proteínas globulares se dirigen hacia el interior de la molécula mientras que grupos polares como el ácido aspártico y la lisina están en la superficie expuestos al disolvente. Cuando los residuos no polares se exponen en la superficie de dos moléculas diferentes, es energéticamente más favorable que sus dos superficies no polares “aceitosas” se acerquen entre sí desplazando de cerca las moléculas polares de agua entre ellas.
La fuerza de las interacciones hidrofóbicas no se ve afectada apreciablemente por cambios en el pH o en la concentración de sal.
Enlaces de hidrógeno
Los enlaces de hidrógeno se pueden formar cuando
- un átomo fuertemente electronegativo (por ejemplo, oxígeno, nitrógeno) se aproxima
- un átomo de hidrógeno que está unido covalentemente a un segundo átomo fuertemente electronegativo
Algunos ejemplos comunes:
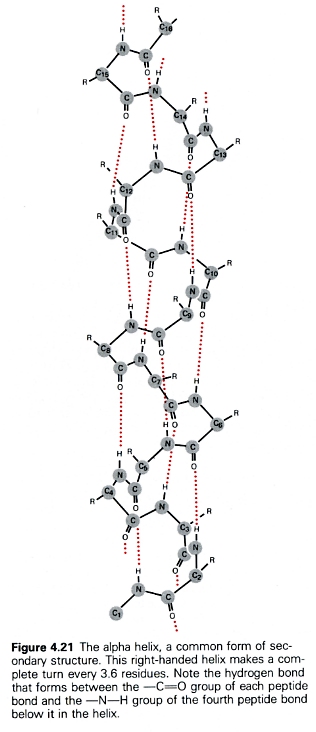
- entre el grupo -C=O y el grupo H-N- de enlaces peptídicos cercanos en proteínas dando lugar a la hélice alfa y la configuración beta

- Entre los grupos −C=O y los grupos hidroxilo (H-O-) en
- residuos de serina y treonina de proteínas y
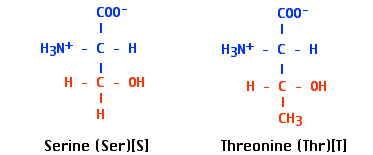
- azúcares
- residuos de serina y treonina de proteínas y
Las interacciones no covalentes son individualmente débiles pero colectivamente fuertes.
Las tres formas de interacciones no covalentes son individualmente débiles (del orden de 5 kcal/mol) en comparación con un enlace covalente (con su 90—100 kcal/mol de energía de enlace). Y qué fuerza tienen estas interacciones requiere que los grupos interactuantes puedan acercarse entre sí de cerca (un angstrom o menos). Por lo que podemos concluir que todos los ejemplos dados en la parte superior de la página requieren:
- un número sustancial de interacciones no covalentes que trabajan juntas para mantener unidas las estructuras
- una topografía superficial que permite que áreas sustanciales de dos superficies que interactúan se acerquen entre sí de cerca; es decir, deben ajustarse entre sí


