6.4: La Traducción del ARN en Proteínas
- Page ID
- 57131
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)

Esta imagen muestra la estructura del ARN de transferencia de alanina (ARNt ala) de levadura. Consiste en una sola hebra de 77 ribonucleótidos. La cadena está plegada sobre sí misma, y muchas de las bases se emparejan entre sí formando cuatro regiones helicoidales. Los bucles se forman en las regiones desapareadas de la cadena. (Las bases rodeadas en azul han sido modificadas quimicamente después de la sintesis de la molecula.)
Al menos un tipo de ARNt está presente por cada uno de los 20 aminoácidos utilizados en la síntesis de proteínas. (Algunos aminoácidos emplean los servicios de dos o tres ARNt diferentes, por lo que la mayoría de las células contienen hasta 32 tipos diferentes de ARNt). El aminoácido se une al ARNt apropiado mediante una enzima activadora (una de las 20 aminoacil-ARNt sintetasas) específica para ese aminoácido así como para el ARNt asignado a él.
Cada tipo de ARNt tiene una secuencia de 3 nucleótidos desapareados —el anticodón — que puede unirse, siguiendo las reglas del apareamiento de bases, al triplete complementario de nucleótidos —el codón — en una molécula de ARN mensajero (ARNm). Así como la replicación y transcripción del ADN implican el emparejamiento de bases de nucleótidos que corren en dirección opuesta, así la lectura de codones en ARNm (5' -> 3') requiere que los anticodones se unan en la dirección opuesta.
Anticodón: 3' CGA 5'
Codón: 5' GCU 3'
Los codones de ARN
| U | C | A | G | ||
|---|---|---|---|---|---|
| U | UUU Fenilalanina (Phe) | Serina UCU (Ser) | Tirosina UAU (Tyr) | Cisteina UGU (Cys) | U |
| UUC Phe | Ser UCC | UAC Tyr | Cys UGC | C | |
| UUA Leucina (Leu) | Ser de la UCA | PARADA UAA | PARADA UGA | A | |
| UUG Leu | UCG Ser | PARADA UAG | UGG Triptófano (Trp) | G | |
| C | CUU Leucina (Leu) | CCU Prolina (Pro) | CAU Histidina (His) | CGU Arginina (Arg) | U |
| CUC Leu | CCC Pro | CAC Su | CGC Arg | C | |
| CUA Leu | CCA Pro | CAA Glutamina (Gln) | CGA Arg | A | |
| CUG Leu | CCG Pro | CAG Gln | CGG Arg | G | |
| A | AUU Isoleucina (Ile) | ACU Treonina (Thr) | AAU Asparagina (Asn) | AGU Serina (Ser) | U |
| AUC Ile | ACC Thr | AAC Asn | AGC Ser | C | |
| AUA Ile | ACA Thr | Lisina AAA (Lys) | AGA Arginina (Arg) | A | |
| AUG Metionina (Met) o INICIO | ACG Thr | AAG Lys | AGG Arg | G | |
| G | GUU Valine Val | GCU Alanina (Ala) | GAU Ácido aspártico (Asp) | GGU Glicina (Gly) | U |
| GUC | CCG Ala | GAC Asp | GGC | C | |
| GUA Val | GCA Ala | GAA Ácido glutámico (Glu) | GGA Gly | A | |
| GUG Val | GCG Ala | GAG Glu | GGG Gly | G |
Nota:
- La mayoría de los aminoácidos están codificados por codones sinónimos que difieren en la tercera posición del codón.
- En algunos casos, un solo ARNt puede reconocer dos o más de estos codones sinónimos.
- Ejemplo: el ARNt de fenilalanina con el anticodón 3' AAG 5' reconoce no solo UUC sino también UUU.
- La violación de las reglas habituales de apareamiento de bases en el tercer nucleótido de un codón se llama “bamboleo”
- El codón AUG cumple dos funciones relacionadas
- Comienza cada mensaje; es decir, señala el inicio de la traducción colocando el aminoácido metionina en el amino terminal del polipéptido a sintetizar.
- Cuando ocurre dentro de un mensaje, orienta la incorporación de metionina.
- Tres codones, UAA, UAG y UGA, actúan como señales para terminar la traducción. Se llaman codones STOP.
Los pasos de la traducción
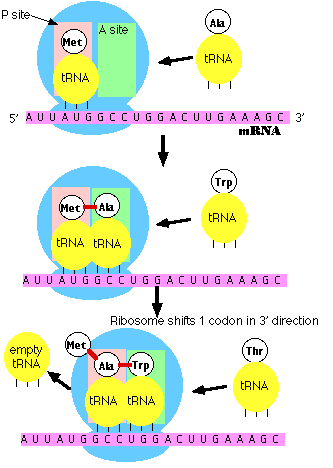
Iniciación
- La subunidad pequeña del ribosoma se une a un sitio “aguas arriba” (en el lado 5') del inicio del mensaje.
- Prosigue aguas abajo (5' -> 3') hasta que encuentra el codón de inicio AUG. (La región entre el capuchón de ARNm y el AUG se conoce como la región 5' no traducida [5'-UTR]).
- Aquí se une la subunidad grande y un ARNt iniciador especial.
- El ARNt iniciador se une al sitio P (mostrado en rosa) en el ribosoma.
- En eucariotas, el ARNt iniciador porta metionina (Met). (Las bacterias usan una metionina modificada designada fMet.)
Alargamiento
- Un aminoacil-ARNt (un ARNt unido covalentemente a su aminoácido) capaz de emparejarse con el siguiente codón en el ARNm llega al sitio A (verde) asociado con:
- un factor de elongación (llamado EF-Tu en bacterias; EF-1 en eucariotas)
- GTP (la fuente de la energía necesaria)
- El aminoácido precedente (Met al inicio de la traducción) se une covalentemente al aminoácido entrante con un enlace peptídico (mostrado en rojo).
- El ARNt iniciador se libera del sitio P.
- El ribosoma se mueve un codón aguas abajo.
- Esto desplaza el ARNt más recientemente llegado, con su péptido unido, al sitio P y abre el sitio A para la llegada de un nuevo aminoacil-ARNt.
- Este último paso es promovido por otro factor de elongación proteica (llamado EF-G en bacterias; EF-2 en eucariotas) y la energía de otra molécula de GTP.
Nota: el ARNt iniciador es el único miembro de la familia de ARNt que puede unirse directamente al sitio P. El sitio P se denomina así porque, con la excepción del ARNt iniciador, se une únicamente a una molécula de p eptidil-ARNt; es decir, un ARNt con el péptido en crecimiento unido.
El sitio A se llama así porque se une solo al minoacil-ARNt entrante; es decir, el ARNt que trae el siguiente aminoácido. Entonces, por ejemplo, el ARNt que lleva a Met al interior del polipéptido puede unirse solo al sitio A.
Terminación
- El final de la traducción ocurre cuando el ribosoma alcanza uno o más codones STOP (UAA, UAG, UGA). (Los nucleótidos desde este punto hasta la cola poli (A) forman la región 3' no traducida [3'-UTR] del ARNm).
- No hay moléculas de ARNt con anticodones para codones STOP.
- Sin embargo, los factores de liberación de proteínas reconocen estos codones cuando llegan al sitio A.
- La unión de estas proteínas —junto con una molécula de GTP — libera el polipéptido del ribosoma.
- El ribosoma se divide en sus subunidades, que luego pueden ser reensambladas para otra ronda de síntesis de proteínas.
Polisomas
Una sola molécula de ARNm suele tener muchos ribosomas viajando a lo largo de ella, en diversas etapas de síntesis de la proteína. Este complejo se llama polisoma.
Sesgo de codón
Todos menos dos de los aminoácidos (Met y Trp) pueden ser codificados por de 2 a 6 codones diferentes. Sin embargo, el genoma de la mayoría de los organismos revela que ciertos codones son preferidos sobre otros. En humanos, por ejemplo, la alanina es codificada por GCC cuatro veces más frecuentemente que por GCG. Esto probablemente refleja una mayor eficiencia de traducción por parte del aparato de traducción para ciertos codones sobre sus sinónimos.
- Al inicio de la traducción, se utilizan alternativamente dos o más de un conjunto de codones sinónimos (por ejemplo, los 6 codones que incorporan leucina en la proteína en crecimiento). La necesidad de localizar primero uno y luego otro ARNt para ese aminoácido ralentiza la velocidad de traducción.
- Esto puede ayudar a evitar que los ribosomas choquen entre sí en el polisoma.
- También puede proporcionar más tiempo para que la proteína naciente comience a plegarse correctamente a medida que emerge del ribosoma.
- Una vez que la traducción está en marcha (después de que se hayan agregado 30-50 aminoácidos), tiende a elegirse un codón en particular cada vez que se requiere su aminoácido. Presumiblemente esto ahora aumenta la eficiencia (velocidad) de la traducción.
- La mayoría de los organismos tienen más de los 61 genes necesarios para codificar un ARNt para cada uno de los 61 codones (tenemos 270 genes de ARNt). La presencia de múltiples genes para ARNt con un anticodón idéntico aumenta la concentración de ARNt capaces de unirse a un codón particular. Los ARN mensajeros —especialmente los de genes activos— tienden a favorecer codones que corresponden a abundantes ARNt portadores del anticodón.
El sesgo de codones incluso se extiende a pares de codones: dondequiera que una proteína humana contenga los aminoácidos Ala-Glu, el gen que codifica esos aminoácidos tiene siete veces más probabilidades de usar los codones GCAGAG en lugar del sinónimo GCCGAA. El sesgo de codones es explotado por la industria biotecnológica para mejorar el rendimiento del producto deseado. La capacidad de manipular el sesgo de codones también puede marcar el comienzo de una era de vacunas más seguras.
Control de Calidad
Las moléculas de ARNm defectuosas pueden producirse por mutaciones en el gen así como errores introducidos durante la transcripción (aunque a una tasa notablemente baja). Además de producir ARNm con codones incorrectos para aminoácidos, estos errores pueden producir moléculas de ARNm que tienen
- Codones de Terminación Prematura (PTC); es decir, la introducción de un codón STOP antes del final normal del mensaje. La traducción de estos ARNm produce una proteína truncada que probablemente sea ineficaz y pueda ser dañina. El problema a veces puede resolverse mediante la desintegración del ARNm mediada por sentido (NMD).
- sin codón STOP. Estos producen transcripciones “sin parar”. El problema se puede resolver con Nonstop mRNA Decay.
Decaimiento del ARNm mediado sin sentido (NMD)
Los codones de terminación prematura (PTC) pueden generarse mediante mutaciones “sin sentido”, cambios de marco y errores de procesamiento de ARN (eliminación de intrones). También son una consecuencia inevitable de la creación de receptores de antígeno en las células B y las células T.
Mecanismos
- Durante el procesamiento del ARN dentro del núcleo, se agregan complejos proteicos en cada punto donde los exones adyacentes se empalman juntos. (Estas son señales importantes para exportar el ARNm al citoplasma).
- En el citoplasma, a medida que el ribosoma desciende por el ARNm, estos complejos se eliminan (y se envían de vuelta al núcleo para su reutilización).
- Si el ribosoma encuentra un codón de terminación prematura, no se eliminan las etiquetas exón-exón finales, y esto marca el ARNm defectuoso para su destrucción (en los cuerpos P).
Las mutaciones que introducen codones de terminación prematura son responsables de algunos casos de enfermedades humanas heredadas como la fibrosis quística y la distrofia muscular de Duchenne (DMD).
Un fármaco, denominado PTC124 o ataluren, hace que el ribosoma se salte las PTCs mientras que aún permite la terminación normal de la traducción. El PTC124 ha demostrado ser prometedor en modelos animales de fibrosis quística y DMD y ahora se están realizando ensayos clínicos de fase II en humanos.
Decaimiento de ARNm sin parar
Las transcripciones sin parar ocurren cuando no hay codón STOP en el mensaje. Como resultado, el ribosoma es incapaz de reclutar los factores de liberación necesarios para abandonar el ARNm. Los transcritos sin parar se forman durante el procesamiento del ARN, por ejemplo, poniendo la cola poli (A) antes de que se alcance el codón STOP.
Mecanismos
Los eucariotas y las bacterias manejan el problema de no codón STOP de manera diferente.
- En eucariotas, cuando el ribosoma se detiene al final de la cola poli (A), se reclutan proteínas para liberar el ribosoma para su reutilización y degradar el mensaje defectuoso.
- En las bacterias, una molécula especial de ARN —llamada tmRNA — salva el día. Se llama ARNmT porque tiene las propiedades tanto de un ARN t ransfer como de un ARN m essenger. La parte de transferencia agrega alanina al sitio A en el ribosoma. Luego, el ribosoma pasa a la parte mensajera que codifica 10 aminoácidos que se dirigen a la molécula para su destrucción (y libera el ribosoma para su reutilización).
Regulación de la Traducción
La expresión de la mayoría de los genes se controla a nivel de su transcripción. Los factores de transcripción (proteínas) se unen a promotores y potenciadores que activan (o desactivan) los genes que controlan. Sin embargo, la expresión génica también se puede controlar a nivel de traducción.
Por Maquinaria General de Degradación de ARN
Cuerpos P
El citosol de eucariotas contiene complejos proteicos que compiten con los ribosomas por el acceso a los ARNm. A medida que estos aumentan su actividad, secuestran ARNm en agregados más grandes llamados cuerpos P (para “cuerpos de procesamiento”, pero este procesamiento no debe confundirse con el procesamiento del pre-ARNm para madurar ARNm que ocurre en el núcleo).
Estos complejos proteicos descomponen el mRNA por
- quitando su “tope”
- quitar su cola de poli (A)
- degradando el mensaje restante (mordisqueando en la dirección 5' -> 3')
Queda por aprender qué controla el equilibrio dinámico entre los ribosomas y los cuerpos P para acceder a los ARNm. Pero este mecanismo prevé
- destrucción de ARNm “malos” (por ejemplo, aquellos con codones PARADA prematuros
- recambio de ARNm aumentando así la flexibilidad de la expresión génica en la célula
Exosomas
Se trata de complejos macromoleculares huecos con dos aberturas. Admiten moléculas de ARN desplegadas y las degradan en la dirección 3' -> 5'. (Ni en estructura ni función estos exosomas se asemejan a los exosomas involucrados en la presentación de antígenos que lamentablemente comparten el mismo nombre).
Por microARN (miRNAs)
Aquí pequeñas moléculas de ARN se unen a una porción complementaria en la 3'-UTR del ARNm y evitan que sea traducido por ribosomas y/o desencadene su destrucción. Ambas actividades se llevan a cabo en los órganos P.
Por Riboswitches
Resulta que la regulación del nivel de ciertos metabolitos está controlada por ribointerruptores. Un riboswitch es una parte de una molécula de ARN mensajero (ARNm) con un sitio de unión específico para el metabolito (o un pariente cercano).
Ejemplos:
- Si el pirofosfato de tiamina (la forma activa de tiamina [vitamina B 1]) está disponible en el medio de cultivo de E. coli,
- Se une a un ARN mensajero cuyo producto proteico es una enzima necesaria para sintetizar tiamina a partir de los ingredientes en un medio mínimo.
- La unión induce un desplazamiento alostérico en la estructura del ARNm para que ya no pueda unirse a un ribosoma y así no pueda traducirse en la enzima.
- E. coli ya no desperdicia recursos al sintetizar una vitamina que está disponible preformada.
- Si la vitamina B 12 está presente en la célula, se une al ARNm que codifica una proteína necesaria para importar la vitamina del medio de cultivo. Esto, también, induce un desplazamiento alostérico en el ARNm que impide que se una a un ribosoma. E. coli ya no desperdicia recursos al sintetizar un transportador para una vitamina de la que ya tiene suficiente.
- Algunas bacterias Gram-positivas (E. coli es Gram-negativa) controlan el nivel de azúcar necesario para sintetizar su pared celular con un riboswitch. En este caso, a medida que se acumula la concentración del azúcar, se une al ARN mensajero (ARNm) cuyo producto es la enzima que produce el azúcar. Esto hace que el ARNm se autodestruya por lo que cesa la producción de la enzima —y así del azúcar—.
Se ha sugerido que estos mecanismos reguladores, que no involucran ninguna proteína, son un relicto de un “mundo del ARN”.


