13.1: Replicación de ADN en procariotas
- Page ID
- 58258
El cromosoma procariota es una molécula circular con una estructura enrollada menos extensa que los cromosomas eucariotas. El cromosoma eucariota es lineal y altamente enrollado alrededor de las proteínas. Si bien hay muchas similitudes en el proceso de replicación del ADN, estas diferencias estructurales requieren algunas diferencias en el proceso de replicación del ADN en estas dos formas de vida. La replicación del ADN en procariotas ha sido ampliamente estudiada, por lo que aprenderemos el proceso básico de replicación del ADN procariota, luego nos enfocaremos en las diferencias entre procariotas y eucariotas.
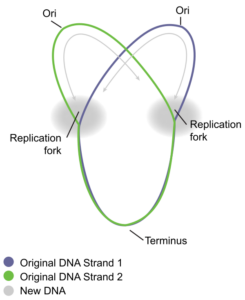
¿Cómo sabe por dónde empezar la maquinaria de replicación? Resulta que existen secuencias nucleotídicas específicas llamadas orígenes de replicación donde comienza la replicación. E. coli tiene un único origen de replicación en su único cromosoma, al igual que la mayoría de los procariotas (Figura\(\PageIndex{1}\)). El origen de replicación es de aproximadamente 245 pares de bases de longitud y es rico en secuencias AT. Esta secuencia de pares de bases es reconocida por ciertas proteínas que se unen a este sitio. Una enzima llamada helicasa desenrolla el ADN rompiendo los enlaces de hidrógeno entre los pares de bases nitrogenadas. La hidrólisis de ATP es necesaria para este proceso porque requiere energía. A medida que el ADN se abre, se forman estructuras en forma de Y llamadas horquillas de replicación (Figura\(\PageIndex{1}\)). Dos bifurcación de replicación se forman en el origen de la replicación y éstas se extienden bidireccionalmente a medida que avanza la replicación. Las proteínas de unión monocatenaria (Figura\(\PageIndex{2}\)) recubren las cadenas simples de ADN cerca de la horquilla de replicación para evitar que el ADN monocatenario vuelva a enrollarse en una doble hélice.
La siguiente enzima importante es la ADN polimerasa III, también conocida como ADN pol III, que agrega nucleótidos uno por uno a la cadena de ADN en crecimiento (Figura\(\PageIndex{2}\)). La adición de nucleótidos requiere energía; esta energía se obtiene de los nucleótidos que tienen tres fosfatos unidos a ellos. El ATP estructuralmente es un nucleótido adenina que tiene tres grupos fosfato unidos; rompiendo el tercer fosfato libera energía. Además de ATP, también hay TTP, CTP y GTP. Cada uno de estos está conformado por el nucleótido correspondiente con tres fosfatos unidos. Cuando se rompe el enlace entre los fosfatos, la energía liberada se utiliza para formar el enlace fosfodiéster entre el nucleótido entrante y la cadena existente.
En procariotas se conocen tres tipos principales de polimerasas: ADN pol I, ADN pol II y ADN pol III. El ADN pol III es la enzima requerida para la síntesis de ADN; el ADN pol I se usa más adelante en el proceso y el ADN pol II se usa principalmente para la reparación (este es otro ejemplo irritante de nomenclatura que se hizo basado en el orden de descubrimiento más que en un orden que tiene sentido).
La ADN polimerasa es capaz de agregar nucleótidos solo en la dirección 5' a 3' (una nueva cadena de ADN solo se puede extender en esta dirección). Requiere un grupo 3′-OH libre (localizado en el azúcar) al que puede agregar el siguiente nucleótido formando un enlace fosfodiéster entre el extremo 3′-OH y el fosfato 5' del siguiente nucleótido. Esto significa esencialmente que no puede añadir nucleótidos si no está disponible un grupo 3′-OH libre. Entonces, ¿cómo agrega el primer nucleótido? El problema se resuelve con la ayuda de una imprimación que proporciona el extremo 3′-OH libre. Otra enzima, la ARN primasa, sintetiza un cebador de ARN que tiene aproximadamente cinco a diez nucleótidos de longitud y complementario al ADN. La ARN primasa no requiere un grupo 3′-OH libre. Debido a que esta secuencia cebra la síntesis de ADN, se le llama apropiadamente el cebador. La ADN polimerasa ahora puede extender este cebador de ARN, agregando nucleótidos uno por uno que son complementarios a la cadena molde (Figura\(\PageIndex{2}\)).
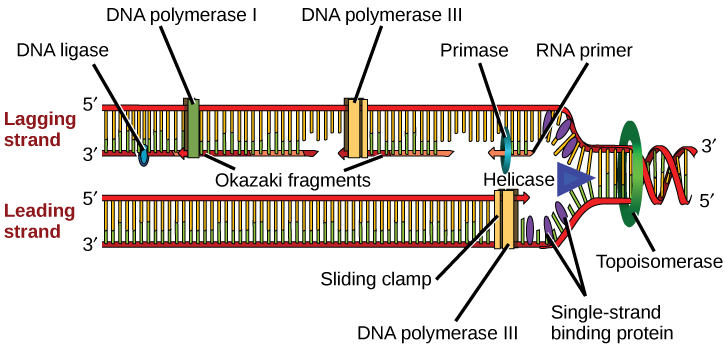
La horquilla de replicación se mueve a razón de 1000 nucleótidos por segundo. La ADN polimerasa solo puede extenderse en la dirección 5′ a 3′, lo que plantea un ligero problema en la horquilla de replicación. Como sabemos, la doble hélice de ADN es antiparalela; es decir, una hebra está en la dirección 5' a 3' y la otra está orientada en la dirección 3' a 5′. Una cadena, que es complementaria a la cadena de ADN parental 3' a 5', se sintetiza continuamente hacia la horquilla de replicación debido a que la polimerasa puede agregar nucleótidos en esta dirección. Esta cadena sintetizada continuamente se conoce como la cadena principal. La otra cadena, complementaria al ADN parental de 5' a 3', se extiende lejos de la horquilla de replicación, en pequeños fragmentos conocidos como fragmentos de Okazaki, cada uno requiriendo un cebador para iniciar la síntesis. Los fragmentos de Okazaki llevan el nombre del científico japonés que los descubrió por primera vez. La hebra con los fragmentos de Okazaki se conoce como la hebra rezagada.
La cadena principal se puede extender con un cebador solo, mientras que la hebra rezagada necesita un nuevo cebador para cada uno de los fragmentos cortos de Okazaki. La dirección general de la hebra rezagada será de 3′ a 5′, y la de la hebra delantera de 5′ a 3′. Una proteína llamada pinza deslizante mantiene la ADN polimerasa en su lugar a medida que continúa agregando nucleótidos. La pinza deslizante es una proteína en forma de anillo que se une al ADN y mantiene la polimerasa en su lugar. La topoisomerasa evita el sobrebobinado de la doble hélice de ADN antes de la horquilla de replicación a medida que el ADN se abre; lo hace causando muescas temporales en la hélice del ADN y luego volver a sellarla. A medida que avanza la síntesis, los cebadores de ARN son reemplazados por ADN pol I, que descompone el ARN y llena los huecos con nucleótidos de ADN. Las mellas que quedan entre el ADN recién sintetizado (que reemplazó al cebador de ARN) y el ADN previamente sintetizado son selladas por la enzima ADN ligasa que cataliza la formación de enlace fosfodiéster entre el extremo 3′-OH de un nucleótido y el extremo 5' fosfato del otro fragmento.
Nota de Lisa
Creo que este proceso es casi imposible de visualizar a partir de la lectura de texto. Te recomiendo encarecidamente que veas un par de animaciones/videos como el que está disponible aquí. Hay enlaces adicionales en Blackboard.
Una vez que el cromosoma se ha replicado completamente, las dos copias de ADN se mueven a dos células diferentes durante la división celular. El proceso de replicación del ADN se puede resumir de la siguiente manera:
- El ADN se desenrolla en el origen de la replicación.
- La helicasa abre las horquillas de replicación formadoras de ADN; estas se extienden en ambas direcciones.
- Las proteínas de unión monocatenaria recubren el ADN alrededor de la horquilla de replicación para evitar el rebobinado del ADN.
- La topoisomerasa se une en la región por delante de la bifurcación de replicación para evitar el superenrollado (sobrebobinado).
- La primasa sintetiza cebadores de ARN complementarios a la cadena de ADN.
- La ADN polimerasa III comienza a agregar nucleótidos al extremo 3′-OH (azúcar) del cebador.
- Continúa el alargamiento tanto de la hebra rezagada como de la hebra delantera.
- Los cebadores de ARN se eliminan y los huecos se rellenan con ADN mediante ADN pol I.
- Los huecos entre los fragmentos de ADN son sellados por ADN ligasa.
Cuadro\(\PageIndex{1}\): Las enzimas involucradas en la replicación del ADN procariota y las funciones de cada una.
| Replicación del ADN procariota: enzimas y su función | |
|---|---|
| Enzima/Proteína | Función específica |
| ADN pol I | La actividad exonucleasa elimina el cebador de ARN y se reemplaza con ADN recién sintetizado |
| ADN pol II | Función de reparación |
| ADN pol III | Enzima principal que añade nucleótidos en la dirección 5′-3' |
| Helicasa | Abre la hélice de ADN rompiendo los enlaces de hidrógeno entre las bases nitrogenadas |
| Ligasa | Sella los huecos entre los fragmentos de Okazaki para crear una cadena de ADN continua |
| Primase | Sintetica los cebadores de ARN necesarios para iniciar la replicación |
| Pinza Deslizante | Ayuda a mantener la ADN polimerasa en su lugar cuando se agregan nucleótidos |
| Topoisomerasa | Ayuda a aliviar el estrés en el ADN al desenrollar al causar roturas y luego volver a sellar el ADN |
| Proteínas de unión monocatenaria (SSB) | Se une al ADN monocatenario para evitar el rebobinado del ADN. |
La replicación del ADN ha sido muy bien estudiada en procariotas, principalmente por el pequeño tamaño del genoma y el gran número de variantes disponibles. Escherichia coli tiene 4.6 millones de pares de bases en un solo cromosoma circular, y todo se replica en aproximadamente 42 minutos, comenzando desde un único origen de replicación y avanzando alrededor del cromosoma en ambas direcciones. Esto significa que se agregan aproximadamente 1000 nucleótidos por segundo. El proceso es mucho más rápido que en eucariotas.
Referencias
A menos que se indique lo contrario, las imágenes de esta página están bajo licencia CC-BY 4.0 de OpenStax.
OpenStax, Conceptos de Biología. OpenStax CNX. mayo 18, 2016 http://cnx.org/contents/s8Hh0oOc@9.10:2ousESf0@5/DNA-Replication

