15.1: El Código Genético
- Page ID
- 58440
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)El Dogma Central: ADN Codifica ARN; ARN Codifica Proteína
Para resumir lo que conocemos hasta este punto, el proceso celular de transcripción genera ARN mensajero (ARNm), una copia molecular móvil de uno o más genes con un alfabeto de A, C, G y uracilo (U). La traducción del molde de ARNm convierte la información genética basada en nucleótidos en un producto proteico. Este flujo de información genética en las células del ADN al ARNm a la proteína es descrito por el Dogma Central (Figura\(\PageIndex{1}\)), que establece que los genes especifican la secuencia de los ARNm, que a su vez especifican la secuencia de proteínas. La decodificación de una molécula a otra es realizada por proteínas y ARN específicos. Debido a que la información almacenada en el ADN es tan central para la función celular, tiene sentido intuitivo que la célula haga copias de ARNm de esta información para la síntesis de proteínas, al tiempo que mantiene el ADN en sí intacto y protegido.
Resulta que el dogma central no siempre es cierto. No discutiremos aquí las excepciones, sin embargo.

Estructura de aminoácidos
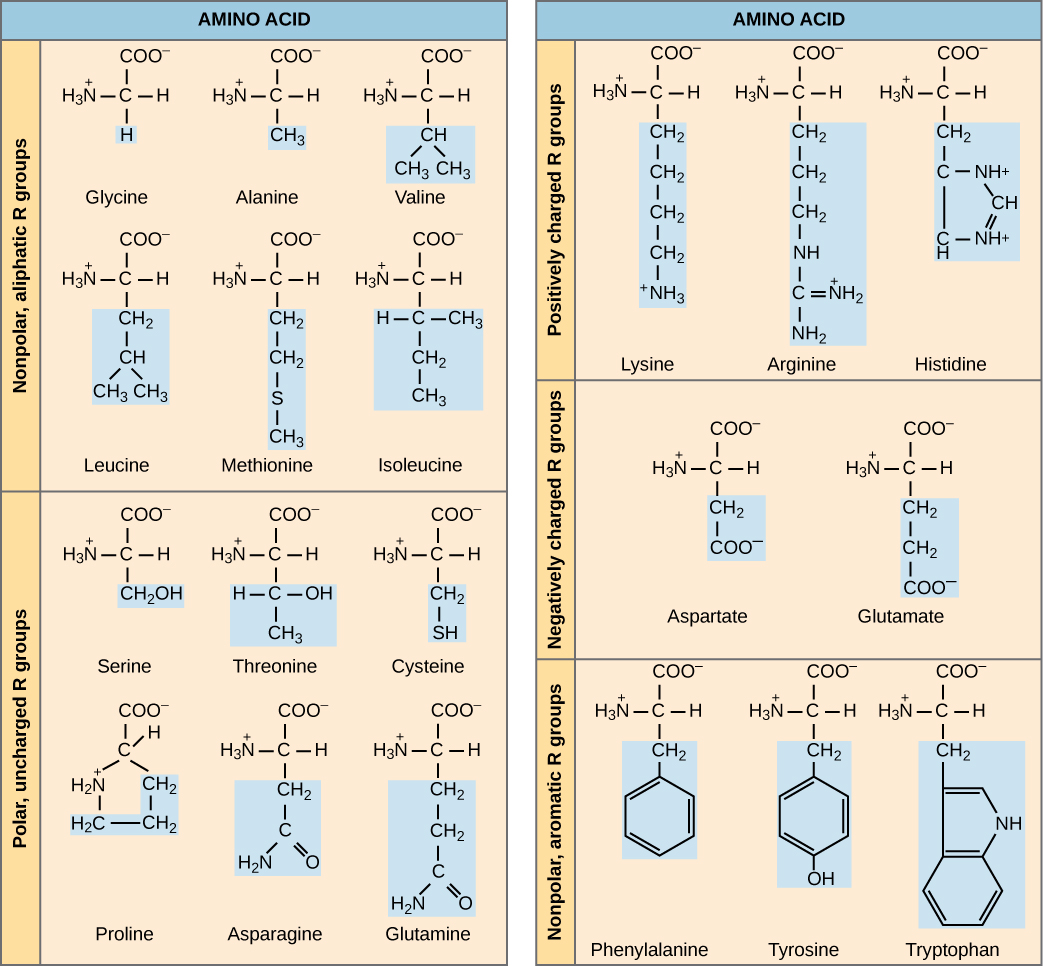
Las secuencias proteicas constan de 20 aminoácidos que ocurren comúnmente (Figura\(\PageIndex{2}\)); por lo tanto, se puede decir que el alfabeto proteico consta de 20 letras. Diferentes aminoácidos tienen diferentes químicas (como ácido versus básico, o polar y no polar) y diferentes restricciones estructurales. La variación en la secuencia de aminoácidos da lugar a una enorme variación en la estructura y función de la proteína.
Código Genético
Cada aminoácido se define por una secuencia de tres nucleótidos llamada codón triplete. La relación entre un codón nucleotídico y su aminoácido correspondiente se denomina código genético. Dados los diferentes números de “letras” en el ARNm (4 — A, U, C, G) y los “alfabetos” de proteínas (20 aminoácidos diferentes) un nucleótido no podía corresponder a un aminoácido. Los dobletes de nucleótidos tampoco serían suficientes para especificar cada aminoácido porque solo hay 16 combinaciones posibles de dos nucleótidos (4 2). En contraste, hay 64 posibles tripletes de nucleótidos (4 3), que es mucho más que el número de aminoácidos. Los científicos teorizaron que los aminoácidos estaban codificados por tripletes de nucleótidos y que el código genético era degenerado. En otras palabras, un aminoácido dado podría estar codificado por más de un triplete de nucleótidos (Figura\(\PageIndex{3}\)). Estos tripletes de nucleótidos se denominan codones.
El mismo codón siempre especificará la inserción de un aminoácido específico. El gráfico que se ve en la Figura\(\PageIndex{3}\) puede usarse para traducir una secuencia de ARNm en una secuencia de aminoácidos. Por ejemplo, el codón UUU siempre provocará la inserción del aminoácido fenilalanina (Phe), mientras que el codón UUA provocará la inserción de leucina (Leu).

Cada conjunto de tres bases (un codón) provoca la inserción de un aminoácido específico en la proteína en crecimiento. Esto significa que la inserción de uno o dos nucleótidos puede cambiar completamente el “marco de lectura” del triplete, alterando así el mensaje para cada aminoácido posterior (Figura\(\PageIndex{4}\)). Aunque la inserción de tres nucleótidos provoca la inserción de un aminoácido adicional durante la traducción, se mantiene la integridad del resto de la proteína.

Tres de los 64 codones terminan la síntesis de proteínas y liberan el polipéptido de la maquinaria de traducción. A estos trillizos se les llama codones de parada. Otro codón, AUG, también tiene una función especial. Además de especificar el aminoácido metionina, también sirve como codón de inicio para iniciar la traducción. El marco de lectura para la traducción se establece mediante el codón de inicio AUG cerca del extremo 5' del ARNm. El código genético es universal. Con algunas excepciones, prácticamente todas las especies utilizan el mismo código genético para la síntesis de proteínas, lo que es una poderosa evidencia de que toda la vida en la Tierra comparte un origen común.
Referencias
A menos que se indique lo contrario, las imágenes de esta página están bajo licencia CC-BY 4.0 de OpenStax.
OpenStax, Biología. OpenStax CNX. enero 2, 2017 https://cnx.org/contents/GFy_h8cu@10...fig-ch15_01_05

