15.5: Traducción
- Page ID
- 58472
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
La síntesis de proteínas consume más energía celular que cualquier otro proceso metabólico. A su vez, las proteínas representan más masa que cualquier otro componente de los organismos vivos (con la excepción del agua), y las proteínas realizan prácticamente todas las funciones de una célula. El proceso de traducción, o síntesis de proteínas, implica la decodificación de un mensaje de ARNm en un producto polipeptídico (proteína). Los aminoácidos están unidos covalentemente por enlaces peptídicos en longitudes que van desde aproximadamente 50 residuos de aminoácidos hasta más de 1,000. Cada aminoácido individual tiene un grupo amino (NH 2) y un grupo carboxilo (COOH). Los polipéptidos se forman cuando el grupo amino de un aminoácido forma un enlace amida (es decir, péptido) con el grupo carboxilo de otro aminoácido (Figura\(\PageIndex{1}\)). Esta reacción es catalizada por ribosomas y genera una molécula de agua.
Recuerda que hay 20 aminoácidos diferentes que se usan comúnmente. En la Figura\(\PageIndex{1}\), R' representa la parte del aminoácido que es diferente entre estas 20 estructuras.

La maquinaria de síntesis de proteínas
Además del molde de ARNm, muchas otras moléculas contribuyen al proceso de traducción y las estructuras y funciones generales de la maquinaria de síntesis de proteínas son comparables de bacterias a células humanas. La traducción requiere la entrada de un molde de ARNm, ribosomas, ARNt y diversos factores enzimáticos (Figura\(\PageIndex{2}\)).
Ribosomas
Incluso antes de que se traduzca un ARNm, una célula debe invertir energía para construir cada uno de sus ribosomas. Los ribosomas son la parte de la célula que lee la información en la molécula de ARNm y une los aminoácidos en el orden correcto. Un ribosoma es una macromolécula muy grande y compleja compuesta por ARNs estructurales y catalíticos, y muchos polipéptidos distintos. En eucariotas, el nucleolo está completamente especializado para la síntesis y ensamblaje de ARNr (el componente de ARN que compone los ribosomas).
Los ribosomas están formados por dos subunidades que se unen para la traducción, más bien como un bollo de hamburguesa se junta alrededor de la carne (el ARNm). La subunidad pequeña es responsable de unirse al molde de ARNm, mientras que la subunidad grande se une secuencialmente a ARNt, un tipo de molécula de ARN que lleva aminoácidos a la cadena en crecimiento del polipéptido. Cada molécula de ARNm puede ser traducida simultáneamente por muchos ribosomas, todos sintetizando proteína en la misma dirección: leer el ARNm de 5' a 3' y sintetizar el polipéptido desde el extremo N hasta el extremo C terminal (consulte la Figura\(\PageIndex{1}\) - el extremo N es el extremo del aminoácido con el Nitrógeno; el extremo C es el final con el Carbono).
Los ribosomas existen en el citoplasma en procariotas y en el citoplasma y retículo endoplásmico rugoso en eucariotas. Las mitocondrias y los cloroplastos también tienen sus propios ribosomas en la matriz y el estroma, que se parecen más a los ribosomas procariotas (y tienen sensibilidades similares a los medicamentos) que los ribosomas justo fuera de sus membranas externas en el citoplasma.

ARNt
Dependiendo de la especie, existen 40 a 60 tipos de ARNt en el citoplasma. Sirviendo como adaptadores, los ARNt específicos se unen a secuencias en el molde de ARNm y agregan el aminoácido correspondiente a la cadena polipeptídica. Por lo tanto, los ARNt son las moléculas que realmente “traducen” el lenguaje del ARN al lenguaje de las proteínas.
Cada ARNt está compuesto por una molécula de ARN lineal que se pliega en una forma compleja (Figura\(\PageIndex{3}\)). En un extremo del ARNt se encuentra un anticodón, que reconoce y pares de bases con uno de los codones de ARNm. En el otro extremo, se une un aminoácido específico. De los 64 posibles codones de ARNm, o combinaciones de tripletes de A, U, G y C, tres especifican la terminación de la síntesis de proteínas y 61 especifican la adición de aminoácidos a la cadena polipeptídica. De estos 61, un codón (AUG) también codifica el inicio de la traducción. Cada anticodón de ARNt puede emparejarse con uno de los codones de ARNm y agregar un aminoácido o terminar la traducción, de acuerdo con el código genético. Por ejemplo, si la secuencia CUA ocurriera sobre un molde de ARNm en el marco de lectura adecuado, se uniría a un ARNt que expresara la secuencia complementaria, GAU, que estaría unida al aminoácido leucina.

Aminoacil ARNt sintetasas
Para que cada ARNt funcione, debe tener su aminoácido específico unido a él. En el proceso de “carga” de ARNt, cada molécula de ARNt está unida a su aminoácido correcto por un grupo de enzimas llamadas aminoacil ARNt sintetasas. Al menos un tipo de aminoacil ARNt sintetasa existe para cada uno de los 20 aminoácidos; el número exacto de aminoacil ARNt sintetasas varía según la especie. Estas enzimas utilizan la energía del ATP para energizar un aminoácido específico, que luego se transfiere al ARNt. De esta manera, las moléculas de ARNt pueden usarse una y otra vez, pero cada ARNt siempre lleva el mismo aminoácido debido a la especificidad de las enzimas aminoacil ARNt sintetasa.
El mecanismo de síntesis de proteínas
Al igual que con la síntesis de ARNm, la síntesis de proteínas se puede dividir en tres fases: iniciación, elongación y terminación. El proceso de traducción es similar en procariotas y eucariotas. Aquí exploraremos cómo se produce la traducción en E. coli, un procariota representativo, y especificaremos cualquier diferencia entre la traducción procariota y eucariota.
Iniciación de la Traducción
La síntesis de proteínas comienza con la formación de un complejo de iniciación. En E. coli, este complejo involucra la subunidad ribosómica pequeña, el molde de ARNm, tres factores de iniciación (IF-1, IF-2 e IF-3) y un ARNt iniciador especial, llamado ARNt F Met. El ARNt iniciador interactúa con el codón de inicio AUG, se une a un aminoácido de metionina formilado llamado fMET, y también puede unirse a IF-2. La metionina formilada es insertada por FMET-ARNt F Met al inicio de cada cadena polipeptídica sintetizada por E. coli, pero generalmente se recorta después de completar la traducción. Cuando se encuentra un AUG en marco durante el alargamiento de la traducción, se inserta una metionina no formilada por un Met-ARNt Met regular.
En el ARNm de E. coli, una secuencia aguas arriba del primer codón AUG, llamada secuencia Shine-Dalgarno (AGGAGG), interactúa con las moléculas de ARNr que componen el ribosoma. Esta interacción ancla la subunidad ribosómica pequeña en la ubicación correcta en el molde de ARNm. El trifosfato de guanosina (GTP), que es un nucleótido trifosfato de purina, actúa como fuente de energía durante la traducción, tanto al inicio del alargamiento como durante la translocación del ribosoma.
En eucariotas, se forma un complejo de iniciación similar, que comprende ARNm, la subunidad ribosómica pequeña, IF y nucleósidos trifosfatos (GTP y ATP). El ARNt iniciador cargado, llamado Met-ARNt i, no se une a fMet en eucariotas, pero es distinto de otros Met-ARNt en que puede unirse a IF. Al igual que en E. coli, se inserta un aminoácido metionina “normal” cuando el ribosoma se encuentra con codones AUG en marco.
En lugar de depositar en la secuencia de Shine-Dalgarno, el complejo de iniciación eucariota reconoce el casquete de 7-metilguanosina en el extremo 5' del ARNm. Una proteína de unión al capuchón (CBP) y varios otros IF ayudan al movimiento del ribosoma a la capota 5'. Una vez en la tapa, el complejo de iniciación rastrea a lo largo del ARNm en la dirección 5' a 3', buscando el codón de inicio AUG. Muchos ARNm eucariotas se traducen desde el primer AUG, pero no siempre es así. La secuencia de bases alrededor de cada AUG ayuda a determinar si ese AUG se utilizará como codón de inicio.
Una vez que se identifica el AUG apropiado, la subunidad ribosómica grande se une al complejo de Met-ARNt i, ARNm y la subunidad ribosómica pequeña. Este paso completa el inicio de la traducción en eucariotas.
Resumen: Tanto en procariotas como en eucariotas, la subunidad ribosómica pequeña se une al iniciador especial ARNt de metionina. Con la ayuda de varios otros factores, este complejo identifica el codón de inicio (AUG) basado en la secuencia de nucleótidos cercanos (Figura\(\PageIndex{4}\), diagrama superior). Luego, la subunidad ribosómica grande se une (Figura\(\PageIndex{4}\), diagrama medio).
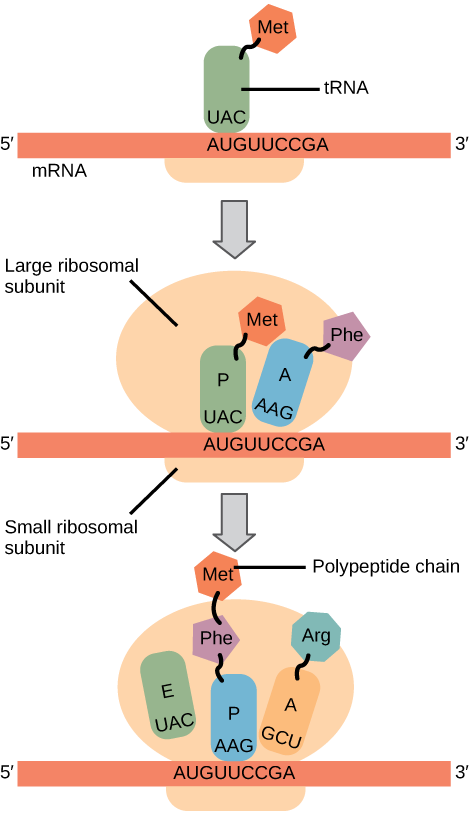
Translación, Alargamiento y Terminación
En procariotas y eucariotas, los fundamentos del alargamiento son los mismos. La subunidad ribosómica grande consta de tres compartimentos: el sitio A (aminoacilo) se une a los ARNt de aminoacilo cargados entrantes. El sitio P (peptidilo) se une a ARNt cargados que portan aminoácidos que han formado enlaces peptídicos con la cadena polipeptídica en crecimiento pero que aún no se han disociado de su ARNt correspondiente. El sitio E (salida) libera ARNt disociados para que puedan recargarse con aminoácidos libres. Hay una excepción a esta línea de ensamblaje de ARNt: en E. coli, FMET-ARNt F Met es capaz de ingresar directamente al sitio P sin ingresar primero al sitio A. De manera similar, el Met-ARNt i eucariota, con ayuda de otras proteínas del complejo de iniciación, se une directamente al sitio P. En ambos casos, esto crea un complejo de iniciación con un sitio A libre listo para aceptar el ARNt correspondiente al primer codón después del AUG.
Durante la elongación de la traducción, el molde de ARNm proporciona especificidad. A medida que el ribosoma se mueve a lo largo del ARNm, cada codón de ARNm entra en registro y se asegura la unión específica con el anticodón de ARNt cargado correspondiente. Si el ARNm no estuviera presente en el complejo de elongación, el ribosoma se uniría a los ARNt de forma no específica y se produciría una proteína sin sentido.
El alargamiento procede con los ARNt cargados que ingresan al sitio A y luego se desplazan al sitio P seguido del sitio E con cada “etapa” de codón único del ribosoma (Figura\(\PageIndex{4}\), diagrama inferior). Los pasos ribosómicos son inducidos por cambios conformacionales que hacen avanzar el ribosoma por tres bases en la dirección 3'. La energía para cada paso del ribosoma es donada por un factor de elongación que hidroliza GTP. Se forman enlaces peptídicos entre el grupo amino del aminoácido unido al ARNt del sitio A y el grupo carboxilo del aminoácido unido al ARNt del sitio P. La formación de cada enlace peptídico es catalizada por peptidil-transferasa, una enzima basada en ARN que se integra en la subunidad ribosómica grande. La energía para cada formación de enlaces peptídicos se deriva de la hidrólisis de GTP, la cual es catalizada por un factor de elongación separado. El aminoácido unido al ARNt del sitio P también está unido a la cadena polipeptídica en crecimiento. A medida que el ribosoma atraviesa el ARNm, el antiguo ARNt del sitio P entra en el sitio E, se separa del aminoácido y se expulsa (Figura\(\PageIndex{4}\)). Sorprendentemente, el aparato de traducción de E. coli tarda solo 0.05 segundos en agregar cada aminoácido, lo que significa que una proteína de 200 aminoácidos se puede traducir en solo 10 segundos.

Referencias
A menos que se indique lo contrario, las imágenes de esta página están bajo licencia CC-BY 4.0 de OpenStax.
OpenStax, Biología. OpenStax CNX. enero 2, 2017 http://cnx.org/contents/s8Hh0oOc@9.10:FUH9XUkW@6/Translation

