16.4: Regulación Postranscripcional Eucariota
- Page ID
- 58409
Después de transcribir el ARN, debe procesarse en una forma madura antes de que pueda comenzar la traducción. Este procesamiento después de que se haya transcrito una molécula de ARN, pero antes de que se traduzca en una proteína, se denomina modificación postranscripcional. Al igual que con las etapas epigenética y transcripcional del procesamiento, esta etapa postranscripcional también se puede regular para controlar la expresión génica en la célula. Si el ARN no se procesa, se traslada o se traduce, entonces no se sintetizará ninguna proteína.
Empalme de ARN alternativo
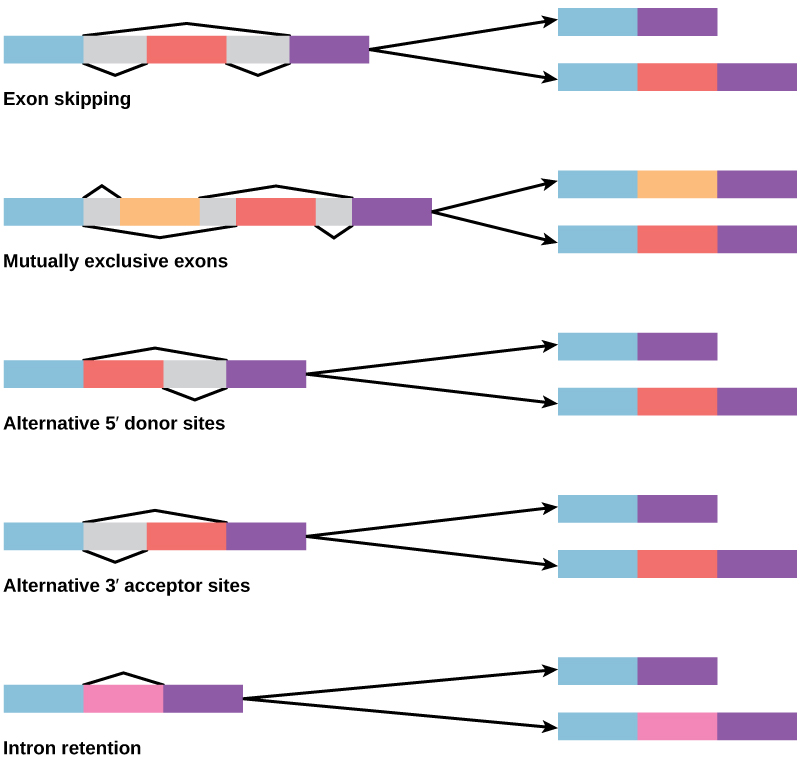
En la década de 1970, se observaron por primera vez genes que exhibieron corte y empalme de ARN alternativo. El empalme alternativo de ARN es un mecanismo que permite producir diferentes productos proteicos a partir de un gen cuando diferentes combinaciones de intrones (y a veces exones) se eliminan del transcrito (Figura\(\PageIndex{1}\)). Este empalme alternativo puede ser fortuito, pero más a menudo se controla y actúa como un mecanismo de regulación génica, con la frecuencia de diferentes alternativas de corte y empalme controladas por la célula como una forma de controlar la producción de diferentes productos proteicos en diferentes células, o en diferentes etapas de desarrollo. Ahora se entiende que el empalme alternativo es un mecanismo común de regulación génica en eucariotas; según una estimación, el 70% de los genes en humanos se expresan como múltiples proteínas a través del corte y empalme alternativo.
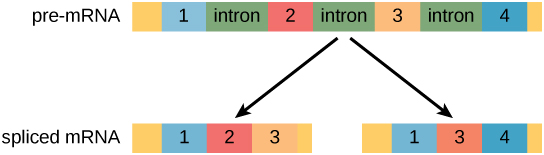
¿Cómo podría evolucionar el empalme alternativo? Los intrones tienen una secuencia de reconocimiento inicial y final, y es fácil imaginar el fracaso del mecanismo de empalme para identificar el final de un intrón y encontrar el final del siguiente intrón, eliminando así dos intrones y el exón intermedio. De hecho, existen mecanismos para prevenir dicho salto de exones, pero es probable que las mutaciones conduzcan a su fracaso. Tales “errores” probablemente producirían una proteína no funcional. De hecho, la causa de muchas enfermedades genéticas es el empalme alternativo en lugar de mutaciones en una secuencia. Sin embargo, el corte y empalme alternativo crearía una variante proteica sin la pérdida de la proteína original, abriendo posibilidades de adaptación de la nueva variante a nuevas funciones. La duplicación génica ha jugado un papel importante en la evolución de nuevas funciones de manera similar, al proporcionar genes que pueden evolucionar sin eliminar la proteína funcional original.
Control de la estabilidad del ARN
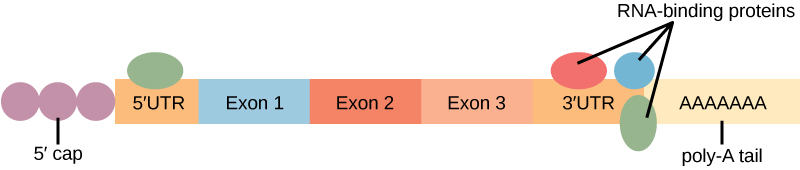
Antes de que el ARNm abandone el núcleo, se le dan dos “tapas” protectoras que impiden que el extremo de la hebra se degrada durante su recorrido. La caperuza 5', que se coloca en el extremo 5' del ARNm, suele estar compuesta por una molécula de trifosfato de guanosina metilada (GTP). La cola poli-A, que está unida al extremo 3', suele estar compuesta por una serie de nucleótidos de adenina. Una vez que el ARN se transporta al citoplasma, se puede controlar el tiempo que el ARN permanece allí. Cada molécula de ARN tiene una vida útil definida y se descompone a una velocidad específica. Esta tasa de descomposición puede influir en la cantidad de proteína que hay en la célula. Si el ARN se descompone más rápidamente, la traducción tiene menos tiempo para ocurrir, por lo que se producirá menos proteína. Por el contrario, si el ARN se descompone menos rápidamente, se producirán más proteínas. Esta tasa de descomposición se conoce como la estabilidad del ARN. Si el ARN es estable, se detectará por periodos de tiempo más largos en el citoplasma. La unión de proteínas al ARN puede influir en su estabilidad (Figura\(\PageIndex{3}\)).
Estabilidad de ARN y microARN
Además de las proteínas que se unen y controlan (aumentan o disminuyen) la estabilidad del ARN, otros elementos llamados microARN pueden unirse a la molécula de ARN. Estos microARN, o miARN, son moléculas de ARN cortas que tienen solo 21 a 24 nucleótidos de longitud. Los miARN se hacen en el núcleo como pre-miARN más largos. Estos pre-miRNAs son cortados en miRNAs maduros por una proteína llamada Dicer. Juntos, los miARN y un gran complejo proteico llamado RISC destruyen rápidamente la molécula de ARN.
Referencias
A menos que se indique lo contrario, las imágenes de esta página están bajo licencia CC-BY 4.0 de OpenStax.
OpenStax, Conceptos de Biología. OpenStax CNX. 3 de enero de 2017. https://cnx.org/contents/GFy_h8cu@10...transcriptiona

