16.5: Regulación Traslacional y Postraduccional Eucariota
- Page ID
- 58423
Después de que el ARN ha sido transportado al citoplasma, se traduce en proteína. El control de este proceso depende en gran medida de la molécula de ARN. Como se discutió anteriormente, la estabilidad del ARN tendrá un gran impacto en su traducción en una proteína. A medida que cambia la estabilidad, también cambia la cantidad de tiempo que está disponible para la traducción.
El complejo de iniciación y la tasa de traducción
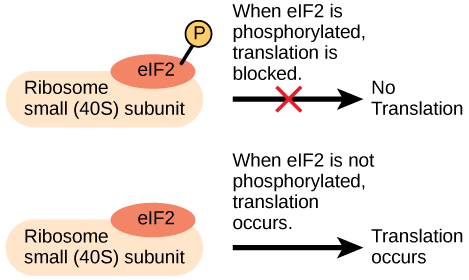
Al igual que la transcripción, la traducción está controlada por proteínas que se unen e inician el proceso. En la traducción, el complejo que se ensambla para iniciar el proceso se conoce como el complejo de iniciación. La regulación de la formación de este complejo puede aumentar o disminuir las tasas de traducción (Figura\(\PageIndex{1}\)).
Modificaciones químicas, actividad proteica y longevidad
Las proteínas pueden modificarse químicamente con la adición de grupos que incluyen grupos metilo, fosfato, acetilo y ubiquitina. La adición o eliminación de estos grupos de las proteínas regula su actividad o el tiempo que existen en la célula. En ocasiones, estas modificaciones pueden regular dónde se encuentra una proteína en la célula, por ejemplo, en el núcleo, el citoplasma o unida a la membrana plasmática.
Las modificaciones químicas ocurren en respuesta a estímulos externos como el estrés, la falta de nutrientes, el calor o la exposición a la luz ultravioleta. Estos cambios pueden alterar la accesibilidad epigenética, la transcripción, la estabilidad del ARNm o la traducción, todo lo que resulta en cambios en la expresión de varios genes. Esta es una manera eficiente para que la célula cambie rápidamente los niveles de proteínas específicas en respuesta al ambiente. Debido a que las proteínas están involucradas en cada etapa de la regulación génica, la fosforilación de una proteína (dependiendo de la proteína que se modifique) puede alterar la accesibilidad al cromosoma, puede alterar la traducción (alterando la unión o función del factor de transcripción), puede cambiar el transporte nuclear (al influir modificaciones al complejo de poro nuclear), puede alterar la estabilidad del ARN (al unirse o no unirse al ARN para regular su estabilidad), puede modificar la traducción (aumentar o disminuir), o puede cambiar modificaciones postraduccionales (agregar o eliminar fosfatos u otras modificaciones químicas).
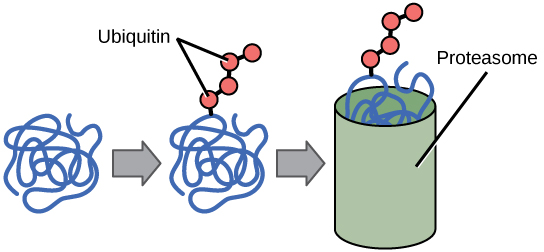
La adición de un grupo ubiquitina a una proteína marca esa proteína para su degradación. La ubiquitina actúa como una bandera que indica que la vida útil de la proteína es completa. Estas proteínas se trasladan al proteasoma, un orgánulo que funciona para eliminar proteínas, para ser degradadas (Figura\(\PageIndex{2}\)). Una forma de controlar la expresión génica, por lo tanto, es alterar la longevidad de la proteína.
Referencias
A menos que se indique lo contrario, las imágenes de esta página están bajo licencia CC-BY 4.0 de OpenStax.
OpenStax, Conceptos de Biología. OpenStax CNX. mayo 18, 2016 http://cnx.org/contents/b3c1e1d2-839...9a8aafbdd@9.10

