20.5: Aplicando Genómica
- Page ID
- 58264
La introducción de proyectos de secuenciación de ADN y secuenciación del genoma completo, particularmente el Proyecto Genoma Humano, ha ampliado la aplicabilidad de la información de secuencias de ADN. La genómica ahora se está utilizando en una amplia variedad de campos, como la metagenómica, la farmacogenómica y la genómica mitocondrial. La aplicación más comúnmente conocida de la genómica es comprender y encontrar curas para enfermedades.
Predecir el riesgo de enfermedad a nivel individual
La predicción del riesgo de enfermedad implica el tamizaje e identificación de individuos actualmente sanos mediante el análisis del genoma a nivel individual. Se puede recomendar la intervención con cambios de estilo de vida y medicamentos antes del inicio de la enfermedad Sin embargo, este enfoque es más aplicable cuando el problema surge de una sola mutación génica. Dichos defectos solo representan alrededor del 5 por ciento de las enfermedades que se encuentran en los países desarrollados. La mayoría de las enfermedades comunes, como las cardiopatías, son multifactoriales o poligénicas, lo que hace referencia a una característica fenotípica que está determinada por dos o más genes, y también factores ambientales como la dieta. En abril de 2010, científicos de la Universidad de Stanford publicaron el análisis del genoma de un individuo sano (Stephen Quake, científico de la Universidad de Stanford, que tenía su genoma secuenciado); el análisis predijo su propensión a adquirir diversas enfermedades. Se realizó una evaluación de riesgo para analizar el porcentaje de riesgo de Quake para 55 padecimientos médicos diferentes. Se encontró una rara mutación genética que demostró que estaba en riesgo de sufrir un ataque cardíaco repentino. También se predijo que tendría un riesgo de 23 por ciento de desarrollar cáncer de próstata y un 1.4 por ciento de riesgo de desarrollar la enfermedad de Alzheimer. Los científicos utilizaron bases de datos y varias publicaciones para analizar los datos genómicos. A pesar de que la secuenciación genómica es cada vez más asequible y las herramientas analíticas son cada vez más confiables, quedan por abordar los problemas éticos relacionados con el análisis genómico a nivel poblacional. Por ejemplo, ¿podrían utilizarse dichos datos legítimamente para cobrar más o menos por los seguros o para afectar las calificaciones crediticias?
Estudios de Asociación de todo el genoma
Desde 2005, ha sido posible realizar un tipo de estudio llamado estudio de asociación de todo el genoma, o GWAS. Un GWAS es un método que identifica diferencias entre individuos en polimorfismos de un solo nucleótido (SNP) que pueden estar involucrados en causar enfermedades. El método es particularmente adecuado para enfermedades que pueden verse afectadas por uno o muchos cambios genéticos a lo largo del genoma. Es muy difícil identificar los genes involucrados en dicha enfermedad utilizando información de antecedentes familiares. El método GWAS se basa en una base de datos genética que ha estado en desarrollo desde 2002 llamada International HapMap Project. El Proyecto HapMap secuenció los genomas de varios cientos de individuos de todo el mundo e identificó grupos de SNP. Los grupos incluyen SNP que se encuentran cerca unos de otros en los cromosomas por lo que tienden a permanecer juntos a través de la recombinación. El hecho de que el grupo permanezca unido significa que identificar un marcador SNP es todo lo que se necesita para identificar todos los SNP en el grupo. Hay varios millones de SNP identificados, pero identificarlos en otros individuos que no han tenido su genoma completo secuenciado es mucho más fácil porque solo es necesario identificar los SNP marcadores.
En un diseño común para un GWAS, se eligen dos grupos de individuos; un grupo tiene la enfermedad y el otro no. Los individuos de cada grupo se emparejan en otras características para reducir el efecto de las variables de confusión que causan diferencias entre los dos grupos. Por ejemplo, los genotipos pueden diferir porque los dos grupos son en su mayoría tomados de diferentes partes del mundo. Una vez elegidos los individuos, y típicamente sus números son mil o más para que el estudio funcione, se obtienen muestras de su ADN. El ADN se analiza mediante sistemas automatizados para identificar grandes diferencias en el porcentaje de SNP particulares entre los dos grupos. A menudo, el estudio examina un millón o más de SNP en el ADN. Los resultados de GWAS se pueden utilizar de dos maneras: las diferencias genéticas pueden usarse como marcadores de susceptibilidad a la enfermedad en individuos no diagnosticados, y los genes particulares identificados pueden ser objetivos para la investigación de la vía molecular de la enfermedad y posibles terapias. Una rama del descubrimiento de asociaciones genéticas con la enfermedad ha sido la formación de empresas que proporcionan la llamada “genómica personal” que identificarán los niveles de riesgo para diversas enfermedades a partir del complemento SNP de un individuo. La ciencia detrás de estos servicios es polémica.
Debido a que GWAS busca asociaciones entre genes y enfermedades, estos estudios proporcionan datos para otras investigaciones sobre causas, en lugar de responder preguntas específicas por sí mismas. Una asociación entre una diferencia génica y una enfermedad no significa necesariamente que exista una relación causa-efecto. Sin embargo, algunos estudios han proporcionado información útil sobre las causas genéticas de las enfermedades. Por ejemplo, tres estudios diferentes en 2005 identificaron un gen para una proteína involucrada en la regulación de la inflamación en el cuerpo que se asocia con una ceguera causante de enfermedad llamada degeneración macular relacionada con la edad. Esto abrió nuevas posibilidades de investigación sobre la causa de esta enfermedad. Se ha identificado un gran número de genes asociados a la enfermedad de Crohn utilizando GWAS, y algunos de estos han sugerido nuevos mecanismos hipotéticos para la causa de la enfermedad.
Farmacogenómica
La farmacogenómica implica evaluar la efectividad y seguridad de los medicamentos a partir de la información de la secuencia genómica de un individuo. La información personal de la secuencia genómica se puede utilizar para recetar medicamentos que serán más efectivos y menos tóxicos sobre la base del genotipo del paciente individual. El estudio de los cambios en la expresión génica podría proporcionar información sobre el perfil de transcripción génica en presencia del fármaco, el cual puede ser utilizado como un indicador temprano del potencial de efectos tóxicos. Por ejemplo, los genes involucrados en el crecimiento celular y la muerte celular controlada, cuando se alteran, podrían conducir al crecimiento de células cancerosas. Los estudios de todo el genoma también pueden ayudar a encontrar nuevos genes involucrados en la toxicidad de los medicamentos. Las firmas genéticas pueden no ser completamente precisas, pero se pueden probar más antes de que surjan síntomas patológicos.
Metagenómica
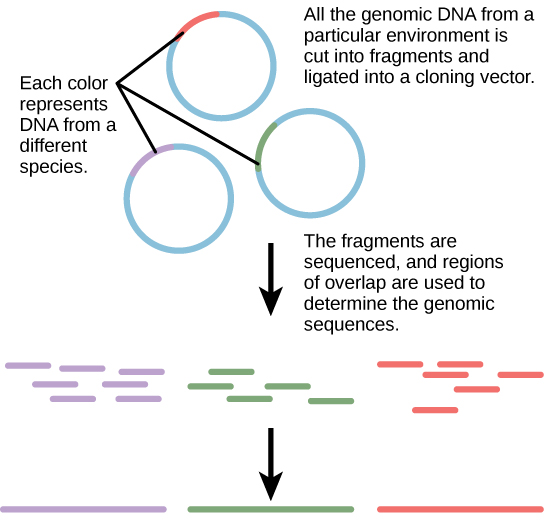
Tradicionalmente, la microbiología se ha enseñado con la idea de que los microorganismos se estudian mejor en condiciones de cultivo puro, lo que implica aislar un solo tipo de célula y cultivarla en el laboratorio. Debido a que los microorganismos pueden pasar por varias generaciones en cuestión de horas, sus perfiles de expresión génica se adaptan muy rápidamente al nuevo entorno de laboratorio. Por otro lado, muchas especies resisten ser cultivadas en aislamiento. La mayoría de los microorganismos no viven como entidades aisladas, sino en comunidades microbianas conocidas como biopelículas. Por todas estas razones, el cultivo puro no siempre es la mejor manera de estudiar microorganismos. La metagenómica es el estudio de los genomas colectivos de múltiples especies que crecen e interactúan en un nicho ambiental. La metagenómica puede ser utilizada para identificar nuevas especies más rápidamente y para analizar el efecto de los contaminantes en el medio ambiente (Figura\(\PageIndex{1}\)). Las técnicas de metagenómica ahora también se pueden aplicar a comunidades de eucariotas superiores, como los peces.
Creación de nuevos biocombustibles
El conocimiento de la genómica de los microorganismos se está utilizando para encontrar mejores formas de aprovechar los biocombustibles de algas y cianobacterias. Las principales fuentes de combustible hoy en día son el carbón, el petróleo, la madera y otros productos vegetales como el etanol. Si bien las plantas son recursos renovables, aún es necesario encontrar más fuentes de energía renovables alternativas para satisfacer las demandas energéticas de nuestra población. El mundo microbiano es uno de los mayores recursos para los genes que codifican nuevas enzimas y producen nuevos compuestos orgánicos, y permanece en gran parte sin explotar. Este vasto recurso genético tiene el potencial de proporcionar nuevas fuentes de biocombustibles (Figura\(\PageIndex{2}\)).

Genómica mitocondrial
Las mitocondrias son orgánulos intracelulares que contienen su propio ADN. El ADN mitocondrial muta a un ritmo rápido y a menudo se usa para estudiar las relaciones evolutivas. Otra característica que hace interesante estudiar el genoma mitocondrial es que en la mayoría de los organismos multicelulares, el ADN mitocondrial se transmite de la madre durante el proceso de fertilización. Por esta razón, la genómica mitocondrial se suele utilizar para rastrear la genealogía.
Genómica en Análisis Forense
La información y las pistas obtenidas de muestras de ADN encontradas en las escenas del crimen han sido utilizadas como evidencia en casos judiciales, y se han utilizado marcadores genéticos en análisis forenses. El análisis genómico también se ha vuelto útil en este campo. En 2001 se publicó el primer uso de la genómica en forense. Fue un esfuerzo colaborativo entre instituciones académicas de investigación y el FBI para resolver los misteriosos casos de ántrax (Figura\(\PageIndex{3}\)) que fue transportado por el Servicio Postal de Estados Unidos. Las bacterias del ántrax se convirtieron en un polvo infeccioso y se enviaron por correo a medios de comunicación y a dos senadores de Estados Unidos. El polvo infectó al personal administrativo y a los trabajadores postales que abrieron o manejaron las cartas. Cinco personas murieron, y 17 se enfermaron por la bacteria. Utilizando la genómica microbiana, los investigadores determinaron que se utilizó una cepa específica de ántrax en todos los envíos; finalmente, la fuente se rastreó hasta un científico en un laboratorio nacional de biodefensa en Maryland.
Genómica en Agricultura
La genómica puede reducir en cierta medida los ensayos y fracasos involucrados en la investigación científica, lo que podría mejorar la calidad y cantidad de los rendimientos de los cultivos en la agricultura (Figura\(\PageIndex{4}\)). Vincular rasgos a genes o firmas génicas ayuda a mejorar la reproducción de cultivos para generar híbridos con las cualidades más deseables. Los científicos utilizan datos genómicos para identificar rasgos deseables, y luego transferir esos rasgos a un organismo diferente para crear un nuevo organismo genéticamente modificado, como se describe en el módulo anterior. Los científicos están descubriendo cómo la genómica puede mejorar la calidad y cantidad de la producción agrícola. Por ejemplo, los científicos podrían usar rasgos deseables para crear un producto útil o mejorar un producto existente, como hacer que un cultivo sensible a la sequía sea más tolerante a la estación seca.

Referencias
A menos que se indique lo contrario, las imágenes de esta página están bajo licencia CC-BY 4.0 de OpenStax.
OpenStax, Biología. OpenStax CNX. mayo 27, 2016 http://cnx.org/contents/s8Hh0oOc@9.10:TE1njgbY@4/Genomics-and-Proteomics

