1.4: Enzimas modificadoras de ADN
- Page ID
- 57204
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Metilasas
Así como el estudio del sistema bacteriano de restricción-modificación ha proporcionado una variedad de endonucleasas específicas, también hay disponibles una variedad de ADN metilasas específicas.
- Las secuencias de reconocimiento de las metilasas son las mismas que las endonucleasas asociadas (por ejemplo, la metilasa EcoR1 reconoce y metila en la secuencia “GAATTC”).
- Todas las metilasas transfieren el grupo metilo de S-adenosilmetionina (SAM) a una base específica en la secuencia de reconocimiento, y SAM es un componente requerido en la reacción de metilación.
- La metilación del ADN generalmente tiene el efecto de proteger el ADN de la endonucleasa de restricción relacionada. Sin embargo, existen metilasas con especificidad mínima. Por ejemplo, la metilasa de Sss I metilará los residuos de citosina en la secuencia 5'... CG... 3'. En este caso, el ADN metilado estará protegido de una amplia variedad de endonucleasas de restricción.
- Algunas endonucleasas de restricción solo cortarán ADN en sus sitios de reconocimiento si el ADN está metilado (por ejemplo, Dpn I).
- Aún otras endonucleasas de restricción cortarán tanto ADN metilado como no metilado en sus secuencias de reconocimiento (por ejemplo, BamH I).
Metilación de dam y dcm
- La metilasa codificada por el gen dam (dam metilasa) transfiere un grupo metilo de SAM a la posición N 6 de la base adenina en la secuencia 5'... GATC... 3'.
- La metilasa codificada por el gen dcm (dcm metilasa) metila la base interna de citosina, en la posición C 5, en las secuencias 5'... CCAGG... 3' y 5'... CCTGG... 3'.
- Casi todas las cepas de E. coli comúnmente utilizadas en la clonación tienen un genotipo dam + dcm +. El punto aquí no es que queramos particularmente que nuestro ADN esté metilado, sino que para hacer un dam-dcm- hospedador alguien tiene que mutar la bacteria y aislar el mutante correcto. Eso al parecer no se ha hecho para muchas cepas bacterianas. Probablemente porque la metilación dam y dcm afecta solo a un pequeño subconjunto de endonucleasas de restricción potenciales
El ADN aislado de cepas dam+dcm+ no se cortará realmente por un subconjunto modesto de endonucleasas de restricción disponibles:
|
|
|
|
|
|---|---|---|---|
| TGATCA |
|
|
|
| GATC |
|
|
|
| ATCGAT |
|
|
|
| TCTAGA |
|
|
|
| TCGA |
|
|
|
| GAAGA |
|
|
|
| GGTGA |
|
|
|
El ADN puede tener que prepararse a partir de cepas de E. coli que son dam-dcm- para ser cortado por estas enzimas.
ADN polimerasas
Se ha caracterizado una amplia variedad de polimerasas y están disponibles comercialmente. Todas las ADN polimerasas comparten dos características generales:
- Añaden nucleótidos al extremo 3'-OH de un cebador
- El orden de los nucleótidos en el polinucleótido naciente está dirigido a molde
.png)
Figura 1.4.1: Replicación de ADN
Además de la actividad polimerasa 5'->3', las polimerasas pueden contener actividad exonucleasa. Esta actividad exonucleasa puede proceder ya sea en la dirección 5'->3', o en la dirección 3'->5'.
- La actividad exonucleasa en la dirección 3'->5' permite a la polimerasa corregir un error si incorpora un nucleótido incorrecto (la llamada "actividad de corrección de errores “). También puede degradar lentamente el extremo 3' del cebador.
- La actividad exonucleasa en la dirección 5'->3' le permitirá degradar cualquier otro cebador hibridado que pueda encontrar. Sin actividad exonucleasa 5'->3', los cebadores obstructores pueden o no ser físicamente deslocados, dependiendo de la polimerasa que se utilice.
Diferentes polimerasas tienen diferentes tasas de error de incorporación errónea y diferentes tasas de polimerización.
|
|
|
|
|
|
|
|---|---|---|---|---|---|
| Actividad exonucleasa 5'->3' |
|
|
|||
| Actividad exonucleasa 3'->5' |
|
|
|
|
|
| Tasa de error (x10 -6) |
|
|
|
|
|
| Desplazamiento del hilo |
|
||||
| Inactivación por calor |
|
|
|
|
Usos de polimerasas
Las diversas actividades de las diferentes polimerasas las prestan a una variedad de aplicaciones. Por ejemplo, las endonucleasas de restricción pueden producir fragmentos de ADN con “voladizos” de nucleótidos 3' o 5'.
- En el caso de los voladizos 5', la actividad polimerasa 5'->3' puede rellenarlos para formar extremos romos.
- En el caso de los voladizos 3', la actividad exonucleasa 3'->5' presente en algunas polimerasas (especialmente la ADN polimerasa T4) puede “masticar” estos extremos para hacer también fragmentos de ADN de extremos romos.
.png)
Figura 1.4.2: Actividad de la polimerasa
“Nick-traducción”
Este método se utiliza para obtener fragmentos de ADN monocatenarios altamente radiomarcada, lo que hace uso de la actividad 5'->3' exonucleasa presente en algunas polimerasas (ADN polimerasa I de E. coli, por ejemplo).
- En este método se “mella” un dúplex de ADN de interés (es decir, se corta una de las hebras; ver DNasa I).
- Luego se agrega ADN pol I junto con nucleótidos radiomarcada. La actividad exonucleasa 5'->3' mastica el extremo 5' en el sitio de “mella” y la actividad polimerasa incorpora los nucleótidos radiomarcados. El polinucleótido resultante estará altamente radiomarcado e hibridará con la secuencia de ADN de interés.
.png)
Figura 1.4.3: Traducción de NICK
- Las polimerasas termoestables tienen la capacidad de permanecer funcionales en rangos de temperatura donde el dúplex de ADN realmente se “fundirá” y se separará. Esto ha permitido el desarrollo de la técnica de “Reacción en Cadena de la Polimerasa” (PCR), la cual ha tenido un profundo impacto en la Biotecnología moderna. Discutiremos este método en una fecha posterior.
- La incorporación de bases didesoxi (es decir, sin grupos hidroxilo en el carbono 2' o 3' del azúcar ribosa) conduce a la terminación de la reacción de la polimerasa. Esto se discutirá con mayor detalle más adelante. Sin embargo, esta terminación de cadena por incorporación de didesoxinucleótidos es la base del método de secuenciación del ADN de Sanger, así como terapias para tratar de inhibir la replicación viral.
Nucleasas
Nucleasa BAL-31
- Esta es una exo nucleasa (comienza en los extremos y trabaja hacia adentro) que degradará los extremos 3' y 5' del ADN bicatenario. No hará escisiones internas (“mellas”), sin embargo, degradará los extremos del ADN en las “mellas” internas existentes (que crean ambos extremos 3' y 5').
- La degradación de los extremos no está coordinada, lo que significa que el producto no es 100% de extremos romos (aunque el dúplex original pueda haber sido romo).
- Tales extremos “rasgados” se pueden hacer romos rellenando y masticando con una polimerasa adecuada (por ejemplo, ADN polimerasa T4). La definición de unidad de 1 unidad es la cantidad de enzima requerida para eliminar 200 pares de bases de cada extremo del ADN dúplex en 10 minutos a 30 °C.
.png)
Figura 1.4.4: Actividad de la nucleasa BAL-31
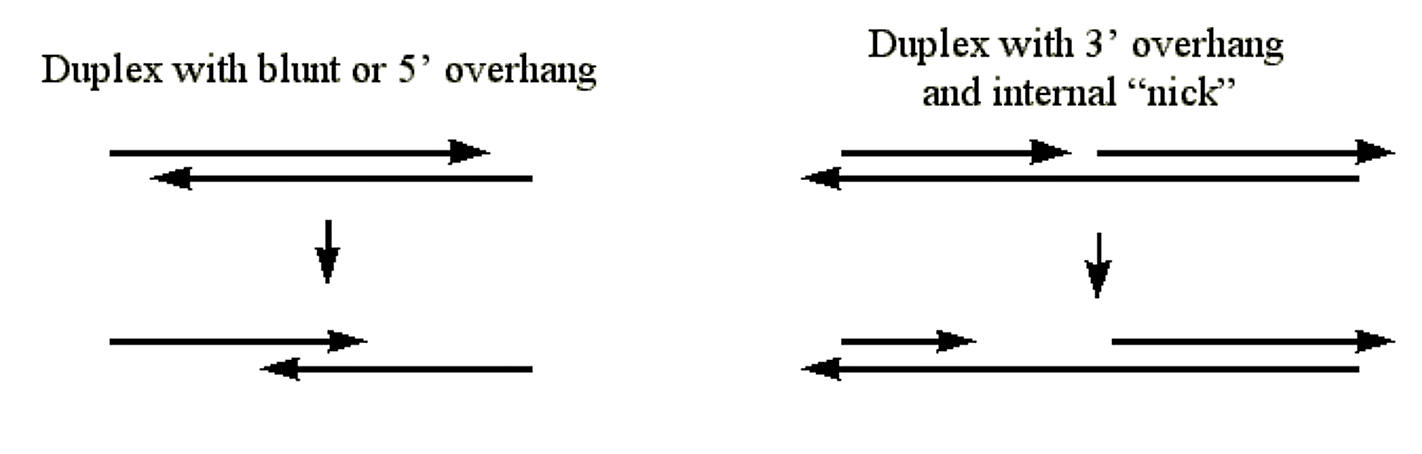
Exonucleasa III
- Cataliza la eliminación por etapas de nucleótidos de los extremos hidroxilo 3' del ADN dúplex.
- La enzima atacará el hidroxilo 3' en el ADN dúplex con extremos romos, con voladizos 5' o con “mellas” internas.
- Dado que se requiere ADN dúplex, la enzima no digiere el extremo 3' del ADN dúplex donde los extremos son sobresalientes 3'.
.png)
Figura 1.4.5: Actividad de la exonucleasa III
Nucleasa de frijol mungo (aislada de brotes de frijol mungo)
- Una endonucleasa específica de ADN y ARN monocatenario que degradará las extensiones monocatenarias de los extremos del ADN y ARN dejando extremos romos.
- Las extensiones monocatenarias pueden ser extensiones 5' o 3'; ambas se eliminan y se deja un dúplex romo.
.png)
Figura 1.4.6: Actividad de nucleasa de frijol mungo
Desoxirribonucleasa I (DNasa I) de pancreasa bovina
- Esta enzima hidroliza cadenas de ADN dúplex o individuales preferentemente en los enlaces fosfodiéster 5' a nucleótidos de pirimidina
- En presencia del ion Mg 2+, la DNasa I ataca cada cadena de forma independiente y produce mellas de manera aleatoria (útil para la traducción de mellas)
- En presencia del ion Mn2+, la DNasa I escinde ambas cadenas de ADN aproximadamente en la misma posición (pero dejando extremos “rasgados”)
Ligasas
- Las ligasas catalizan la formación de un enlace fosfodiéster entre los extremos 5' fosfato y 3' hidroxilo yuxtapuestos de nucleótidos (potencialmente ARN o ADN dependiendo de la ligasa).
- En cierto sentido, son lo opuesto a las endonucleasas de restricción, pero no parecen estar influenciadas por la secuencia local, per se.
- Las ligasas requieren rATP o NAD+ como cofactor, y esto contrasta con las endonucleasas de restricción.
Los siguientes son diferentes tipos de ligasas y sus características.
ADN ligasa T4
- Aislado del bacteriófago T4.
- Ligará los extremos del ADN o ARN dúplex.
- Esta enzima unirá extremos romos así como extremos con extremos sobresalientes cohesivos (complementarios) (ya sea 3' o 5' complementarios sobresalientes).
- Esta enzima también reparará mellas monocatenarias en dúplex de ADN, ARN o dúplex de ADN/ARN. Requiere ATP como cofactor.
Taq ADN ligasa
- Esta ligasa catalizará un enlace fosfodiéster entre dos oligonucleótidos adyacentes que se hibridan con una cadena de ADN complementaria:
.png)
Figura 1.4.7: Actividad de ADN ligasa Taq
- La ligación es eficiente solo si los oligonucleótidos hibridan perfectamente con la cadena molde.
- La enzima es activa a temperaturas relativamente altas (45 - 65 °C). Requiere NAD+ como cofactor.
ARN ligasa T4
- Catalizará la formación de un enlace fosfodiéster entre oligonucleótidos ARN/ARN, oligonucleótidos ARN/ADN u oligonucleótidos ADN/ADN.
- Requiere ATP como cofactor.
- Esta enzima no requiere una cadena molde.
La ARN ligasa T4 se puede usar para una variedad de propósitos, incluyendo la construcción de moléculas híbridas de ARN/ADN.
.png)
Figura 1.4.8: Actividad de ARN ligasa de T4
ADN ligasa (E. coli)
- Catalizará un enlace fosfodiéster entre el ADN dúplex que contiene extremos cohesivos.
- No ligará de manera eficiente los fragmentos de extremos blund.
- Requiere NAD+ como cofactor.
.png)
Figura 1.4.9: Actividad de ADN ligasa (E. Coli)
Polinucleotido quinasa
- Cataliza la transferencia e intercambio de un grupo fosfato desde la posición g de rATP (nucleótido trifosfato de adenina ribosa) hasta el extremo 5' hidroxilo del ADN o ARN bicatenario y monocatenario, y los nucleósidos 3' monofosfatos.
- La enzima también eliminará los grupos 3' fosforilo.
- Los oligonucleótidos que se obtienen de sintetizadores automatizados carecen de un grupo fosfato 5' y, por lo tanto, no pueden ligarse a otros polinucleótidos.
La polinucleótido quinasa T4 se puede usar para fosforilar el extremo 5' de dichos polinucleótidos:
.png)
Figura 1.4.10: Actividad de polinucleótido quinasa T4
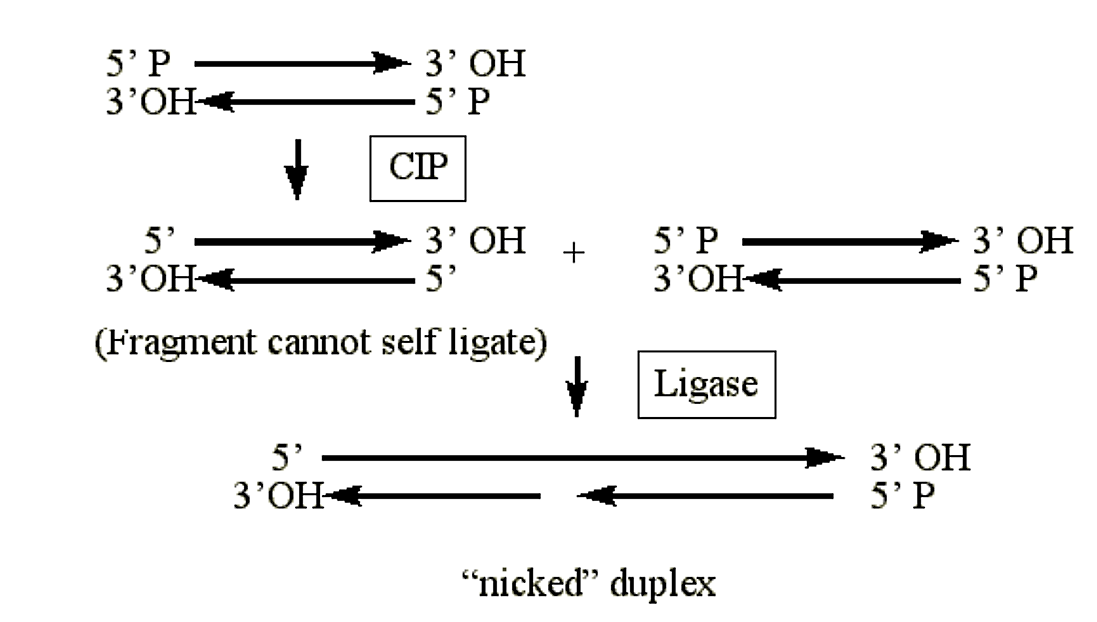
Fosfatasa intestinal de ternero (CIP)
- Cataliza la eliminación de grupos fosfato 5' de ARN, ADN y ribo- y desoxirribo- nucleósidos trifosfatos (p. ej. ATP, rATP).
- El ADN dúplex tratado con CIP no puede autoligarse.
- Los dúplex hemifosforilados se ligarán en una cadena (la cadena fosforilada) y permanecerán “mellados” en la otra.
.png)
Figura 1.4.11: Actividad CIP


