3.1: Electroforesis en gel
- Page ID
- 57237
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)La electroforesis en gel se utiliza para caracterizar una de las propiedades más básicas, la masa molecular, tanto de polinucleótidos como de polipéptidos. También se puede usar electroforesis en gel para determinar: (1) la pureza de estas muestras, (2) heterogeneidad/grado de degradación y (3) composición de subunidades.
ADN
Los materiales de electroforesis en gel más comunes para las moléculas de ADN son la agarosa y la acrilamida.
Geles de agarosa de ADN
La tasa de migración electroforética del ADN a través de geles de agarosa depende de cuatro parámetros principales:
1. El tamaño molecular del ADN. Las moléculas de ADN dúplex lineal viajan a través de geles de agarosa a una velocidad que es inversamente proporcional al log de su peso molecular.
\[M_r \propto 1/log (Mw)\]
Ejemplo: Comparar la masa molecular frente a la tasa de migración esperada:
|
Masa molecular (Da) |
tronco (Molec. Masa) |
1/tronco (Molec. Masa) |
|---|---|---|
|
100,000 |
5.0 |
0.20 |
|
50,000 |
4.7 |
0.21 |
|
10,000 |
4.0 |
0.25 |
|
5,000 |
3.7 |
0.27 |
|
1,000 |
3.0 |
0.33 |
.png)
Figura 3.1.1: Tasa de migración relativa con masa molecular
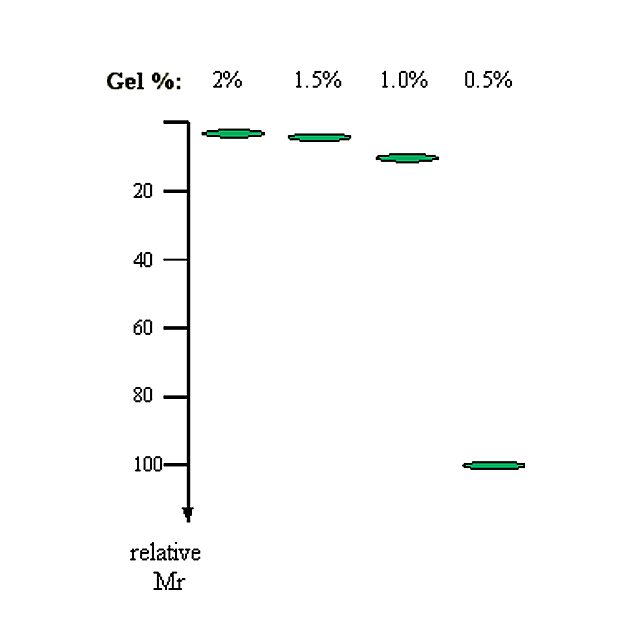
2. La concentración de agarosa. Existe una relación lineal inversa entre el logaritmo de la movilidad electroforética y la concentración de gel.
\[\log (M_r) \propto 1/[gel]\]
|
% Gel |
1/Gel% |
log inv (1/% de gel) (es decir, Mr relativo) |
|---|---|---|
|
2.0 |
0.50 |
3.2 |
|
1.5 |
0.67 |
4.6 |
|
1.0 |
1.00 |
10.0 |
|
0.5 |
2.00 |
100.0 |
.png)
Figura 3.1.2: Tasa de migración relativa con concentración de gel
3. La conformación del ADN.
- ADN circular cerrado (forma I) - típicamente superenrollado
- circular mellado (Forma-II)
- ADN lineal (Forma-III)
Estas diferentes formas del mismo ADN migran a diferentes velocidades a través de un gel de agarosa. Casi siempre la forma lineal (forma III) migra a la velocidad más lenta de las tres formas y el ADN superenrollado (forma I) generalmente migra más rápido.
4. El voltaje aplicado.
- El valor típico para hacer funcionar un gel de agarosa es de 5 voltios por cm (longitud del gel).
|
Agarosa (%) |
Rango de separación de ADN lineal (en kilobases) |
|---|---|
|
0.3 |
60 - 5 |
|
0.6 |
20 - 1 |
|
0.7 |
10 - 0.8 |
|
0.9 |
7 - 0.5 |
|
1.2 |
6 - 0.4 |
|
1.5 |
4 - 0.2 |
|
2.0 |
3 - 0.1 |
- Los geles de agarosa generalmente se vierten y se ejecutan horizontalmente
Finalmente, siendo el ADN una molécula ácida, migra hacia el electrodo cargado positivamente (cátodo).
.png)
Figura 3.1.3: Configuración de electroforesis en gel
Geles de acrilamida de ADN
Los geles de acrilamida son útiles para la separación de pequeños fragmentos de ADN típicamente oligonucleótidos <100 pares de bases. Estos geles suelen ser de baja concentración de acrilamida (<= 6%) y contienen el agente desnaturalizante no iónico Urea (6M). El agente desnaturalizante evita la formación de estructuras secundarias en oligonucleótidos y permite una determinación relativamente precisa de la masa molecular.
Electroforesis en gel para proteínas
La electroforesis en gel de proteínas utiliza casi exclusivamente poliacrilamida. La solución de acrilamida generalmente contiene dos componentes: acrilamida y bis acrilamida. Un valor típico para la relación acrilamida:bis es 19:1. La bis acrilamida es esencialmente un componente reticulante del polímero de acrilamida. La concentración total de acrilamida en el gel afecta la migración de proteínas a través de la matriz (como ocurre con la concentración de agarosa).
Los geles proteicos generalmente se realizan bajo condiciones desnaturalizantes en presencia del detergente dodecilsulfato de sodio (SDS). Las proteínas se desnaturalizan por calor en presencia de SDS. El SDS se une, a través de interacciones hidrofóbicas, a las proteínas en una cantidad aproximadamente proporcional al tamaño de la proteína. Debido a la naturaleza cargada de la molécula SDS las proteínas tienen así una relación carga/masa algo constante y migran a través del gel a una velocidad proporcional a su masa molecular, Las proteínas migran hacia el ánodo.
|
Acrilmida (%) |
Rango de separación de Polipéptidos (en kilodaltones) |
|---|---|
|
8 |
200 - 25 |
|
10 |
100 - 15 |
|
12.5 |
70 - 10 |
|
15 |
60 - 6 |
|
20 |
40 - 4 |
Dado que el tratamiento con SDS disociará los complejos proteicos no covalentes, pueden exhibir una masa molecular mucho menor de lo esperado en la electroforesis en gel de poliacrilamida SDS (SDS PAGE). Los geles de proteína PAGE generalmente se polimerizan entre dos placas de vidrio y se ejecutan en dirección vertical.
.png)
Figura 3.1.4: Efecto del tratamiento con SDS
La PAGE también se puede ejecutar en presencia de agentes reductores, tales como b-mercaptoetanol (BME). El BME es un agente reductor que reducirá cualquier enlace disulfuro (por ejemplo, como existe entre algunos pares de residuos de cisteína en una proteína). Esto ayuda a eliminar la estructura secundaria residual en la proteína tratada con SDS, pero también puede permitir la separación de fragmentos de polipéptidos entre sí (es decir, su interacción covalente estaba compuesta completamente por uno o más enlaces disulfuro). Así, una proteína aparentemente única puede exhibir un conjunto de pequeños fragmentos bajo condiciones de PAGE reductoras.
Manchas para ADN
Etidio
El método más conveniente para visualizar el ADN en electroforesis en gel es la tinción con el colorante fluorescente bromuro de etidio. Este compuesto contiene un grupo plano que se intercala entre las bases apiladas del ADN. La orientación y proximidad del etidio con las bases apiladas hace que el tinte muestre una florescencia incrementada en comparación con el tinte libre (en solución). La radiación U.V. a 254 nm es absorbida por el ADN y transmitida al colorante unido y la energía es reemitida a 590 nm en la región rojo-naranja del espectro.
.png)
Figura 3.1.5: Etidio
El bromuro de etidio generalmente se prepara como una solución madre de 10 mg/ml en agua, se almacena a temperatura ambiente y se protege de la luz. El colorante generalmente se incorpora al gel y al tampón de funcionamiento, o a la inversa, el gel se tiñe después de correr remojándolo en una solución de bromuro de etidio (0.5 pg/mL durante 30 min). La mancha se visualiza irradiando con una fuente de luz UV (es decir, usando un transiluminador) y fotgrafiando con película polaroide. La sensibilidad habitual de detección es mejor que 0.1 ug de ADN.
Debido a que el etidio es un agente intercalante de ADN, es un mutágeno potente. La incorporación del etidio en el ADN de los organismos vivos (es decir, usted y yo) puede causar mutaciones (no deseadas).
Nota:
- La intercalación del bromuro de etidio provoca una reducción en el número de pares de bases efectivos por torsión.
- Así, el ADN quiere adoptar una conformación con menos pares de bases por giro.
- El número de giros aumentará para la longitud dada del ADN
- Writhe disminuirá si el número de Vinculación permanece sin cambios
- El resultado neto es que la adición de bromuro de etidio introducirá superbobinas negativas en el ADN
- La adición de bromuro de etidio a una muestra de ADN antes de la electroforesis puede hacer que el ADN superenrollado positivamente migre como ADN relajado (mellado o forma II)
- Después de la replicación, el ADN está típicamente subenrollado (superbobinas negativas), por lo que se volverá más subenrollado con el tratamiento con EtBr (la tasa de migración puede aumentar debido a un estado más compacto)
Sombreado de flourescencia:
- Los fragmentos de ADN resueltos en geles de poliacrilamida también se pueden visualizar mediante el método de sombreado UV.
- En este método el gel se coloca encima de un material fluorescente, generalmente una placa de sílice TLC fluorescente.
- Luego, el gel es iluminado por una fuente de luz UV.
- Las bandas de ADN en el gel bloquearán la transmitancia de la luz UV al sustrato.
- Esto dará como resultado un área oscura (es decir, no fluorescente) en el sustrato.
- Este método se usa generalmente en la purificación en gel de oligonucleótidos (es decir, las bandas oscuras se pueden cortar con una cuchilla de afeitar y extraer el oligonucleótido)
Manchas para Proteínas
Coomassie azul brillante
- El azul de Coomassie es un tinte textil de trifenilmetano que es capaz de manchar proteínas.
- Después de que se ejecuta un gel de poliacrilamida, generalmente se “fija” colocándolo en una solución de metanol al 50% /ácido acético 10 durante 30 minutos (para precipitar las proteínas y evitar la difusión fuera del gel).
- El gel fijado se remoja luego en una solución de metanol/ácido acético que contiene 2.5 g/litro de azul Coosmassie.
- El desteñido del gel de fondo se logra remojando los cambios de metanol al 10% /ácido acético al 7%. Este método puede detectar típicamente muestras de proteínas de 0.1 ug o mayores.
Mancha de plata
- La tinción con plata es un método que utiliza una solución de nitrato de plata para teñir proteínas en un gel de acrilamida.
- El método es de naturaleza similar al uso de la plata en placas fotográficas.
- La sensibilidad es aproximadamente dos órdenes de magnitud más sensible que la tinción de Coomassie (es decir, se puede detectar aproximadamente 1 ng de proteína).
- Debido a su alta sensibilidad este método suele emplearse para determinar la presencia de trazas de contaminantes en muestras de proteínas.
Estándares de peso molecular
- Tanto la electroforesis de ADN como de gel de proteínas utilizan patrones de peso molecular para calibrar el tamaño o tamaños de las muestras que se están analizando
- Los patrones de peso molecular de ADN consistirán en una mezcla de fragmentos de ADN de tamaños conocidos (masa molecular)
- Un patrón de peso molecular de ADN conveniente se construye por ligación parcial de un fragmento de 100 pares de bases de ADN dúplex.
- La ligadura parcial dará como resultado la formación de dímeros (200 pb), trímeros (300 pb) y así sucesivamente, así como cierta cantidad del fragmento original (100 pb)
- Esto produce una “escalera” de ADN después de la electroforesis en gel
- Otro fragmento estándar de ADN podría ser una secuencia de ADN conocida, tal como el plásmido pBR322, que se ha digerido con una endonucleasa de restricción de cuatro cortadores (por ejemplo, Alu I).
- Esto produce una variedad de fragmentos con diferentes tamaños
- Se reproduce fácilmente (solo corta más PBR322)
- Un patrón de peso molecular de ADN conveniente se construye por ligación parcial de un fragmento de 100 pares de bases de ADN dúplex.
- Los marcadores de peso molecular de proteínas generalmente consisten en una mezcla de media docena de proteínas puras con masas moleculares conocidas
|
Proteína |
Masa molecular (Da) |
|---|---|
|
Fosforilasa B |
94,000 |
|
Albúmina de suero bovino |
67,000 |
|
Ovoalbúmina |
43,000 |
|
Anhidrasa carbónica |
30,000 |
|
Inhibidor de tripsina de soja |
20,100 |
|
A-lactoalbúmina |
14,400 |
- Tanto en los geles de ADN como de proteínas, los marcadores de peso molecular se ejecutan en un carril en un extremo del gel
.png)
Figura 3.1.6: estándares de tamaño de ADN


