10.2: Transformación Bacteriana (Actividad)
- Page ID
- 56267
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Transformación Bacteriana
Escherichia coli son bacterias gramnegativas comensales que se encuentran en las tripas de los humanos. Tienen la capacidad de duplicarse cada veinte minutos y hacer un portador favorable de ADN recombinante. El ADN plasmídico se puede introducir en E. coli fácilmente después de hacerlos competentes. Un método para lograrlo es a través de la competencia química con el choque térmico. En este proceso, las bacterias se incuban en solución de CaCl 2 sobre hielo. El frío sirve para ralentizar el movimiento molecular de la membrana plasmática mientras que los iones Ca 2+ eliminan la repulsión carga-carga entre los fosfolípidos y el ADN cargado negativamente buscando ingresar a la célula. Las células se colocan por un corto periodo de tiempo a 42°C para inducir choque térmico. Este choque térmico da como resultado que la célula tome el ADN. Este método es de muy baja eficiencia por lo que muchas bacterias no toman ningún ADN. Se permite que las células se recuperen del choque térmico a 37°C en caldo nutritivo rico para permitir la producción de las proteínas de resistencia a antibióticos codificadas en el vector como marcador de selección. Luego, las células transformadas se extienden a través de una placa de agar que contiene el antibiótico que luego destruirá todas las células no transformadas. Solo las bacterias que contienen el vector con el gen de resistencia a antibióticos sobrevivirán y se replicarán para formar pequeñas colonias en la superficie del agar.
Ejercicio: Transformación de bacterias con plásmidos identificados con RE
- Cada grupo recupera los 2 plásmidos mini-preparados de la semana anterior en el congelador y se dejan descongelar en hielo.
- Llevar 2 placas de agar a temperatura ambiente
- 2 placas contendrán antibiótico, X-Gal y arabinosa
- Para cada plásmido, obtener 250μl de tampón de transformación (50 mM CaCl 2) en tubos de microcentrífuga y colocar en hielo durante 10 minutos.
- Tome un asa de inoculación y retire una sola colonia de bacterias de una placa recién rayada cultivada durante la noche.
- Remolino las bacterias en cada tubo que contiene la solución transformadora para distribuir las bacterias por toda la solución.
- Pipetear 5 μl del plásmido en el tubo e incubar en hielo durante 10 minutos.
- Durante esta incubación, voltea las placas calentadas y etiquétalas con los nombres de tu grupo.
- Colocar los tubos de transformación en bloque térmico de 42°C durante 1 minuto para impactar térmicamente las celdas.
- Agregar 500μl de medio SOC fresco (o LB) e incubar a 37°C por 15 minutos.
- Pipetear 150 μl de solución de transformación en cada placa y esparcir por la placa.
- Gire las placas de agar hacia arriba y colóquelas en una incubadora a 37 °C (su instructor los recuperará y los colocará en el refrigerador).
Hipótesis: ¿Qué esperaré de mis células transformadas?
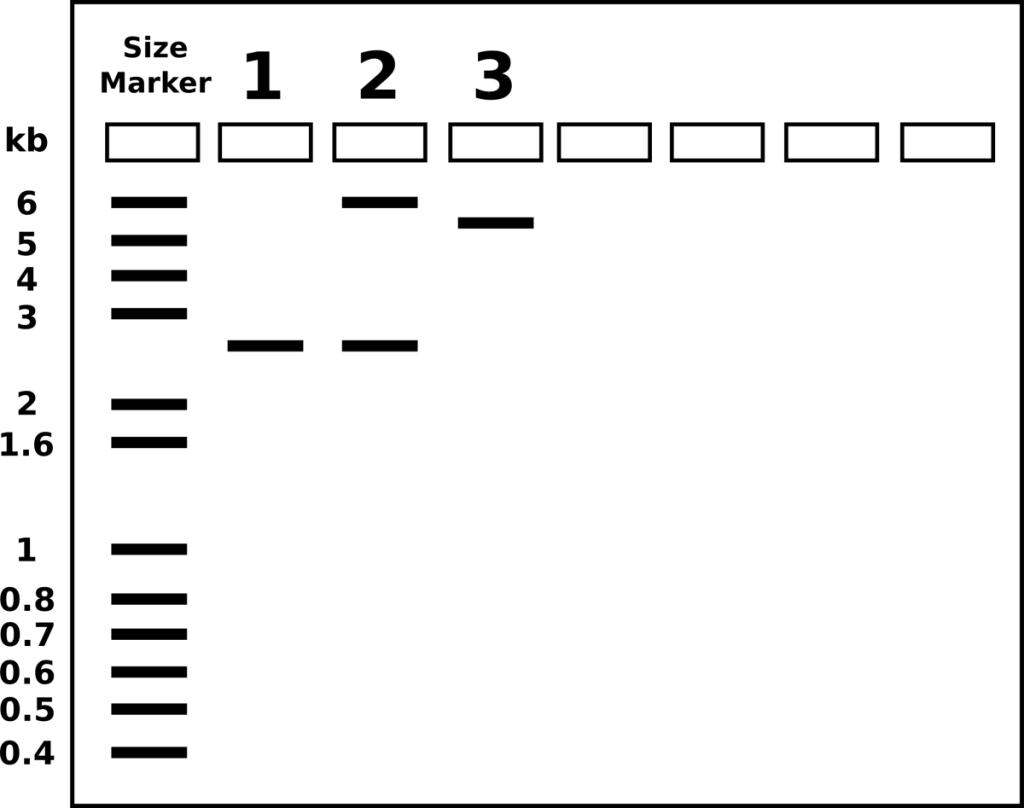
Del laboratorio anterior, podemos identificar nuestros plásmidos. Los plásmidos son pGlo, pUC18/19 o pUC18/19 con un inserto de 6kb alterando el gen lacZ. pglo contiene un gen que codifica la proteína GFP que fluorescerá verde bajo luz UV y es de 5.4kb. pUC es típicamente 2.7kb de tamaño. LacZ es un gen que codifica la proteína β-galactosidasa, la enzima que hidroliza la lactosa en los monosacáridos galactosa y glucosa. X-Gal es un químico parecido a la lactosa. Sin embargo, tras la hidrólisis, la molécula deposita una coloración azul en la célula.


- Si los plásmidos mencionados anteriormente fueron digeridos por EcoRI, etiquetar los carriles de abajo con el plásmido apropiado (pGlo, pUC y pUC insertado)
- Predice si tus transformantes serán verdes bajo UV, blancos en todas las condiciones o azules.
- Para obtener ayuda adicional sobre este problema, utilice la actividad de digestión in silico.