1.9: Regulación de la Expresión Génica
- Page ID
- 56618
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)- Comprender el concepto de expresión génica.
- Comprender la regulación transcripcional de la expresión génica.
- Entender la regulación genética epigenética.4
- Entender la regulación postranscripcional de la expresión génica (nivel de ARN).
- Entender la modificación y regulación de proteínas postraduccionales.
Introducción
Cada célula de una planta contiene la misma información genética, el mismo conjunto de genes. Sin embargo, por lo tanto, se requieren diferentes conjuntos de genes para las diversas funciones de diferentes células o tejidos, así como para las respuestas de las plantas a estímulos o tensiones ambientales. Esto se logra regulando la actividad de los genes de acuerdo con las demandas fisiológicas de un tipo celular particular, etapa de desarrollo o condición ambiental. Esta regulación de la actividad se conoce como expresión génica.
El término expresión se puede utilizar de diferentes maneras que a veces son confusas. Por lo general, si se produce un producto génico, el gen se considera “expresado”. Sin embargo, a veces ocurre que se puede producir un transcrito pero no una proteína, o que se produce una proteína pero se encuentra en un estado inactivo. En tales casos, aunque se produce un producto génico, la actividad biológica codificada por ese gen no está presente. Para los efectos de esta sección, el punto clave es cómo se regula la actividad biológica codificada por un gen.
La expresión de genes en células vegetales, tejidos y órganos específicos y el momento de esta expresión requieren un nivel preciso de regulación. La expresión, o función genética, puede ser potencialmente regulada en cualquiera de los pasos de transcripción, procesamiento de ARN, traducción, a través de la modificación post-traduccional de proteínas, como se discute en la lección 1. La regulación puede ser cualitativa (es decir, la expresión génica está “activada” o “desactivada”) o cuantitativa (es decir, los niveles de expresión pueden modularse “hacia arriba” o “hacia abajo”). Las fluctuaciones en las intensidades de estímulos externos acopladas a cambios que ocurren a nivel genómico dan como resultado diferentes resultados de desarrollo o estados fisiológicos. La regulación de la expresión génica a nivel de transcripción se puede lograr a través de modificaciones de cromatina e histona. Además, se puede empalmar diferencialmente una secuencia génica para producir productos de ARNm de longitudes variables que conducen a nuevos productos proteicos con funciones novedosas. Algunos genes no codifican proteínas sino formas cortas de ARN con funciones reguladoras como la inducción de la floración. Finalmente, los productos proteicos pueden someterse a modificaciones como fosforilación o desfosforilación para alterar sus funciones, o pueden degradarse completamente para desactivar un gen.
Regulación génica transcripcional
Dado que la transcripción es el primer paso en la expresión génica, tiene sentido en términos de economía celular, regular la expresión en este punto, y de hecho este es uno de los puntos reguladores más importantes. Ya describimos la implicación de la ARN polimerasa en el proceso de transcripción, pero de hecho existen otros factores proteicos que se requieren. Las proteínas involucradas en la regulación transcripcional se conocen como factores de transcripción. Es la interacción de estos factores de transcripción con secuencias específicas de ADN lo que regula el proceso de transcripción génica.
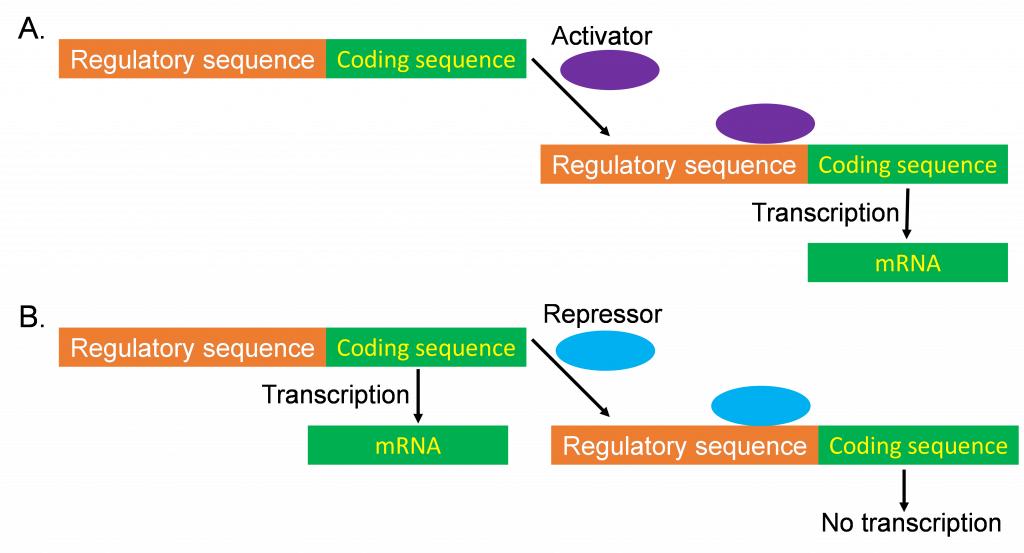
A. El concepto de expresión génica diferencial (regulada)
Como se acaba de describir, no todos los genes se expresan todo el tiempo. Cuando un gen muestra diferentes niveles de expresión en diferentes circunstancias, esto se conoce como expresión diferencial. Las circunstancias que podrían aplicarse incluyen, pero no se limitan a, diferentes tejidos vegetales (raíz vs hoja), diferentes etapas de desarrollo (germinación vs. desarrollo reproductivo) o en respuesta a diferentes estímulos ambientales (estrés por frío o ataque de patógenos).
El término expresión diferencial también se puede utilizar para comparar la expresión de diferentes genes. Si dos genes muestran diferentes patrones de expresión (entre tejidos vegetales o en respuesta a estímulos ambientales), se consideran expresados diferencialmente, mientras que los genes que mostraron patrones de expresión muy similares se considerarían coexpresados.
B. Promotores
Como se mencionó, la transcripción se regula a través de las interacciones de proteínas, factores de transcripción, con secuencias específicas de ADN. La mayoría de las secuencias de ADN reguladoras que gobiernan la transcripción génica se encuentran en el borde 5' de la región transcrita. A esta región se le llama el promotor génico.
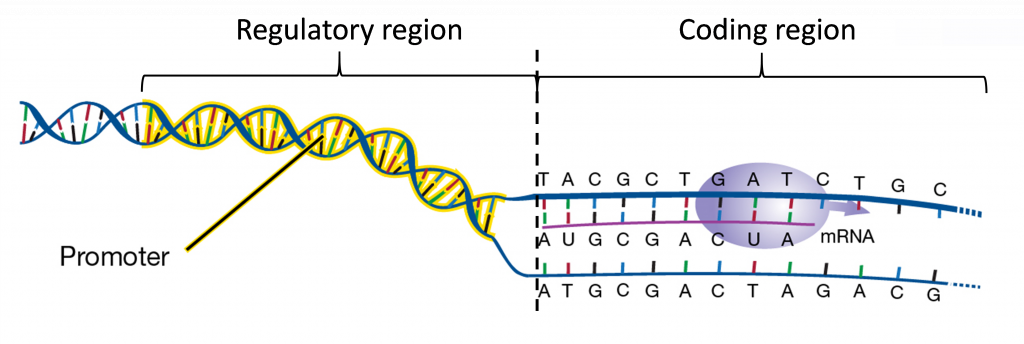
Los promotores contienen un núcleo, el cual es requerido para la unión de la “maquinaria transcripcional basal”, incluyendo la ARN polimerasa. La caja “TATA”, con una secuencia consenso TATAA, se encuentra dentro del promotor central, generalmente 25-30 nucleótidos aguas arriba del sitio de inicio de la transcripción. Los promotores también contienen secuencias reguladoras que determinan cuándo, dónde y a qué nivel se transcriben los genes. Los promotores pueden variar en longitud de cien a unos pocos miles de nucleótidos.
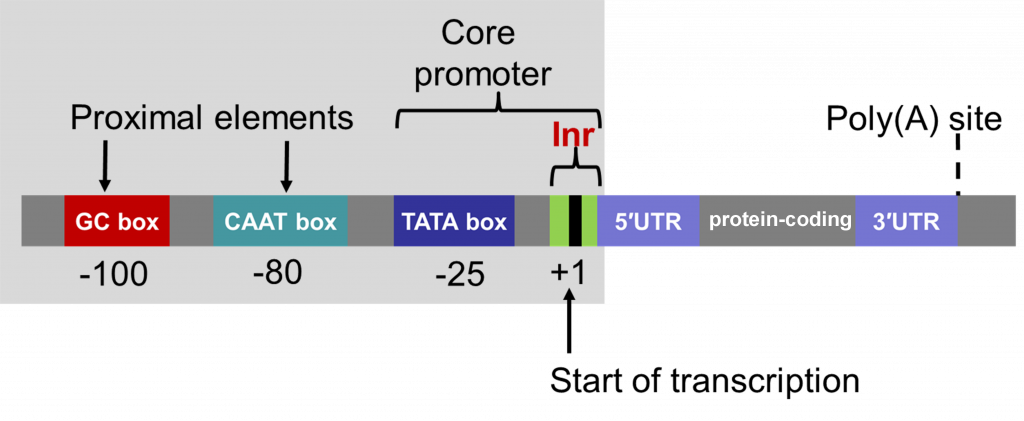
Las secuencias promotoras completas de diferentes genes que se expresan de manera similar pueden ser diferentes. Sin embargo, tales promotores a menudo contienen “motivos” de secuencia corta que son similares, denominados elementos cis. Los primeros trabajos (Benfy y Chua, 1990) para comprender la función de diferentes elementos promotores en la regulación de la expresión génica en células y tejidos vegetales revelaron que diversas combinaciones de elementos cis son capaces de ser interpretadas por la célula y controlar la expresión génica. A veces los elementos cis promueven la transcripción génica y a veces funcionan para restringir la transcripción de genes en células y tejidos particulares.
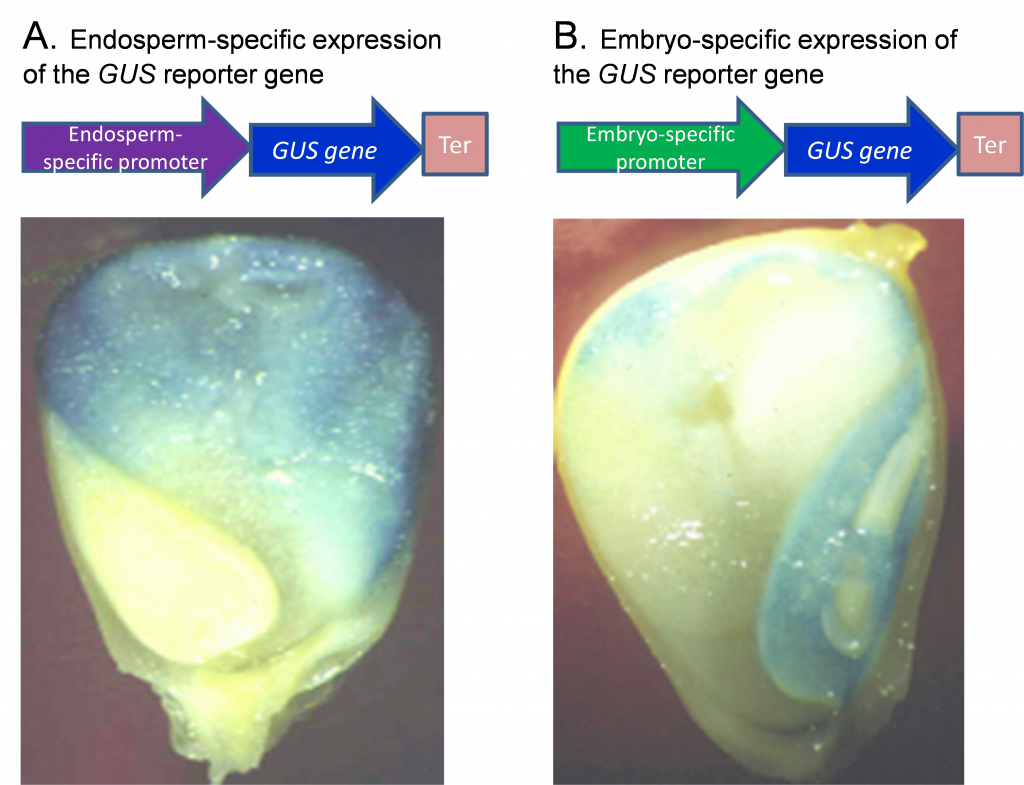
El análisis del promotor se facilita mediante el uso de genes informadores. El gen indicador produce efectos que son fácilmente identificables y cuantificables, los cuales pueden ser utilizados para determinar la función de una región reguladora de otro gen (promotor, elementos promotores o potenciadores) en células, tejidos u órganos. Dichos análisis son críticos para la biotecnología de cultivos donde a menudo es deseable la expresión dirigida de genes en tejidos particulares. Para probar si un promotor es efectivo para conferir la expresión de un gen a un tejido en particular, los científicos fusionan el promotor putativo con un gen informador e introducen la fusión promotor-gen en las plantas. Un ejemplo de un reportero es el gen GUS, que codifica una enzima GUS (beta-glucuronidasa) que, cuando se expresa, produce un color azul tras la adición de un sustrato. La Figura 1 muestra un ejemplo de expresión de GUS en semillas de maíz utilizando dos promotores que actúan ya sea en el endospermo o en el embrión.
C. Potenciadores
Los potenciadores son secuencias de ADN que aumentan la tasa de transcripción de un gen cuando están presentes, aunque por sí solas no pueden causar que se produzca la transcripción. Los potenciadores suelen ser independientes de la posición y la orientación. Aunque normalmente se localizan aguas arriba del promotor, también pueden localizarse en la región 3' del gen o incluso con la región codificante. Los potenciadores pueden aumentar la transcripción cuando se agregan a genes con los que normalmente no están asociados. Esta es una propiedad útil para la biotecnología, permitiendo que los promotores sean manipulados para aumentar los niveles de regulación transcripcional. Algunos potenciadores funcionan en todo momento en todas las células y tejidos y se les conoce como constitutivos. Otros potenciadores funcionan en tejidos específicos en etapas específicas de desarrollo. Algunos están activos solo en respuesta a las señales ambientales. El potenciador AACCA en el promotor del gen β-conglicinina de soja que codifica una proteína de almacenamiento de semillas funciona específicamente en semillas. Se cree que los potenciadores interactúan con proteínas nucleares específicas involucradas en la transcripción. Por ejemplo, los potenciadores podrían facilitar la unión de factores de transcripción y dirigir estos factores a lo largo de la cadena de ADN hacia la dirección del promotor. Alternativamente, los potenciadores pueden facilitar cambios en la estructura del ADN tales como la modificación de la estructura de la cromatina.
La contraparte de un potenciador es un silenciador. Los silenciadores tienen todas las propiedades que se acaban de describir para los potenciadores, excepto que funcionan para amortiguar, o disminuir, los niveles de transcripción controlados por un promotor.
D. Factores de transcripción
La ARN polimerasa se une al promotor en la caja TATA y en cooperación con otras proteínas impulsa la transcripción génica. Las proteínas que interactúan con la ARN polimerasa para facilitar su unión al promotor y regular su actividad se conocen como factores de transcripción. Algunos factores de transcripción, conocidos como “factores de transcripción basales” son fundamentales para la unión y función de la ARN polimerasa, y se expresan en todas las células vivas.
Otros factores de transcripción se unen a secuencias de ADN reguladoras cis de promotores, potenciadores o silenciadores. Estas proteínas interactúan de manera compleja con la maquinaria de transcripción basal, para regular la actividad de la ARN polimerasa, y por lo tanto la transcripción génica. Así, los factores de transcripción regulan cuándo o dónde se expresan los genes individuales, y a qué nivel.
Los factores de transcripción pueden funcionar como reguladores positivos o negativos. Es decir, pueden funcionar para inducir (aumentar) la transcripción génica o para reprimirla. La consecuencia de muchos factores de transcripción depende de sus interacciones con otras proteínas. Dos factores juntos pueden ser necesarios para la actividad génica, y la exclusión de uno de los factores en el espacio y el tiempo ofrece un mecanismo para la expresión génica diferencial. Algunos factores de transcripción pueden funcionar como un regulador positivo en un contexto pero como un regulador negativo en otro contexto, dependiendo de qué otros elementos cis y/o factores de transcripción puedan estar presentes.
Regulación epigenética
A. Estructura de la cromatina y modificación de histonas
A nivel molecular, la cromatina es producto de un empaque ordenado y apretado del ADN bicatenario alrededor de los nucleosomas (un núcleo de proteínas llamadas histonas), y una asociación con proteínas adicionales.
La regulación transcripcional a menudo implica la modificación de la estructura de la cromatina mediada por la regulación postraduccional (cambios en la acetilación y metilación) de las histonas. La acetilación de histonas implica la adición de grupos acetilo y cuando las histonas están fuertemente acetiladas, el ADN se asocia menos estrechamente con ellas. Esto a menudo se correlaciona con el aumento de la actividad transcripcional de genes específicos. La idea es que cuando el ADN se asocia libremente con las histonas, es más accesible a factores de transcripción que requieren interacción con el ADN para iniciar la transcripción. En consecuencia, la desacetilación de histonas (eliminación de grupos acetilo) por enzimas histonas desacetilasas (por ejemplo, FLD, p462) estabiliza los nucleosomas y reprime la transcripción. Por otro lado, la acetilación de histonas por la histona ecetiltransferasa desestabiliza los nucleosomas y promueve la transcripción.
Como se describe en el texto, otra forma de modificación de histonas es la adición de grupos metilo a las proteínas histonas, que de manera similar regula la condensación o descondensación de la cromatina.
B. Metilación del ADN
Las bases nucleotídicas del ADN se pueden modificar mediante la unión de grupos metilo en diversas localizaciones. La adición de estos grupos metilo ocurre después de que el ADN ha sido sintetizado y es controlado por enzimas que agregan restos metilo a regiones específicas del ADN. La base nucleotídica modificada más común es la metilcitosina C5 (m5c) debido a la actividad de la enzima ADN (citosina-5) metiltransferasa (MET). MET reconoce la cadena de ADN no metilada recién replicada e incorpora un grupo metilo si la cadena molde estaba metilada (hemimetilada).
El estado de metilación se correlaciona con la expresión génica (Figura 5) como lo demuestra el bajo nivel de metilación en regiones del genoma sometidas a transcripción activa.
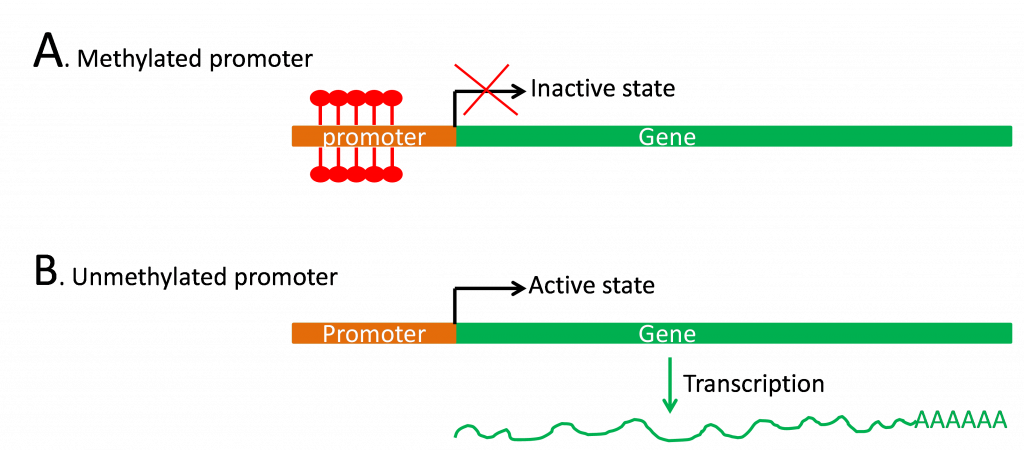
Los estudios han demostrado que alterar el estado general de metilación del ADN de una planta puede afectar el crecimiento y el desarrollo. Por ejemplo, el tratamiento con frío (vernalización) induce la floración en bianuales como Arabidopsis y trigo de invierno, y disminuye el nivel de metilación de genes particulares. Además, el tratamiento de las plantas con el fármaco 5 azacitidina evita la metilación en la posición 5 de la citosina y estimula la floración. Estudios recientes también sugieren una relación entre la regulación genética epigenética y la heterosis, con híbridos que muestran mayores niveles globales de transcripción, mayores niveles de acetilación de histonas y menores niveles de metilación del ADN (revisado en He et al., 2011).
Regulación a nivel de ARN
La regulación de la expresión génica ocurre en muchos niveles, incluso postranscripcionalmente. Desde el punto de vista del nivel de expresión para un gen dado, un factor crítico es el nivel de ARNm maduro completamente procesado. Cuando consideramos el nivel de la forma madura, una transcripción particular en un momento dado refleja el equilibrio en estado estacionario entre síntesis, procesamiento y degradación. La regulación postranscripcional del ARN se produce a través de varios mecanismos.
Estabilidad y degradación del ARN
El control de la tasa de degradación del ARNm, o recambio, es un mecanismo regulador importante en la expresión génica. Como se mencionó, el nivel de una transcripción particular en un momento dado refleja el equilibrio en estado estacionario entre síntesis y degradación. Por lo tanto, aunque un gen pueda transcribirse a una tasa alta, una alta tasa de recambio de ARN podría efectivamente cerrar el gen. La estabilidad del ARNm puede regularse globalmente, para afectar a todos o a la mayoría de los transcritos, o puede ser muy específica para un ARNm particular. El recambio regulado del ARNm puede ser particularmente importante en las plantas, ya que las plantas no pueden moverse para evitar las duras condiciones ambientales y se sabe que los estreses inducen cambios específicos en la estabilidad del ARN. También se sabe que otros factores como la luz y las hormonas vegetales regulan la estabilidad del ARNm.
La degradación del ARNm es catalizada por enzimas llamadas ribonucleasas. Las ribonucleasas incluyen exonucleasas, que se degradan solo desde un extremo del transcrito, y endonucleasas, que pueden atacar la molécula de ARNm internamente.
Por lo tanto, controlar el acceso de ribonucleasa al sustrato de ARNm es un mecanismo regulador importante en la expresión génica. Este control se logra a través de diversos mecanismos que incluyen, controlar la cantidad de nucleasas presentes, regular la actividad de la nucleasa, secuestrar la nucleasa a ubicaciones celulares particulares para restringir su acceso al ARN, y controlar la estabilidad del ARNm disminuyendo la accesibilidad a nucleasa.
Cuatro regiones de un ARNm son importantes para su estabilidad general. Estos incluyen, la región 5' no traducida y la caperuza, región codificante, región 3' no traducida y la cola poli (A).
La caperuza en el extremo 5' protege al ARNm de las exonucleasas que degradan el ARN desde la dirección 5' a la 3'.
La región 3' no traducida puede contener secuencias cortas que influyen en la estabilidad del ARNm. Por ejemplo, la repetición de la secuencia AUUUA en la región 3′ no traducida de muchos genes animales y vegetales se asocia con ARNm con semividas cortas (el tiempo que tarda la mitad del ARN en degradarse, después de que se detiene la transcripción).
La cola poli (A) aumenta la estabilidad del ARNm pero no es suficiente sola. Solo proporciona estabilidad cuando se une a proteínas como la proteína de unión a poli (A) (PABP). Los experimentos con probeta han demostrado que al eliminar PABP de un ARNm estable la desestabiliza, mientras que la adición de PABP nuevamente purificada restaura la estabilidad.
Regulación traslacional
Como se describe, para la mayoría de los genes es el producto proteico el que realiza la función biológica. Como tal, regular la cantidad de producción de proteínas regula eficazmente la expresión génica. Existen varios mecanismos conocidos por los cuales se sabe que se regula el proceso de traducción. Una consideración detallada de estos mecanismos está más allá del alcance de este curso, pero en general es importante tener presente su importancia. Por ejemplo, bajo ciertas condiciones de estrés, se detiene la traducción “normal” y solo se permite que los ARNm relacionados con la tolerancia al estrés se traduzcan selectivamente en proteínas. Esta traducción selectiva es importante para permitir que las plantas respondan rápidamente al estrés y conserven energía en condiciones de estrés.
La traducción de los ARNm puede desempeñar un papel importante en la determinación de su estabilidad general. Las mutaciones que agregan codones de parada prematuros a menudo conducen a una rápida degradación del ARNm. Las ribonucleasas (figura 9) u otros factores involucrados en la degradación pueden reconocer el número o espaciamiento de ribosomas en un ARNm, y degradar aquellos que no se producen adecuadamente. Esto puede ayudar a prevenir la síntesis de proteínas con funciones incorrectas que impactarán negativamente en los procesos celulares. Los errores durante la transcripción podrían agregar u omitir nucleótidos que alterarían la secuencia de codones apropiada creando proteínas mutantes.
Regulación a nivel de proteínas
Después de la traducción, las proteínas están sujetas a una variedad de modificaciones que pueden regular su actividad. Hay muchas maneras diferentes por las cuales las proteínas pueden ser potencialmente modificadas y con ello reguladas, y solo algunos de los mecanismos básicos serán considerados aquí.
Tipos comunes de modificaciones postraduccionales
El primer mecanismo es la modificación covalente mediante la adición de diversos grupos químicos. Se puede involucrar una amplia variedad de grupos, incluyendo pequeños grupos orgánicos (metilación, acetilación) lípidos (miristoilación, farnesilación, palmitilación), carbohidratos (glicosilación, glucosilación), proteínas pequeñas (ubiquitinación, sumoilación) y moléculas inorgánicas (fosforilación, sulfatación). Dichas modificaciones covalentes se logran generalmente por la actividad de enzimas especializadas para realizar estas modificaciones. Muchas de estas modificaciones también son reversibles; es decir, estos grupos pueden agregarse a una proteína y posteriormente eliminarse. La fosforilación es una modificación reversible particularmente notable que es común en la regulación de muchas proteínas. Las enzimas que agregan grupos fosfato a otras proteínas se llaman proteína quinasas, y las que eliminan los fosfatos se llaman fosfatasas. Como tal, las proteínas quinasas y fosfatasas son fundamentales para muchos sistemas reguladores celulares.
Un segundo mecanismo común de modificación de proteínas es a través de la escisión proteolítica. La proteólisis ocurre como parte del recambio general de las proteínas celulares, que se requiere para eliminar las proteínas dañadas y reciclar los aminoácidos. La proteólisis es importante para el procesamiento de ciertas proteínas, por ejemplo en la eliminación del péptido señal de proteínas dirigidas a compartimentos celulares específicos. También puede ocurrir de una manera altamente específica mediante la cual proteínas particulares se dirigen para la degradación o para la escisión en un sitio específico dentro de la proteína. Las modificaciones proteolíticas no son reversibles.
Las proteínas pueden sufrir modificaciones a través de la formación de complejos. Tales complejos pueden ocurrir entre proteínas o entre una proteína y un cofactor.
Las proteínas también se pueden modificar de acuerdo a las condiciones del entorno celular. El estado redox puede dar como resultado la oxidación o reducción de proteínas, particularmente de grupos laterales sulfhidrilo. El pH celular puede afectar la carga de grupos laterales ionizables.
Tipos comunes de regulación de proteínas
Todos los tipos de modificación de proteínas mencionados anteriormente pueden alterar las conformaciones proteicas y, por lo tanto, tener consecuencias reguladoras sobre la función o actividad de la proteína. La regulación de proteínas es altamente compleja y hay una miríada de formas diferentes por las cuales esto ocurre. Nuevamente vamos a considerar brevemente algunos de los mecanismos más comunes.
Una forma principal en que la modificación de proteínas puede regular la función de la proteína es alterando su actividad. Esto puede ser cierto para muchos tipos de proteínas incluyendo, pero no limitado a, enzimas, factores de transcripción, proteínas de señalización y proteínas estructurales.
Las células se compartimentan en varios orgánulos unidos a membrana, incluyendo el núcleo, cloroplastos, mitocondrias, peroxisomas, retículo endoplásmico (ER), golgi y vacuolas. Cada uno de estos compartimentos realiza funciones metabólicas únicas que requieren un conjunto de proteínas. La compartimentación está regulada para algunas proteínas. Por ejemplo, tras la exposición a la luz, la proteína fitocroma se mueve hacia el núcleo donde afecta la expresión de genes regulados por la luz.
El procesamiento proteolítico está involucrado en varios procesos regulatorios importantes. Muchas proteínas se sintetizan en una forma inactiva que requiere escisión proteolítica para su activación. El producto de traducción de longitud completa antes del procesamiento a menudo se llama precursor, o preproteína. Como se mencionó, las células contienen compartimentos unidos a membrana. Dado que las membranas son impermeables a la mayoría de las proteínas, es necesario un mecanismo activo para mover una proteína a través de una membrana. Para una correcta entrega a sus destinos organelares, secuencias específicas de aminoácidos, llamadas señales diana, deben estar presentes en una proteína (para que sirvan como “dirección”). Por ejemplo, la entrada en el cloroplasto se logra mediante la presencia de una señal diana llamada péptido de tránsito. Esto se encuentra en el extremo amino terminal de la proteína y se escinde proteolíticamente durante la importación.
Otro ejemplo de procesamiento proteolítico se observa en una respuesta de defensa vegetal en miembros de las solanáceas (por ejemplo, tomate y papa). Una hormona peptídica de 18 aminoácidos llamada sistemina es secretada por células vegetales que son dañadas por insectos o heridas mecánicas. La producción de sistemina por células heridas es necesaria para inducir la síntesis de proteínas involucradas en la defensa. Systemin induce respuestas de defensa en células heridas y en toda la planta. Análogamente a las hormonas peptídicas animales (p. ej., insulina), la sistemina se sintetiza inicialmente como un precursor mucho más grande (200 aminoácidos) llamado pro-sistemina. Pro-sistema es inactivo; sin embargo, al herirse experimenta escisión proteolítica para producir sistemina activada.
Degradación proteica dirigida
La cantidad de proteína presente en una célula o tejido está determinada tanto por su velocidad de síntesis como por su tasa de degradación. Por lo tanto, la degradación de proteínas es un mecanismo importante mediante el cual la planta puede regular la actividad biológica (es decir, una función genética). Por ejemplo, una forma de cerrar una ruta metabólica es degradando una de las enzimas clave que controlan la velocidad de toda la ruta. Por lo tanto, la degradación de proteínas es un componente esencial de la regulación génica para satisfacer las demandas celulares de crecimiento, desarrollo y defensa.
La degradación de las proteínas debe controlarse cuidadosamente para ajustar la expresión génica y permitir que las plantas se adapten a las nuevas condiciones ambientales. A menudo, las células adoptarán varios mecanismos complejos para la degradación proteolítica de las proteínas. Las enzimas que escinden o degradan proteínas se denominan proteasas. Por ejemplo, las vacuolas vegetales son ricas en proteasas que desempeñan una función similar en la degradación de proteínas a la de los lisosomas en las células animales. La actividad de la proteasa debe estar estrechamente regulada para evitar la degradación accidental de las proteínas esenciales. Secuestrar proteasas en orgánulos particulares como la vacuola las separa de otros orgánulos y es una de las que controlan su actividad.
Un mecanismo importante por el cual se dirigen proteínas específicas para la degradación es a través de la degradación proteasómica mediada por ubiquitina. El proteasoma es un gran complejo de múltiples subunidades proteicas que tienen una actividad proteasa para degradar proteínas. Las proteínas se marcan para la degradación proteasomal con una pequeña proteína llamada ubiquitina. La ubiquitina se une covalentemente a proteínas específicas en respuesta a señales ambientales o de desarrollo. Esto permite que las plantas se adapten rápidamente a las condiciones cambiantes al eliminar proteínas cuyas funciones no son ventajosas bajo las nuevas condiciones. Por ejemplo, el fotorreceptor fitocromo mencionado anteriormente se convierte en blanco de degradación cuando la luz ya no está disponible. Esto permite que las plantas cambien sus funciones fisiológicas pasando de condiciones diurnas a nocturnas. La degradación de proteínas por el sistema proteasoma también es un importante mecanismo regulador para la señalización de hormonas vegetales, por ejemplo, la señalización de la hormona de defensa ácido jasmónico y ácido giberélico de la hormona del crecimiento.
Las proteínas tienen tiempos de vida que van desde unos pocos minutos hasta semanas o más. Las células producen continuamente proteínas y las descomponen en aminoácidos. Una de las funciones de la degradación de proteínas es eliminar las proteínas aberrantes o dañadas que podrían dañar la célula. La segunda función es facilitar el reciclaje de aminoácidos. Por ejemplo, la mayoría de los aminoácidos requeridos para el crecimiento de la plántula se derivan de la degradación de las proteínas de almacenamiento de semillas. Por el contrario, en las plantas de cultivo anuales, muchos de los aminoácidos en las proteínas de almacenamiento de semillas se derivan de proteínas degradadas en las hojas y otras partes de la planta durante la senescencia.
Resumen de la lección
La expresión de genes en células vegetales, tejidos y órganos específicos y el momento de esta expresión requieren un nivel preciso de regulación. Un solo promotor puede no ser suficiente para regular la expresión de dicho gen o genes en el espacio y el tiempo. Por lo tanto, las regiones codificantes con la misma función pueden tener diferentes promotores, y dichos genes se denominan regulados diferencialmente. La mayoría de las secuencias reguladoras que gobiernan la expresión génica se encuentran en el borde 5' de la región codificante. La transcripción a menudo se inicia entre 20 y 60 nucleótidos aguas arriba del sitio de inicio ATG. Los potenciadores suelen ser independientes de la posición y la orientación. Aunque normalmente se localizan aguas arriba del promotor, también pueden localizarse en la región 3' del gen o incluso con la región codificante. Los potenciadores pueden aumentar la transcripción cuando se agregan a genes con los que no están asociados. La ARN polimerasa se une al promotor en la caja TATA y en cooperación con otras proteínas impulsa la transcripción génica. Las proteínas que interactúan con la ARN polimerasa se unen a secuencias reguladoras aguas arriba y aguas abajo del sitio de transcripción.
Los factores de transcripción pueden desempeñar un papel regulador al determinar dónde se expresan los genes individuales. La regulación transcripcional a menudo implica la modificación de la cromatina por cambios en la acetilación y metilación de la histona. Las bases nucleotídicas del ADN se pueden modificar mediante la unión de grupos metilo en diversas localizaciones. El estado de metilación se correlaciona con la expresión génica. El corte y empalme alternativo describe un mecanismo alternativo de procesamiento previo al ARNm para generar ARNm que tienen diferentes combinaciones de exones. El control de la tasa de degradación del ARNm, o recambio, es un mecanismo regulador importante en la expresión génica. La interferencia de ARN (ARNi) es un proceso postranscripcional que implica la degradación del ARNm iniciada por la formación de ARN bicatenario (ARNds) del ARNm diana. La traducción de los ARNm puede desempeñar un papel importante en la determinación de su estabilidad general. Las mutaciones que agregan codones de parada prematuros a menudo conducen a una rápida degradación del ARNm. La degradación de proteínas es un componente esencial de la regulación génica para satisfacer las demandas celulares de crecimiento, desarrollo y defensa. La planta puede alterar la actividad de una vía metabólica, degradando una de las enzimas clave que controlan la velocidad de toda la ruta.
Ejercicios
1a. ¿Cuál de los siguientes podría alterar la regulación génica?
- Suprimiendo un promotor
- Alterar la tasa de aplicación
- Elevar la temperatura del invernadero
- Aplicación de herbicidas
- Reducir la frecuencia de riego
1b. La detección de un producto génico, por ejemplo, ARN o proteína en cualquier momento es un reflejo del “estado estacionario” del producto. Describir el término “estado estacionario” en este contexto.
2. Estás estudiando la expresión de un gen responsable de la resistencia a un patógeno. Clonaste el gen y hiciste anticuerpo para detectar la proteína que codifica mediante un procedimiento llamado análisis de transferencia Western, y el ARNm mediante un procedimiento llamado PCR de transcripción inversa. Explicar el tipo de regulación a partir de los escenarios en la tabla.
|
Escenarios |
No tratado con patógeno
Tratados con patógeno
mRNA
Proteína
mRNA
Proteína
1
Ausente
Ausente
Presente
Presente
2
Presente
Ausente
Presente
Presente
3
Presente
Presente
Ausente
Ausente
4
Presente
Presente
Presente
Ausente
5
Presente
Presente
Presente
Presente
3. Estás estudiando la expresión de un gen que controla la altura en tu especie de cultivo. Usted ha clonado el gen y está interesado en determinar su expresión en diferentes partes de la planta. Se utiliza el procedimiento de RT-PCR como en el problema anterior. Después de la parte de PCR, se cargan los productos en un gel y se observa el siguiente patrón. Explique sus resultados en el contexto de la regulación génica.
Referencias
Benfey, P. N., N. Chua. 1990. El promotor 35S del virus del mosaico de la coliflor: Regulación combinatoria de la transcripción en plantas. Ciencia 16:959-966.
Chung, H. S., G. A. Howe. 2009. Un papel crítico para el motivo TIFY en la represión de la señalización de jasmonato mediante una variante de empalme estabilizada de la proteína JASMONATE ZIM-dominio JAZ10 en Arabidopsis. Célula Vegetal 21:131-145.
Él, G., Eling, A.A., y Deng, X.W. (2011). El Epigenoma y el Desarrollo Vegetal. Revisión Anual de Biología Vegetal 62, 411-435.
ISHIHAMA, N., R. YAMADA, M. YOSHIOKA, S. KATOU y H. YOSHIOKA, 2010 Fosforilación del Factor de Transcripción de Nicotiana benthamiana WRKY8 por MAPK Funciones en la Respuesta de Defensa. Célula Vegetal 23:1153-1170.
Nakaminami, K., Matsui, A., Shinozaki, K., y Seki, M. (2011). Regulación del ARN en las respuestas al estrés abiótico vegetal. Biochimica et Biophysica Acta (BBA) — Mecanismos Reguladores de Genes.
Sheen, J., 1991. Mecanismos moleculares subyacentes a la expresión diferencial de piruvato de maíz, genes de ortofosfato diquinasa. Célula Vegetal 3:225-245.


