1: Propiedades Fundamentales de los Genes
- Page ID
- 58711

Mapeo de genes por transferencia conyugal
La transferencia conyugal también se puede utilizar para el mapeo genético. Mediante el uso de muchas cepas diferentes de hfr, cada una con el factor F integrado en una parte diferente del colicromosoma E., se mapearon las posiciones de muchos genes. Estos estudios mostraron que el mapa genético del cromosoma de E. coli es circular. Durante la transferencia conyugal, los genes más cercanos al sitio de integración de F se transfieren primero. Al interrumpir el apareamiento en diferentes momentos, se puede determinar qué genes están más cerca del sitio de integración. Así, en el cromosoma de E. coli, los genes se mapean en términos de minutos (es decir, el tiempo que lleva transferirse al receptor).
Por ejemplo, para una cepa hfr con el factor F integrado a los 0 min en el mapa de E. coli, la transferencia conyugal a una receptora femenina transferiría
- LeuACBD a 1.7 min
- PyRH a 4.6 min
- ProAb a 5.9 min
- BioABFCD a 17.5 min.

Bacteriófagos
Bacteriófagosson virus que infectan bacterias. Debido a su gran número de progenie y capacidad para recombinarse en infecciones mixtas (más de una cepa de bacterias en una infección), se han utilizado ampliamente en la definición de genes de alta resolución. Gran parte de lo que sabemos sobre la estructura genética fina, previo al advenimiento de las técnicas de aislamiento y secuenciación de genes, derivan de estudios de forma en bacteriófagos.
Los bacteriófagos han sido un poderoso sistema genético modelo, porque tienen genomas pequeños, tienen un ciclo de vida corto y producen mucha progenie a partir de una célula infectada. Proporcionan un medio muy eficiente para la transferencia de ADN dentro o entre células. El gran número de progenie permite medir eventos de recombinación muy raros.
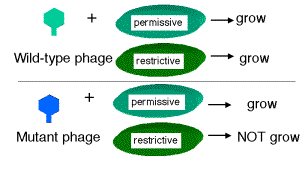
Los bacteriófagos líticos forman placas en el césped de bacterias; estas son regiones de aclaramiento donde las bacterias infectadas se han lisado. Los primeros trabajos se centraron en mutantes con diferente morfología de placa, por ejemplo T2 r, que muestra lisis rápida y genera placas más grandes, o en mutantes con diferente rango de hospedadores, por ejemplo T2 h, que matarán tanto las cepas hospedadoras B como B/2.
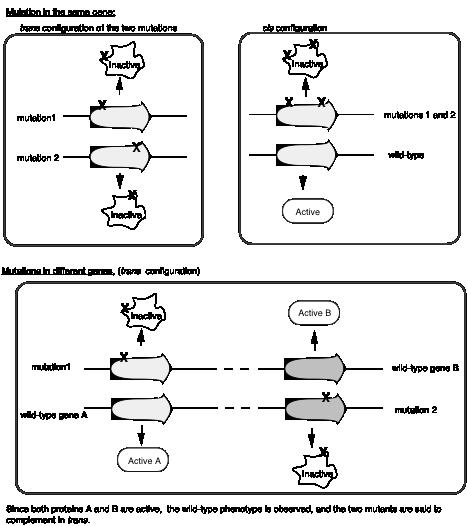
Una prueba de complementación cis-trans define un cistrón, que es un gen
Seymour Benzer utilizó los r IIOCUs del fago T4 para definir genes en virtud de su comportamiento en una prueba de complementación, y también para proporcionar una visión fundamental de la estructura de los genes (en particular, la disposición de los sitios mutables - ver la siguiente sección). La diferencia en la morfología de la placa entre los fagos r y r+ es fácil de ver (grande versus pequeño, respectivamente), y Benzer aisló muchos mutantes r del fago T4. El tipo silvestre, pero no ningún mutante de RII, crecerá en la cepa de E. coli K12 (l), mientras que tanto el fago de tipo silvestre como el mutante crecen igualmente bien en la cepa B. Por lo tanto, el fenotipo silvestre se detecta fácilmente por su capacidad para crecer en la cepa K12 (l).
Si la cepa de E. coli K12 (l) está coinfectada con 2 fagos portadores de mutaciones en diferentes posiciones en RllA, no se obtiene multiplicación del fago (excepto los extremadamente raros recombinantes de tipo silvestre, que ocurren aproximadamente en 1 de cada 106 progenie). En el siguiente diagrama, cada línea representa el cromosoma de uno de los fagos parentales.
RIIa RIIb
fago 1 _|__x______|________|_
fago 2 _|_______x_|________|_
De igual manera, si los dos fagos en la coinfección portan mutaciones en diferentes posiciones en RIIb, no se obtiene multiplicación del fago (excepto los extremadamente raros recombinantes de tipo silvestre, aproximadamente 1 de cada 106).
RIIa RIIb
fago 3 _|_________|_x______|_
fago 4 _|_________|______x_|_
Sin embargo, si uno de los fagos coinfectantes porta una mutación en RIIa y el otro una mutación en RIIb, entonces se ve multiplicación del fago, formando un número muy grande de placas en la cepa K12 (l) de E. coli.
RIIa RIIb
fago 1 _|__x______|________|_ Proporciona proteína wt RIIb
fago 4 _|_________|______x_|_ Proporciona proteína RIIa wt
Juntos, estos dos fagos proporcionan todas las funciones del fago, se complementan entre sí. Esta es una prueba de complementación positiva. Los dos primeros ejemplos no muestran complementación, y los colocamos en el mismo grupo de complementación. Los mutantes que no complementan se colocan en el mismo grupo de complementación; son diferentes alelos mutantes del mismo gen. Benzer demostró que había dos grupos de complementación (y por lo tanto dos genes) en el locus r II, al que llamó A y B.
En la infección mixta con fago 1 y fago 4, también se obtienen los raros recombinantes de tipo silvestre, pero hay más recombinantes que los que se ven en las coinfecciones con diferentes alelos mutantes. ¿Por qué?
Considerar los resultados de infección de un cultivo bacteriano con dos alelos mutantes del gen RIIa.
T4 RIIa6 _|_______________________x______|_
y T4 RIIa27 _|_______x______________________|_
(x marca la posición de la mutación en cada alelo).
Los fagos de progenie de esta infección incluyen aquellos con un genotipo parental (en la gran mayoría), y con una frecuencia mucho menor, dos tipos de recombinantes:
tipo salvaje T4 r+ _|______________________________|_
mutante doble T4 RIIA6 RIIA27 _|_______x_______________x______|_
El tipo salvaje se puntuará fácilmente porque, y no ningún mutante de RII, crecerá en la cepa de E. coli K12 (l), mientras que tanto el fago de tipo silvestre como el mutante crecen igualmente bien en la cepa B. De esta manera se puede seleccionar para el tipo silvestre (y verá solo el recombinante deseado). Encontrar los mutantes dobles es más laborioso, ya que se obtienen únicamente mediante el cribado a través de la progenie, probando fagos que cuando se retrocruzan con el fago parental no dan como resultado progenie recombinante de tipo silvestre.
Se obtuvieron números iguales de recombinantes de tipo silvestre y de doble mutante, demostrando que la recombinación puede ocurrir dentro de un gen, y que esto ocurre por cruce recíproco. Si la recombinación fuera solo entre genes, entonces no resultaría ningún fago de tipo silvestre. Se obtuvo un amplio espectro de valores de recombinación en cruces para diferentes alelos, al igual que se obtiene para cruces entre mutantes en genes separados.
Como resultado de estos experimentos de recombinación dentro de los genes rII podrían hacerse varias conclusiones importantes.
- Un gran número de sitios mutables ocurren dentro de un gen, excediendo unos 500 para los genes RIIa y RIIb. Ahora nos damos cuenta de que estos corresponden a los pares de bases individuales dentro del gen.
- Los mapas genéticos son claramente lineales, lo que indica que el gen es lineal. Ahora sabemos que un gen es un polímero lineal de nucleótidos.
- La mayoría de las mutaciones son cambios en un sitio mutable (mutaciones puntuales). Muchos genes pueden ser restaurados a tipo salvaje sometiéndose a una mutación inversa en el mismo sitio (reversión).
- Otras mutaciones provocan la deleción de uno o más sitios mutables, reflejando una pérdida física de parte del gen rII. Las eliminaciones de uno o más sitios mutables (par de bases) son extremadamente improbables que vuelvan al tipo salvaje original.
Un gen codifica un polipéptido
Una de las ideas fundamentales sobre cómo funcionan los genes es que un gen codifica una enzima (o más precisamente, un polipéptido). Beadle y Tatum llegaron a esta conclusión con base en su análisis de complementación de los genes requeridos para la biosíntesis de arginina en hongos. Mostraron que una mutación en cada gen condujo a una pérdida de actividad de una enzima en la vía multietapa de la biosíntesis de arginina. Como se discutió anteriormente en la sección sobre disección genética, se aislaron un gran número de auxótrofos Arg (que requieren Arg para el crecimiento), y luego se organizaron en un conjunto de grupos de complementación, donde cada grupo de complementación representa un gen.
El trabajo clásico de Beadle y Tatum demostró una relación directa entre los genes definidos por los mutantes auxotróficos y las enzimas requeridas para la biosíntesis de Arg. Mostraron que una mutación en un gen resultó en la pérdida de una actividad enzimática particular, por ejemplo, en el esquema generalizado a continuación, una mutación en el gen 2 condujo a una pérdida de actividad de la enzima 2. Esto condujo a una acumulación del sustrato para esa reacción (N intermedio en el diagrama a continuación). Si hubo 4 grupos de complementación para los auxótrofos Arg, es decir, 4 genes, entonces se encontraron 4 enzimas en la vía para la biosíntesis de Arg. Cada enzima fue afectada por mutaciones en uno de los grupos de complementación.
Intermedios:
M® N® O® P® Arg
enzima 1 enzima 2 enzima 3 enzima 4
gen 1 gen 2 gen 3 gen 4
Figura 1.15. Un esquema general que muestra las relaciones entre los intermedios metabólicos (M, N, O, P), y el producto final (Arg), las enzimas y los genes que los codifican.
En general, cada etapa en una vía metabólica es catalizada por una enzima (identificada bioquímicamente) que es el producto de un gen particular (identificado por mutantes incapaces de sintetizar el producto final, o incapaces de descomponer el compuesto de partida, de una ruta). El número de genes que pueden generar mutantes auxotróficos es (generalmente) el mismo que el número de etapas enzimáticas en la ruta. A los mutantes auxotróficos en un gen dado les falta la enzima correspondiente. Así, Beadle y Tatum concluyeron que un gen codifica una enzima. A veces se requiere más de un gen para codificar una enzima porque la enzima tiene múltiples subunidades polipeptídicas diferentes. Así, cada polipéptido es codificado por un gen.
Los intermedios metabólicos que se acumulan en cada mutante pueden usarse para colocar las enzimas en su orden de acción en una vía. En el diagrama de la Figura 1.15, los mutantes en el gen 3 acumularon la sustancia O. Alimentando la sustancia O a mutantes en el gen 1 o en el gen 2 permite el crecimiento en ausencia de Arg. Se concluye que los defectos en la enzima 1 o la enzima 2, respectivamente, están aguas arriba de la enzima 3. En contraste, alimentar la sustancia O a mutantes en el gen 4 no permitirá el crecimiento en ausencia de Arg. A pesar de que este mutante puede convertir la sustancia O en sustancia P, no tiene una enzima activa 4 para convertir P en Arg. La incapacidad de los mutantes en el gen 4 para crecer sobre la sustancia O muestra que la enzima 4 está aguas abajo de la enzima 3.
Imagina que estás estudiando la biosíntesis de serina en un hongo. Aíslas auxótrofos de serina, haces todos los cruces por pares de los mutantes y descubres que los auxótrofos pueden agruparse en tres grupos de complementación, llamados A, B y C. También descubres que un intermedio metabólico diferente se acumula en miembros de cada grupo de complementación - sustancia A en auxótrofos en el grupo de complementación A, sustancia B en el grupo de complementación B y sustancia C en el grupo de complementación C. Cada uno de los intermedios se alimenta a auxótrofos de cada uno de los tres grupos de complementación como se tabula a continuación. A + significa que el auxótrofo pudo crecer en medio en ausencia de serina cuando se alimentó con la sustancia indicada; a - denota no crecimiento en ausencia de serina.
| Fed: | mutante en el grupo de complementación A | mutante en el grupo de complementación B | mutante en el grupo de complementación C |
|---|---|---|---|
| sustancia A | - | + | + |
| sustancia B | - | - | - |
| sustancia C | - | + | - |
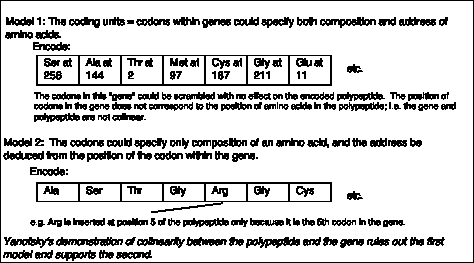
Los ARNm eucariotas tienen unión covalente de nucleótidos en los extremos 5' y 3', y en algunos casos los nucleótidos se agregan internamente (un proceso llamado edición de ARN). Un trabajo reciente muestra que también se agregan nucleótidos adicionales después de la transcripción a algunos ARNm bacterianos.
Las señales reguladoras pueden considerarse partes de genes
Para expresar un gen en el momento correcto, el ADN también transporta señales para iniciar la transcripción (por ejemplo, promotores), señales para regular la eficiencia de iniciar la transcripción (por ejemplo, operadores, potenciadores o silenciadores) y señales para detener la transcripción (por ejemplo, terminadores). Como mínimo, un gen incluye la unidad de transcripción, que es el segmento de ADN que se copia en ARN en el transcrito primario. Las señales que dirigen a la ARN polimerasa para comenzar en el sitio correcto, y otros segmentos de ADN que influyen en la eficiencia de este proceso son elementos reguladores para el gen. También se pueden considerar que forman parte del gen, junto con la unidad de transcripción.
Un problema contemporáneo: encontrar la función de los genes
Los genes fueron detectados originalmente por el fenotipo heredable generado por sus alelos mutantes, como los ojos blancos en la Drosophila normalmente de ojos rojos o la forma de células falciformes de la hemoglobina (HB) en humanos. Ahora que tenemos la capacidad de aislar prácticamente cualquier, y quizás todos, segmentos de ADN del genoma de un organismo, surge la cuestión de cuáles de esos segmentos son genes, y cuál es la función de esos genes. (El genoma es todo el ADN en los cromosomas de un organismo.) Los genetistas anteriores sabían cuál era la función de los genes que estaban estudiando (al menos en términos de algún fenotipo macroscópico), incluso cuando no tenían idea de cuál era la naturaleza del material genético. Ahora los biólogos moleculares se enfrentan al problema opuesto: podemos encontrar y estudiar mucho ADN, pero ¿qué regiones son funciones? Se están desarrollando muchos enfoques computacionales para guiar en este análisis, pero eventualmente volvemos a esa definición clásica, es decir, que las mutaciones apropiadas en cualquier gen funcional deberían generar un fenotipo detectable. El enfoque de hacer bioquímicamente mutaciones en el ADN en el laboratorio y luego probar los efectos en células vivas u organismos completos se llama “genética inversa”.
Lecturas adicionales
- Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin, R. C. y Gelbart, W. M. (1993) An Introduction to Genetic Analysis, Quinta Edición (W. H. Freeman and Company, Nueva York).
- Cairns, J., Stent, G. S. y Watson, J. D., editores (1992) Phage and the Origins of Molecular Biology, Edición Expandida (Cold Spring Harbor Laboratory Press, Plainview, NY).
- Brock, T. D. (1990) La emergencia de la genética bacteriana (Cold Spring Harbor Laboratory Press, Plainview, NY).
- Benzer, S. (1955) Estructura fina de una región genética en bacteriófago. Actas de la Academia Nacional de Ciencias, EUA 47:344-354.
- Yanofsky, C. (1963) Reemplazos de aminoácidos asociados con mutación y recombinación en el gen A y su relación con datos de codificación in vitro. Simposios de Cold Spring Harbor sobre Biología Cuantitativa 18:133-134.
- Crick, F. (1970) Dogma central de la biología molecular. Naturaleza 227:561-563
Preguntas
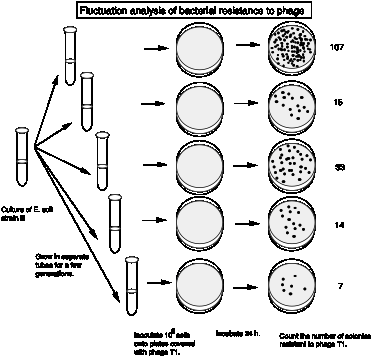
Los resultados reales de Luria y Delbrück se resumen en la siguiente tabla. Examinaron 87 cultivos, cada uno con 0.2 ml de bacterias, para colonias resistentes a fagos.
| Número de bacterias resistentes | Número de culturas |
| 0 | 29 |
| 1 | 17 |
| 2 | 4 |
| 3 | 3 |
| 4 | 3 |
| 5 | 2 |
| 6-10 | 5 |
| 11-20 | 6 |
| 21-50 | 7 |
| 51-100 | 5 |
| 101-200 | 2 |
| 201-500 | 4 |
| 501-1000 | 0 |
Los estudiantes interesados tal vez deseen leer sobre el reexamen del origen de las mutaciones por Cairns, Overbaugh y Miller (1988, The origin of mutants. Naturaleza 335:142-145). Utilizando un agente selectivo no letal (lactosa), obtuvieron resultados que indican tanto mutaciones preadaptativas (espontáneas) como algunas aparentemente inducidas por el agente selectivo.


