3.1: ADN recombinante, reacción en cadena de la polimerasa y aplicaciones a la estructura y función de genes eucariotas
- Page ID
- 58676
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
Los métodos para purificar algunas proteínas abundantes se desarrollaron a principios del siglo XX, y algunos de los experimentos sobre la estructura fina del gen (colinealidad del gen y la proteína para trp A y triptófano sintasa) utilizaron genética microbiana y secuenciación de proteínas. Sin embargo, los métodos para aislar genes no se desarrollaron hasta la década de 1960, y los fueron aplicables solo a unos pocos genes.
Todo esto cambió a finales de la década de 1970 con el desarrollo de la tecnología de ADN recombinante, o clonación molecular. Esta técnica permitió a los investigadores aislar cualquier gen de cualquier organismo del que se pudiera aislar ADN (o ARN) intacto. El potencial completo para proporcionar acceso a todos los genes de los organismos se está realizando ahora a medida que se secuencian los genomas completos. Uno de los subproductos de la intensa investigación de moléculas de ADN individuales después del advenimiento del ADN recombinante fue un procedimiento para aislar cualquier ADN para el que se conozca la secuencia. Esta técnica, llamada reacción en cadena de la polimerasa (PCR), es mucho más fácil que los métodos tradicionales de clonación molecular, y se ha convertido en un elemento básico de muchos laboratorios en las ciencias de la vida. Después de cubrir las técnicas básicas en tecnología de ADN recombinante y PCR, se discutirá su aplicación a estudios de estructura y función de genes eucariotas.
Al igual que muchos avances en genética molecular, la tecnología del ADN recombinante tiene sus raíces en la genética bacteriana.
Fago transduciendo
Los primeros genes aislados fueron genes bacterianos que podrían ser captados por bacteriófagos. Al aislar estos bacteriófagos híbridos, el ADN para el gen bacteriano podría recuperarse en una forma altamente enriquecida. Este es el principio básico detrás de la tecnología de ADN recombinante.
Algunos bacteriófagos se integrarán en un cromosoma bacteriano y residirán en estado latente (Figura 3.1). El ADN del fago integrado se llama profago, y la bacteria ahora es un lisógeno. Los fagos que hacen esto son lisogénicos. La inducción del lisógeno dará como resultado la escisión del profago y la multiplicación para producir muchas progenie, es decir, entra en una fase lítica en la que las bacterias se abren y destruyen. La nomenclatura es descriptiva. Las bacterias portadoras del profago no muestran signos obvios del fago (excepto inmunidad a la superinfección con el mismo fago, cubierta más tarde en la Cuarta Parte), pero cuando se inducen (por ejemplo, por estrés o radiación UV) generarán un estado lyti c, de ahí que se les llame lisógenos. Los lisógenos inducidos producen fagos a partir del profago que se integró. Los fagos que siempre se multiplican cuando infectan una célula se llaman líticos.
La escisión de un profago de un lisógeno no siempre es precisa. Por lo general, solo se corta el ADN del fago del cromosoma bacteriano, pero de vez en cuando se incluye algún ADN hospedador adyacente con el ADN del fago escindido y se encapsida en la progenie. Estos fagos transductores suelen ser biológicamente inactivos debido a que la pieza del cromosoma bacteriano reemplaza parte del cromosoma del fago; estos pueden propagarse en presencia de fagos cooperadores que proporcionan los genes faltantes cuando se coinfectan en la misma bacteria. Cuando el ADN del fago transducidor se inserta en la célula recién infectada, los genes bacterianos pueden recombinarse en el cromosoma huésped, trayendo así nuevos alelos o incluso nuevos genes y alterando genéticamente la célula infectada. Este proceso se llama transducción.
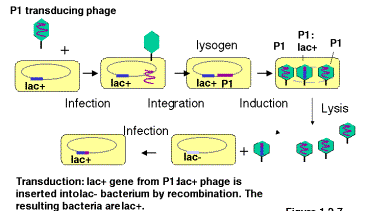
Obsérvese que los fagos transductores son portadores de uno o un pequeño número de genes bacterianos. Esta es una forma de aislar los genes. El gen bacteriano en el fago transducidor se ha separado de los otros 4000 genes bacterianos (en E. coli). Mediante el aislamiento de grandes cantidades de fagos transductores, el ADN del fago, incluyendo los genes bacterianos, se puede obtener en grandes cantidades para la investigación bioquímica. Se pueden aislar cantidades de mg o mg de una sola molécula de ADN, lo que permite una determinación estructural precisa y una investigación detallada.
Un fago transducidor generalizado puede integrarse en muchas ubicaciones diferentes en el cromosoma bacteriano. La escisión imprecisa de cualquiera de esas localizaciones genera un fago transducidor particular, portando secciones cortas del genoma bacteriano adyacentes al sitio de integración. Así, un fago transducidor generalizado como P1 puede recoger muchas partes diferentes del genoma de E. coli.
Un fago de transducción especializado se integra en solo uno o muy pocos sitios en el genoma del huésped. De ahí que pueda llevar solo unos pocos genes bacterianos específicos, por ejemplo, l lac (Figura 3.2).

Este proceso de aislamiento de un gen bacteriano particular en un fago transducible se mimetiza en la tecnología de ADN recombinante, en la que se aísla un gen o fragmento de genoma de cualquier organismo en un fago o plásmido recombinante.


