Mecanismos básicos de replicación
- Page ID
- 58459
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\){{Template.dropdown {path:” /Genetics/ "}}}
La replicación del ADN es semiconservativa
Comenzamos nuestra investigación describiendo el modelo básico de cómo se unen los nucleótidos en un orden específico durante la replicación del ADN. A principios de la década de 1950, estaba claro que el ADN era una cadena lineal de desoxirribonucleótidos. En ese punto, se podrían postular tres formas diferentes de replicar el ADN de una célula. Primero, una célula podría tener una “máquina” sintetizadora de ADN que podría programarse para hacer una cadena particular de nucleótidos para cada cromosoma. Una segunda posibilidad es que el proceso de replicación pueda romper el ADN parental en pedazos y utilizarlos para sembrar la síntesis de nuevo ADN.
Se podría proponer un tercer modelo a partir de la estructura del ADN deducida por Watson y Crick. Cuando describieron la estructura de doble hélice del ADN en un artículo de una página en Nature en 1953, incluyeron esta breve declaración de un tercer modelo:
“No ha escapado a nuestro aviso que el emparejamiento específico que hemos postulado sugiere de inmediato un posible mecanismo de copia para el material genético”.
En un trabajo posterior se elaboró este mecanismo. La complementariedad entre pares de bases (A con T y G con C) no sólo mantiene unidas las dos hebras de la doble hélice, sino que la secuencia de una hebra es suficiente para determinar la secuencia de la otra. De ahí que una tercera posibilidad de un mecanismo de replicación del ADN era clara: una cadena parental podría servir como atemplate dirigiendo la síntesis de una cadena complementaria en las moléculas de ADN hijas. Este artículo de 1953 es, por supuesto, el más famoso por su descripción de la estructura de doble hélice del ADN que se mantiene unida por la complementariedad de bases, pero también es importante porque la estructura propuesta sugiere un modelo comprobable de cómo ocurre un proceso particular, en este caso la replicación.
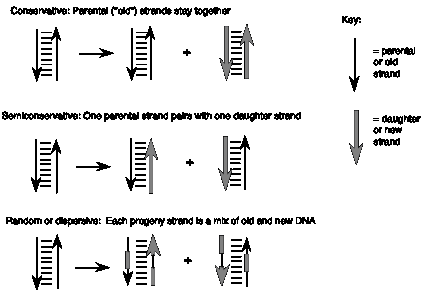
Estos tres modelos hacen diferentes predicciones sobre el comportamiento de las dos cadenas del ADN parental durante la replicación (Figura 5.1). En el primer modelo de máquina programado, las dos hebras del ADN parental pueden permanecer juntas, ya que no son necesarias para determinar la secuencia de las hebras hijas. Este modelo de replicación se llama conservador: las moléculas de ADN parentales son las mismas en la progenie que en la célula parental. En el segundo modelo, cada hebra de las moléculas de ADN hijas sería una combinación de ADN antiguo y nuevo. Este tipo de replicación se conoce como aleatoria (o dispersiva). El tercer modelo, en el que una hebra del ADN parental sirve como molde que dirige el orden de los nucleótidos en la nueva cadena de ADN, es un modo semiconservativo de replicación, ya que la mitad de cada dúplex parental (es decir, una cadena) permanece intacta en las moléculas hijas.
Cuando eran estudiantes de posgrado en el Instituto Tecnológico de California, Matthew Meselson y Franklin Stahl se dieron cuenta de que podían probar estos tres modelos para su replicación distinguiendo experimentalmente entre hebras viejas y nuevas de ADN. Marcaron el ADN antiguo o parental con nucleótidos compuestos por un isótopo pesado de nitrógeno (15 N) cultivando células de E. coli durante varias generaciones en medios que contenían [15 N] NH4Cl. El amoníaco es un precursor en la biosíntesis de las bases de purina y pirimidina, y de ahí que este procedimiento marcara el nitrógeno en las bases nucleotídicas en el ADN de las células de E. coli con 15 N. Posteriormente, las células se desplazaron para crecer en medios que contenían la luz altamente abundante isótopo de nitrógeno, 14 N, en el NH4Cl, de manera que el ADN recién sintetizado tendría una densidad “ligera”. El ADN marcado, pesado (viejo) podría separarse del ADN ligero (nuevo) no marcado en un gradiente de densidad de CsCl, en el que las bandas de ADN en la posición en el gradiente donde la concentración de CsCl tiene una densidad igual a la de la macromolécula. En tiempos progresivos después del cambio al crecimiento en [14 N] NH4Cl, se recolectaron muestras de las células, luego se aisló el ADN de las células y se separó en un gradiente de CsCl.


A. B.
Figura 5.2. Resultados del experimento Meselson y Stahl demostrando replicación semiconservativa del ADN. A. El panel izquierdo (a) muestra fotografías de absorción ultravioleta del ADN después de la sedimentación en equilibrio en un gradiente de CsCl, en función del número de generaciones desde el cambio de medios que marcaron ADN con una alta densidad (15 N marcado) a un medio en el que el ADN es normal, o densidad de luz (14 N-ADN). La densidad del gradiente de CsCl aumenta hacia la derecha. El panel de la derecha (b) muestra una traza de la cantidad de ADN a lo largo del gradiente. El número de generaciones desde el cambio a los medios con 14 sustratos N se muestra en el extremo derecho. Los experimentos de mezcla en la parte inferior muestran las posiciones de ADN uniformemente ligero y pesado (generaciones 0 y 4.1 mezclados) y la mezcla de aquellos más ADN híbrido ligero y pesado (generaciones 0 y 1.9 mezclados). El ADN parental forma una banda a la densidad pesada (marcada con 15 N), mientras que después de una generación en medios ligeros (14 N), todo el ADN forma una banda a una densidad híbrida (entre pesada y ligera). El crecimiento continuo en medios ligeros conduce a la síntesis de ADN que es solo densidad lumínica. B. La interpretación de los resultados experimentales como demostración de un modelo semiconservativo de replicación. La parte A de esta figura es la figura 4 y la parte B es la figura 6 de M. Meselson y F. Stahl (1958) “The Replication of DNA in Escherichia coli” Proceedings of the National Academy of Sciences, USA 44:671-682.
Los resultados se ajustan al patrón esperado para la replicación semiconservativa (Figura 5.2). Para citar de Meselson y Stahl, “hasta que haya transcurrido un tiempo de generación, las moléculas semitarcadas se acumulan, mientras que el ADN completamente etiquetado se agota. Una generación de tiempo después de la adición de 14 N, se observan estas moléculas semitarcadas o 'híbridas' solas. Posteriormente, solo se encuentran ADN semicarcado y ADN completamente sin marcar. Cuando han transcurrido dos tiempos de generación después de la adición de 14 N, el ADN semimarcado y no marcado están presentes en cantidades iguales”. Se descarta un modo conservador de replicación por la observación de que todo el ADN formó una banda a una densidad híbrida después de una generación en el medio que contenía [14N] NH4Cl. Sin embargo, es consistente con los modelos semiconservativos o aleatorios. Como se esperaba para la replicación semiconservativa, la mitad del ADN estaba a una densidad híbrida y la mitad a una densidad de luz después de dos generaciones en medio que contenía [14N] NH4Cl. Un mayor crecimiento en el medio 14N resultó en un incremento en la cantidad de ADN en la banda LL.
Ejercicio
Pregunta 5.1: ¿Qué datos de este experimento descartan un modo aleatorio de replicación?
Estos experimentos demostraron que cada cadena de ADN parental se usa como molde que dirige la síntesis de una nueva cadena durante la replicación del ADN. La síntesis de nuevo ADN está dirigida por la complementariedad de bases. Las enzimas que llevan a cabo la replicación no son “máquinas” programadas con una especificidad inherente para sintetizar una secuencia dada, sino que la cadena molde de ADN determina el orden de nucleótidos a lo largo de la cadena de ADN recién sintetizada (Figura 5.3).

La asociación de una cadena de ADN parental con una cadena de ADN recién sintetizada observada en este importante resultado experimental es consistente con el uso de cada cadena de ADN parental como molde para dirigir la maquinaria de replicación para colocar los nucleótidos en un orden particular. Watson y Crick propusieron que la complementariedad base guiaría la maquinaria de replicación para insertar una A opuesta a una T, una T opuesta a una A, una G opuesta a una C y C opuesta a una G (Figura 5.3). Esto se verificó una vez que se aislaron las enzimas que realizan la síntesis de ADN, y se comparó la composición química de los productos de replicación con la de los moldes. Estas enzimas se discuten en detalle más adelante en el capítulo, así como lo será la química del proceso de agregar nucleótidos individuales a la cadena de ADN en crecimiento (un proceso llamado elongación). Cabe recordar que estas enzimas también se utilizaron para demostrar la disposición antiparalela de las cadenas de ADN predichas por Watson y Crick (problema de recuerdo 2.5). Con esta comprensión de cómo se especifica la secuencia de nucleótidos, podemos examinar los tipos de estructuras de ADN que se encuentran durante la replicación.


