8.10: Sinapsis e Invasión de Hilos Individuales
- Page ID
- 58563
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
El emparejamiento de las dos moléculas de ADN recombinantes (sinapsis) y la invasión de una sola cadena desde el dúplex de inicio hacia el otro dúplex son catalizadas por la proteína multifuncional RecA. Esta invasión del ADN dúplex por un ADN monocatenario da como resultado el reemplazo de una de las cadenas del dúplex original por la cadena invasora, y la cadena reemplazada se desplaza del dúplex. De ahí que esta reacción también se pueda denominar asimilación de cadenas o intercambio de cadenas. RecA tiene muchas actividades, incluyendo estimular la función proteasa de LexA y uMUD (ver Capítulo 7), unirse y recubrir ADN monocatenario, estimular el emparejamiento homólogo entre ADN monocatenario y dúplex, asimilar ADN monocatenario en un dúplex y catalizar la hidrólisis de ATP en presencia de ADN (es decir, es una ATPasa dependiente de ADN). Se requiere en las 3 vías para la recombinación. Por ejemplo, la molécula de ADN con un extremo 3' monocatenario generada por la enzima recBCD puede asimilarse en una región homóloga de otro dúplex, catalizada por RecA y que requiere la hidrólisis de ATP (Figura 8.14).
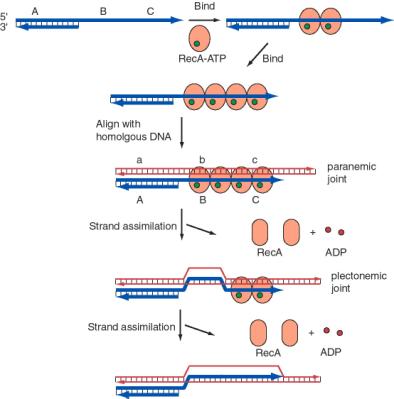
El proceso de asimilación monocatenaria ocurre en tres etapas, como se ilustra en la Figura 8.15. Primero, RecA polimeriza sobre ADN monocatenario en presencia de ATP para formar el filamento presináptico. La cadena única de ADN se encuentra dentro de un surco profundo de la proteína RecA, y muchas moléculas de recA-ATP recubren el ADN monocatenario. Una molécula de la proteína RecA cubre de 3 a 5 nucleótidos de ADN monocatenario. Los nucleótidos se extienden axialmente por lo que están separados aproximadamente 5 Angstroms en el ADN monocatenario, aproximadamente 1.5 veces más largos que en ausencia de RecA-ATP.
A continuación, el filamento presináptico se alinea con regiones homólogas en el ADN dúplex. Una longitud sustancial de las tres cadenas se mantienen unidas por un polúmero de moléculas de RecA-ATP. El dúplex alineado y la hebra simple forman una articulación paranémica, lo que significa que la hebra simple no está entrelazada con la doble hebra en este punto. El ADN dúplex, al igual que el ADN monocatenario, se extiende hasta aproximadamente 1.5 veces más que en el ADN de forma B normal (18.6 pb por giro). Se cree que esta extensión es importante en el emparejamiento homólogo.
Finalmente, las hebras se intercambian para formar una articulación plectonémica. En esta etapa, la hebra simple invasora ahora se entrelaza con la hebra complementaria en el dúplex, y una hebra del dúplex invadido ahora está desplazada. En E. coli, el intercambio ocurre en una dirección 5' a 3' con respecto a la cadena simple y requiere hidrólisis de ATP. En contraste, el homólogo de levadura, Rad51, hace que la cadena sencilla invada con la polaridad opuesta, es decir, 3' a 5'. Así, la dirección de esta polaridad no es una característica universalmente conservada de los mecanismos de recombinación.
El producto de la asimilación de la cadena es un heterodúplex en el que una cadena del dúplex era el ADN monocatenario original. La otra hebra del dúplex original está desplazada.
Muchos detalles de la actividad de RecA han sido revelados por ensayos in vitro para la asimilación monocatenaria o el intercambio de cadenas. Los sustratos de ADN para el intercambio de cadenas catalizado por RecA deben cumplir tres requisitos. Debe haber una región de ADN monocatenario en la que RecA pueda unirse y polimerizarse, las dos moléculas que experimentan intercambio de cadenas deben tener una región de homología, y debe haber un extremo libre dentro de la región de homología. Este último requisito puede superarse proporcionando una topoisomerasa.
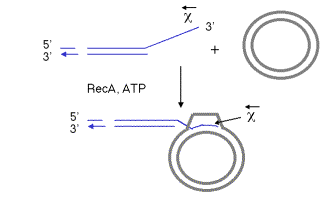
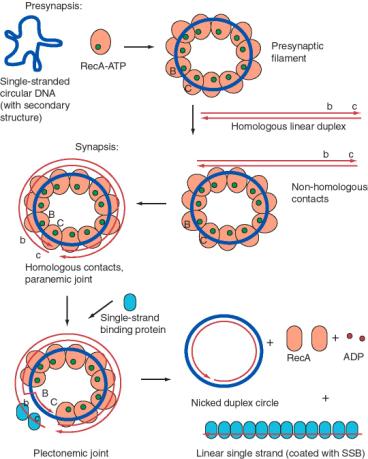
Uno de esos ensayos es la conversión de un ADN circular monocatenario en un círculo dúplex (Figura 8.16). Los sustratos para esta reacción son un ADN monocatenario circular y un dúplex lineal homólogo. Estos se mezclan en presencia de RecA y ATP. Muchas moléculas de recA-ATP recubren el círculo monocatenario para formar el filamento presináptico de nucleoproteína, como se discutió anteriormente. Durante la sinapsis, la reasociación se inicia con el extremo 3' de la cadena complementaria al círculo monocatenario. Así, la hebra sencilla invade con polaridad de 5' a 3' (con referencia a su propia polaridad). El desplazamiento de la cadena, impulsado por la hidrólisis de ATP para disociar la RecA, da como resultado la formación de un círculo mellado (una hebra del cual era el círculo monocatenario original) y una cadena simple lineal de ADN.
Ejercicio
Trate de relacionar este ensayo in vitro con los pasos en el modelo de doble hebra para recombinación. ¿Qué paso (s) en el modelo imita esto? ¿Qué más se necesita para llegar a las articulaciones recombinantes (uniones Holliday)?
La estructura de recA de E. coli unida por ADP, tanto monómero como polímero, se ha resuelto mediante cristalografía de rayos X. Como se muestra en la Figura 8.17, el dominio central tiene el sitio de unión para ATP y ADP, y es presumiblemente el sitio de unión del ADN monocatenario y bicatenario. Los dominios que se extienden lejos de la región central están involucrados en la polimerización de proteínas RecA y en las interacciones entre las fibras presinápticas.

Las proteínas homólogas a la recA de E. coli se encuentran en levaduras (Rad51 y Dmc1) y en ratones (Rad51). Dada la universalidad de la recombinación, es probable que se encuentren homólogos en prácticamente todas las especies. Las mutaciones en el gen recA de E. coli reducen la recombinación conjugacional hasta en 10,000 veces, por lo que es evidente que RecA juega un papel central en la recombinación. Sin embargo, las mutaciones nulas en RecA no son letales, ni las mutaciones nulas en los homólogos de levadura RAD51 y DMC1. En contraste, los ratones homocigotos para una mutación knockout en el gen Rad51 mueren muy temprano en el desarrollo, en la etapa de 4 células. Esto indica que en ratones, este homólogo de RecA está desempeñando un papel novedoso en la replicación o reparación, presumiblemente además de su papel en la recombinación.


