11: Transcripción: Promotores, terminadores y ARNm
- Page ID
- 58573
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)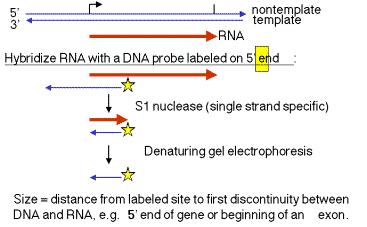
1. “Ensayo de protección S1"
Este ensayo mide la distancia entre un marcador final (en un sitio conocido específico en el ADN) y el extremo de un dúplex entre el ARN y el ADN marcado. Un fragmento de ADN (complementario al ARN) que se extiende más allá del extremo 5' del ARN se marca en un sitio de restricción dentro de la región complementaria del ARN. El ADN marcado se hibrida con el ARN y luego se digiere con la nucleasa S1 específica monocatenaria. El fragmento resultante de ADN protegido se ejecuta en un gel desnaturalizante para determinar su tamaño. Obsérvese que este fragmento va desde el sitio marcado hasta la interrupción más cercana entre el ADN y el ARN. Esto podría ser el comienzo del ARN, o podría ser un intrón, o podría ser un sitio sensible a S1.
3. ¿Cómo se etiqueta el ADN en los extremos?
- Etiqueta en el extremo 5': polinucleótido quinasa T4 y [g 32P] ATP. La reacción es más eficiente si se elimina el fosfato 5' (mediante fosfatasa alcalina) antes del tratamiento con quinasa.
- Etiqueta en el extremo 3': ADN polimerasa de Klenow más [a 32P] dNTP. El dNTP marcado se elige para que sea complementario a la primera posición más allá del cebador. Un fragmento de restricción con un saliente 5' es ideal para este etiquetado de “relleno”.
- La digestión con una segunda endonucleasa de restricción frecuentemente funcionará para eliminar la etiqueta en el “otro” extremo. También se pueden usar geles electroforéticos que separan hebras.
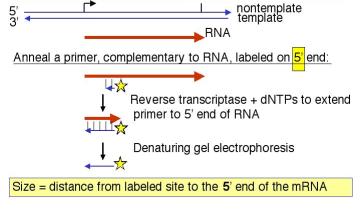
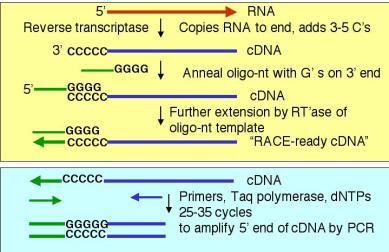
4. Una técnica basada en PCR para determinar los extremos 5' de ARNm y genes
B. Métodos generales para identificar el sitio para proteínas de unión específicas de secuencia
1. ¿Una proteína se une a una región en particular?
a. Ensayo de desplazamiento de movilidad electroforética (EMSA), o ensayo de retraso en gel: Este ensayo probará la capacidad de una secuencia particular para formar un complejo con una proteína. Muchos complejos de proteína-ADN son lo suficientemente estables como para que permanezcan juntos durante la electroforesis a través de un gel de poliacrilamida (no desnaturalizante). Un fragmento de restricción seleccionado o oligonucleótido dúplex sintético se marca (para hacer una sonda) y se mezcla con una proteína (o mezcla bruta de proteínas). Si el fragmento de ADN se une a la proteína, el complejo migrará mucho más lento en el gel que la sonda libre; se mueve aproximadamente con la movilidad de la proteína unida. La presencia de una señal que se mueve lentamente es indicativa de un complejo entre la sonda de ADN y algunas proteínas. Al incubar la sonda y las proteínas en presencia de cantidades crecientes de fragmentos de ADN de la competencia, se puede probar la especificidad e incluso obtener cierta información sobre la identidad de la proteína de unión.
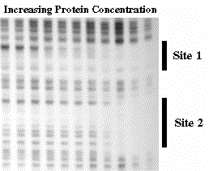
Figura 3.2.4. Diagrama de resultados de un ensayo de desplazamiento de movilidad electroforética

En este ejemplo, dos proteínas reconocen secuencias en la sonda marcada, formando los complejos A y B (carril 2). Las proteínas en los complejos A y B reconocen secuencias específicas de ADN en la sonda. Esto lo demuestran los ensayos de competencia en los carriles 3-8. Un exceso de oligonucleótido no marcado con la misma secuencia que la sonda marcada (“self”) evita la formación de los complejos con la sonda marcada, mientras que el “ADN inespecífico” en forma de ADN de E. coli no compite eficazmente (comparar los carriles 6-7 con los carriles 3-5).
Este experimento también proporciona cierta información sobre la identidad del complejo A formador de proteínas. Reconoce un sitio de unión a SP1, como lo demuestra la capacidad de un oligonucloetida con una unión a SP1 para competir por el complejo A, pero no por el complejo B (carriles 9-11). De ahí que la proteína podría ser Sp1 o un pariente de la misma. Las proteínas que forman los complejos A y B no reconocen un sitio de unión a OCT1 (carriles 12-14).
b. Unión a nitrocelulosa: El ADN dúplex libre no se adherirá a una membrana de nitrocelulosa, sino que se unirá un complejo proteína-ADN.
2. ¿A qué secuencia en el ADN sonda se une la proteína?
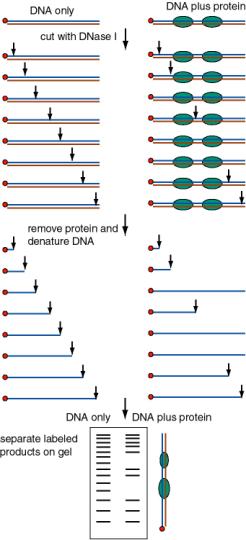
La presencia de una proteína protegerá un segmento de ADN del ataque de una nucleasa u otro reactivo degradante, o en algunos casos potenciará la escisión (por ejemplo, a una secuencia adyacente que está distorsionada de la forma B normal). Un fragmento de ADN marcado en el extremo en complejo con proteína se trata con una nucleasa (u otro reactivo de escisión), y los fragmentos protegidos se resuelven en un gel de poliacrilamida desnaturalizante y se miden sus tamaños.
- a. Ensayo de protección de exonucleasa: La proteína bloqueará el progreso de una exonucleasa, por lo que el fragmento protegido se extiende desde el sitio marcado hasta el borde de la proteína más lejos del sitio marcado. Se puede usar una combinación de una exonucleasa 3' a 5' (ExoIII) y una exonucleasa 5' a 3' (l exonucleasa) para mapear ambos bordes.
- b. Análisis de huella de DNasa: DNasa I cortará en muchos (pero no todos) enlaces fosfodiéster en el ADN libre. El complejo proteína-ADN se trata ligeramente con DNasa I, de manera que en promedio cada molécula de ADN se escinde una vez. La presencia de una proteína unida bloqueará el acceso de la DNasa, y la región unida será visible como una región del gel que no tiene bandas, es decir, que no fue cortada por el reactivo.
4. Cromatografía de afinidad de secuencia de ADN para purificar proteínas de unión a ADN
Los sitios de unión específicos (a menudo de 6 a 8 pb) pueden servir como un ligando de afinidad para la cromatografía. Los multímeros del sitio de unión se preparan uniendo oligonucleótidos dúplex que contienen el sitio específico. Después de algunos pasos iniciales crudos (por ejemplo, aislar todas las proteínas de unión a ADN en DNA-Sepharose) el extracto se aplica a la columna de afinidad. La mayoría de las proteínas no se unen, y posteriormente se eluyen las proteínas unidas específicamente.
C. Promotores e Iniciación de la Transcripción: Propiedades Generales
- Un promotor es la secuencia de ADN requerida para el correcto inicio de la transcripción
- Fenotipo de mutantes promotores
a. Acción cis: Un elemento regulador que actúa en cis funciona como un segmento de ADN para afectar la expresión de genes en el mismo cromosoma en el que se encuentra. Los elementos que actúan en cis no codifican un producto difusible. El promotor es un elemento regulador que actúa en cis.
Comparar los fenotipos de mutaciones en el gen que codifica b‑galactosidasa (lacZ) versus mutaciones en su promotor (p).
Considera un heterocigoto que sea p + LacZ ‑/p + LacZ +.
El fenotipo es Lac+. LacZ + complementa LacZ ‑ en trans. En este caso, LacZ + es dominante a lacZ-.
Considera un heterocigoto que sea p + LacZ ‑/p ‑ LacZ +.
El fenotipo es Lac‑. p + no complementa p ‑ en trans.
p ‑ opera en cis para prevenir la expresión de lacZ + en este cromosoma. El promotor mutante es dominante sobre el tipo silvestre cuando el promotor mutante está en ci s al wt lacZ.
Considera un heterocigoto que sea p + LacZ +/p ‑ lacZ ‑.
El fenotipo es Lac+. LacZ + ahora complementa lacZ ‑ en trans porque es impulsado por un promotor funcional en ci s, p +
b. dominancia en cis: el promotor “alelo” que está en cis al gen estructural de tipo silvestre (lacZ) es dominante sobre el otro alelo promotor.
c. Las mutaciones promotoras afectan la cantidad de producto del gen pero no afectan la estructura del producto génico.
D. Promotores bacterianos
- Los promotores bacterianos ocurren justo 5' y se solapan con el sitio de inicio para la transcripción (generalmente)
- Los promotores bacterianos son el sitio de unión para la holoenzima ARN polimerasa de E. coli. El promotor cubre aproximadamente 70 pb de aproximadamente ‑50 a aproximadamente +20.
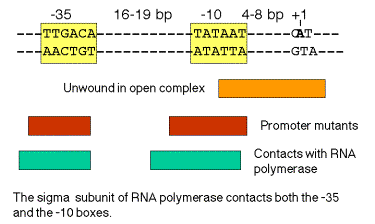
- Secuencias consenso en el promotor de E. coli
a. ‑35 y ‑10 secuencias
‑35 16‑19 pb ‑10+1
‑‑‑‑‑‑‑‑‑TTGACA‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑‑—TATAAT‑‑‑‑C A T
Reconocimiento por Permite convertir el complejo binario
ARN polimerasa de cerrado a abierto
holoenzima
b. Las secuencias se conservan en todos los genes de E. coli transcritos por holoenzima con s70
4. Mutantes promotores
- a. Tienden a caer dentro o cerca de uno de estos hexanucleótidos
- b. Afectar el nivel de expresión génica, no la estructura del producto génico
- c. Mutaciones del promotor descendente: disminuir el nivel de transcripción. Tienden a hacer que la secuencia promotora sea menos parecida al consenso.
- d. Mutaciones promotoras arriba: aumentar el nivel de transcripción. Tienden a hacer que la secuencia promotora se parezca más al consenso.
- e. Mutaciones del promotor descendente en la secuencia ‑35: disminuyen la tasa de formación del complejo cerrado, lo que indica que esta es la secuencia necesaria para el reconocimiento inicial por la holoenzima polimerasa.
- f. Mutaciones del promotor descendente en la secuencia ‑10: disminuir la tasa de conversión del complejo cerrado al abierto, apoyando nuevamente el papel propuesto para este hexanucleótido rico en A+T.
- g. Los puntos de contacto críticos entre la ARN polimerasa y el promotor tienden a estar en o inmediatamente aguas arriba de los recuadros de consenso ‑35 y ‑10. (Ver Figura 3.2.7). Así, los datos bioquímicos y genéticos apoyan la importancia de estas secuencias conservadas.
Figura 3.2.7. Correlación de secuencias conservadas, localización de mutantes promotores y regiones de contacto con polimerasa en promotores bacterianos
5. Los factores alternos pueden controlar la expresión de conjuntos de genes
- a. Los factores alternativos hacen complejos con la polimerasa central para dirigir la nueva holoenzima a un conjunto particular de promotores que difieren en secuencia de la secuencia promotora general de E. coli. Así, la polimerasa puede ser dirigida a transcribir un nuevo conjunto de genes. Esta es una forma de controlar la expresión génica.
- b. Los ejemplos incluyen factores s para la respuesta al choque térmico (s32), transcripción de genes involucrados en la quimiotaxis y formación flagelar (s28) y la inanición de nitrógeno (s54). Los factores s son nombrados por su tamaño en kDa.
- c. Tres de los factores de E. colis tienen regiones de similitud de secuencia (s70, s32 y s28) mientras que s54 es una molécula claramente diferente que funciona de manera bastante diferente.
| Factor | Gene | Uso | ‑35 | Separación | ‑10 |
| s70 | RPOD | General | TTGACA | 16-19 pb | TATAAT |
| s32 | RPoH | Choque térmico | CCCTTGAA | 13-15 pb | CCCGATNT |
| s28 | Flia | Flagelos | CTAAA | 15 pb | GCCGATAA |
| s54 | RPON | Inanición de nitrógeno | CTGGNA | 6 pb | TTGCA |
E. Promotores para ARN polimerasas eucariotas
Los promotores contienen sitios de unión para proteínas nucleares, pero ¿cuáles de estos sitios de unión tienen una función en la expresión génica? Esto requiere un enfoque genético para una respuesta.
1. Uso de la “genética sustituta” para definir el promotor
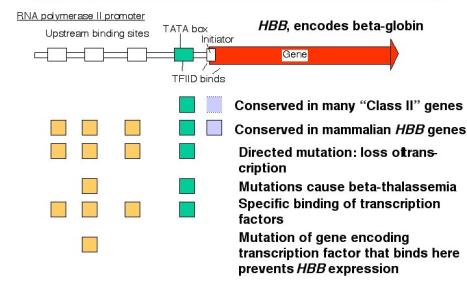
a. Mutagénesis in vitro (deleciones o mutaciones puntuales)
- Las mutaciones de los sitios de unión para proteínas activadoras conducen a una disminución en el nivel de transcripción del gen. [Pérdida de función].
- La adición de un fragmento de ADN que contiene estos sitios de unión activará (algunos) promotores heterólogos. [Ganancia de función].
- Las secuencias de los sitios de unión están frecuentemente bien conservadas en promotores para genes homólogos de especies relacionadas.
- Una región reguladora potencial se examina inicialmente construyendo deleciones progresivas desde el extremo 5' (con respecto a la dirección de transcripción) y también desde el extremo 3'. Posteriormente se pueden hacer agrupaciones de mutaciones puntuales (por ejemplo, mediante mutagénesis de barrido de enlazadores) o mutaciones puntuales individuales.
b. Prueba en un ensayo de expresión
(1) El promotor mutagenizado se une a un gen informador para que el ARN o la proteína de ese gen se pueda medir cuantitativamente
- (a) El gen mismo: medir la producción de ARN mediante protección S1, extensión de cebador u otro ensayo que sea específico para un ARN particular
- b) Gen informador heterólogo: codifica una enzima cuya actividad es fácil de medir cuantitativamente. Obsérvese que estas medidas de expresión requieren tanto transcripción como traducción, en contraste con la medición del ARN directamente. Por ejemplo, los genes que codifican:
- b‑galactosidasa: ensayo colorimétrico, controlar la escisión del o‑nitrofenil‑b‑galactósido
- cloranfenicol (Cm) acetil transferasa (CAT): medir la acetilación de Cm, usualmente usar [14C] Cm; esta es la enzima que confiere resistencia a Cm en bacterias
- luciferasa: monitorear la emisión de fotones resultantes de la oxidación de luciferina dependiente de ATP; esta es la enzima que cataliza la producción de luz en las colas de luciérnaga
(2) Los constructos de ADN promotor-informador se introducen en un sistema de ensayo que permitirá que se exprese el informador.
a) Celdas enteras
microinyección en ovocitos de Xenopus
transfección de líneas celulares: introducir el ADN mediante electroporación o haciendo que las células capten un precipitado de ADN y fosfato de Ca por pinocitosis
(b) Animales enteros = animales transgénicos
Introducir el ADN en la línea germinal de un animal, en mamíferos mediante microinyección en un óvulo fertilizado y colocándolo en una hembra pseudopreñada. Esta tecnología permite examinar los efectos de la mutación a lo largo del desarrollo del animal.
c) Sistemas libres de celdas
Extractos de núcleos, o sistemas purificados (es decir, con todos los componentes necesarios purificados)
2. Promotor para RNA Pol II
a. El promotor mínimo es necesario para la actividad basal y un inicio preciso.
- Necesario para el montaje del complejo de iniciación en el sitio correcto
- Secuencias de ADN
a) Caja TATA
- Inicialmente identificado como un motivo de secuencia bien conservado aproximadamente 25 pb 5' del sitio cap (El sitio cap es el sitio de inicio habitual para la transcripción)
- El factor de transcripción TFIID se une a la caja TATA
- Las mutaciones en la caja TATA generan extremos 5' heterogéneos de los ARNm, lo que indica una pérdida de especificidad del sitio de inicio
b) Iniciador
- Las secuencias en el sitio de inicio para la transcripción tienen consenso YANWYY (Y = C o t, W = T o A)
- El modo de acción aún está bajo investigación. Datos recientes indican que el TFIID también se une al iniciador; se une a uno de los TAF (ver abajo).
- El iniciador TATA plus es el promotor mínimo más simple.
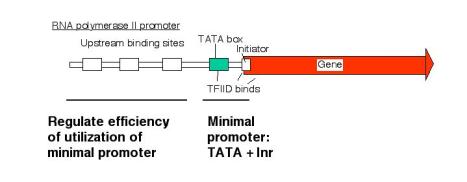
b. La cantidad de expresión se regula a través de elementos ascendentes.
- Las proteínas se unen a secuencias específicas (normalmente) 5' a la caja TATA para regular la eficiencia de utilización del promotor.
- Estos son frecuentemente activadores, pero también se están caracterizando proteínas que ejercen control negativo.
- Ejemplos de proteínas activadoras
Sp1: se une GGGGCGGGGG = caja GC
Oct n: se une a ATTTGCAT = motivo octámero
Oct1 es un factor general (ubicuo)
OcT2 es específico para células linfoides
CP1, CTF = NF1, C/EBP se unen a CCAAT = caja CCAAT (caja pronunciada “cat”)
Estas son diferentes familias de proteínas, CP1 y CTF se encuentran en muchos tipos celulares, C/EBP se encuentra en hígado y tejido adiposo.
(4) Estos elementos de control aguas arriba pueden ser inducibles (por ejemplo, por hormonas), pueden ser específicos de tipo celular, o pueden estar presentes y activos en prácticamente todos los tipos celulares (es decir, ubicuos y constitutivos).
Figura 3.2.10.

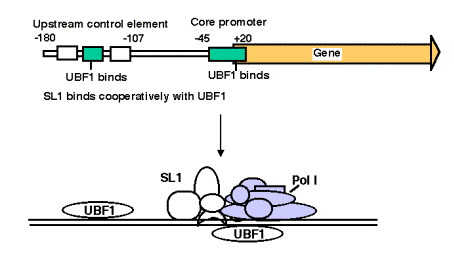
3. Promotor para ARN Pol I
- a. El promotor central cubre el sitio de inicio de la transcripción, de aproximadamente -40 a aproximadamente +30. El promotor también contiene un elemento de control aguas arriba localizado aproximadamente 70 pb más 5', extendiéndose de ‑170 a ‑110.
- b. El factor UBF1 se une a una secuencia rica en G+C tanto en el elemento control aguas arriba como en el promotor central. Un complejo multisubunidad llamado SL1 se une al complejo UBF1-DNA, nuevamente tanto en los elementos aguas arriba como en el núcleo. Una de las subuntis de SL1 es TBP.
- c. La ARN polimerasa I luego se une a este complejo de DNA+UBF1+SL1 para iniciar la transcripción en el nucleótido correcto y el alargado para hacer pre-ARNr.
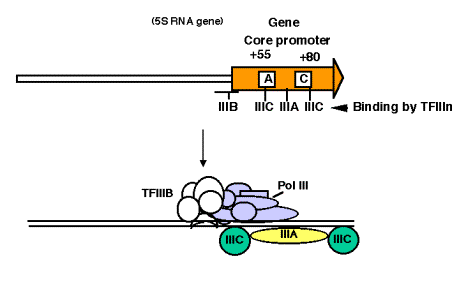
4. Promotor para ARN Pol III
a. Este promotor tiene secuencias de control interno. La deleción del ADN flanqueante 5' todavía permite la transcripción eficiente de (la mayoría) de los genes transcritos por ARN poliII. Incluso la parte inicial del gen es prescindible, al igual que el extremo 3'. Las secuencias internas al gen (por ejemplo, +55 a +80 en genes de ARNr 5S) son necesarias para una iniciación eficiente, en contraste con la situación familiar en bacterias, donde la mayoría de las secuencias promotoras están 5' con respecto al gen.
F. Potenciadores
- Los potenciadores son secuencias de ADN que provocan un aumento en el nivel de expresión de un gen con un promotor intacto. Pueden actuar para aumentar la eficiencia de utilización de un promotor, o pueden aumentar la probabilidad de que un promotor se encuentre en una conformación de cromatina transcripcionalmente competente. Esto se explorará más a fondo en la Cuarta Parte.
- Se definen operacionalmente por su capacidad para actuar en cualquier orientación y en una variedad de posiciones y distancias de un gen, es decir, actuar independientemente de la orientación y posición. Esto contrasta con promotores, que actúan (generalmente) en una sola orientación y (generalmente) están en o cerca del extremo 5' del gen.
- Consisten en sitios de unión para proteínas activadoras específicas. Siempre tienen múltiples sitios de unión, a menudo para varias proteínas activadoras diferentes.
- Conjuntos particulares de genes pueden ser regulados por su necesidad de conjuntos definidos de proteínas activadoras en sus potenciadores.
G. Alargamiento de la transcripción
- La ARN polimerasa debe ser liberada del complejo de iniciación para transcribir el resto del gen. El alargamiento debe ser altamente procesivo, es decir, una vez que la polimerasa comienza la elongación, debe transcribir ese molde hasta el final del gen.
- Los factores requeridos para el inicio no son necesarios (y pueden inhibir) la elongación, y se disocian.

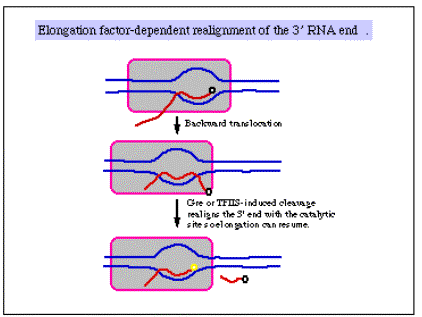
3. Hay algún indicio de que los factores que incrementan la procesividad del complejo de transcripción se unen a la polimerasa alargadora. Entre los ejemplos se incluyen los siguientes.
- NuSA en bacterias
- GreA y GreB en bacterias
- TFIIS en eucariotas, posiblemente muchos otros.

a. Sitios r‑independientes [Nota: r = rho]
- Identificado in vitro
- Horquilla rica en G+C seguida de alrededor de 6 U's
- Se cree que la horquilla es un sitio en el que la ARN polimerasa se detiene, y los débiles pares de bases Ru‑DA en el heterodúplex de ARN‑ADN permiten la fusión del dúplex y la terminación.
- Algunos de los mejores ejemplos de terminadores independientes de r son partes integrales del mecanismo de regulación. Los ejemplos incluyen los atenuadores en el operón trp y otros operones biosintéticos de aminoácidos. Los terminadores independientes de r pueden ser una adaptación especializada para la regulación.
b. Sitios dependientes de r
- Estiramiento rico en C y pobre en G
- Requiere la acción de la proteína r tanto in vitro como in vivo
- Los terminadores dependientes de r se utilizan en los extremos 3' de muchos genes eubacterianos.
2. factor r
- a. Hexamero, cada subunidad 46 kDa
- b. ATPasa dependiente de ARN
- c. El gen para r es esencial para E. coli
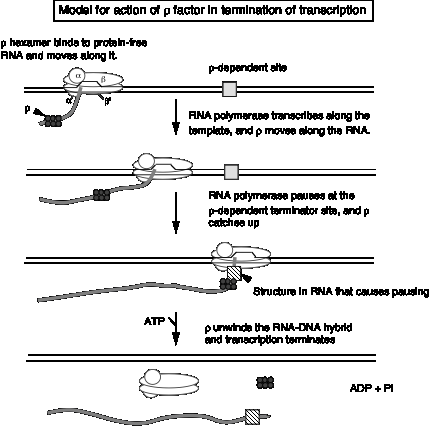
3. Modelo para la acción del factor r
- a. r se une al ARN libre de proteínas y se mueve a lo largo de él
- b. Cuando alcanza una polimerasa en pausa, provoca que la polimerasa se disocie y desenrolla el dúplex ARN‑ADN, con lo que se termina la transcripción. Este último paso utiliza la energía de la hidrólisis de ATP. La proteína r sirve como ATPasa.
Figura 3.2.18.
I. Terminación de la transcripción en eucariotas
1. Terminación por ARN Pol II
- a. No hay evidencia clara de un terminador discreto para la ARN polimerasa II
- b. El extremo 3' del ARNm se genera por escisión y poliadenilación
- c. Señal para escisión y poliadenilación:
(1) AAUAAA, aproximadamente 20 nt antes del extremo 3' del ARNm
(2) Otras secuencias 3' al sitio de escisión
- d. Enzima de escisión no bien caracterizada en este punto; el snRNP U4 puede desempeñar un papel en la escisión. Se ha identificado una poliA polimerasa.
- e. La poliadenilación es necesaria para la terminación por ARN Pol II; posiblemente también haciendo una pausa por la ARN polimerasa
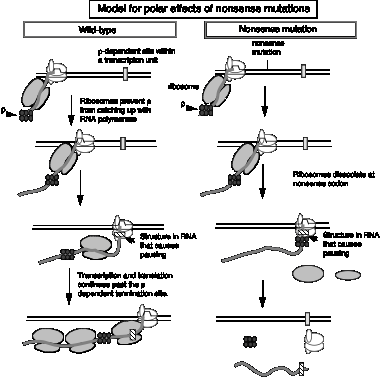
J. Estructura del ARNm en bacterias
1. El ARNm bacteriano suele ser policistrónico.

2. Modelo para r acción puede explicar por qué detener la traducción también puede conducir a un cese de la transcripción.
- a. Supongamos que un terminador de transcripción dependiente de r está presente en el primer gen de un operón. Normalmente no provoca que la transcripción se detenga porque está cubierta por ribosomas que traducen el ARNm, y se transcriben los genes posteriores en el operón. Recordemos que r requiere ARN libre de proteínas para unirse y moverse a lo largo.
- b. Una mutación sin sentido antes del terminador críptico dependiente de r haría que los ribosomas se disociaran, exponiendo ahora el terminador críptico en un tramo de ARN libre de proteínas. El hexámero r puede unirse y moverse a lo largo del ARN, y cuando encuentre una ARN polimerasa estancada, o pausada, en el terminador, provocará que la ARN polimerasa se disocie y se libere el ARN, evitando así la transcripción de los genes posteriores en el operón.
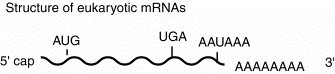
Esta estructura general es cierta para casi todos los ARNm eucariotas. La estructura de la tapa es casi ubicua. Se han encontrado algunos ejemplos de ARNm sin poli A en el extremo 3'. Algunos de los ARNm más abundantes sin poli A codifican las histonas. Sin embargo, la mayoría de los ARNm tienen la cola 3' poli A.
La cola poli A en el extremo 3' se puede usar para purificar ARNm de otros ARN. El ARN total de una célula (que es aproximadamente 90% de ARNr y menos de 10% de ARNm) se puede pasar sobre una columna de oligo (dT) -celulosa. Los ARNm que contienen poli A se unirán, mientras que otros ARN eluirán.


