13: Código genético
- Page ID
- 58550
Una vez completada la transcripción y procesamiento de ARNr, ARNt y ARNsn, los ARN están listos para ser utilizados en la célula, ensamblados en ribosomas o snRNP y utilizados en corte y empalme y síntesis de proteínas. Pero el ARNm maduro aún no es funcional para la célula. Debe traducirse a la proteína codificada. Las reglas para traducir del “lenguaje” de los ácidos nucleicos al de las proteínas es el código genético.
Introducción
Los experimentos que prueban los efectos de las mutaciones de desplazamiento de marco mostraron que la deleción o adición de 1 o 2 nucleótidos causó una pérdida de función, mientras que la deleción o adición de 3 nucleótidos permitió la retención de una función considerable. Esto demostró que la unidad codificante es de 3 nucleótidos. El triplete de nucleótidos que codifica un aminoácido se denomina codón. Cada grupo de tres nucleótidos codifica un aminoácido. Dado que hay 64 combinaciones de 4 nucleótidos tomados de tres a la vez y solo 20 aminoácidos, el código es degenerado (más de un codón por aminoácido, en la mayoría de los casos). La molécula adaptadora para la traducción es ARNt. Un ARNt cargado tiene un aminoácido en un extremo, y en el otro extremo tiene un anticodón para emparejar un codón en el ARNm; es decir, “habla el lenguaje” de los ácidos nucleicos en un extremo y el “lenguaje” de las proteínas en el otro extremo. La maquinaria para sintetizar proteínas bajo la dirección del ARNm molde es el ribosoma.
Figura 3.4.1. Los ARNt sirven como un adaptador para traducir de ácido nucleico a proteína
A. Tamaño de un codón: 3 nucleótidos
1. Tres es el número mínimo de nucleótidos por codón necesario para codificar 20 aminoácidos.
a. 20 aminoácidos están codificados por combinaciones de 4 nucleótidos
b. Si un codón fuera de dos nucleótidos, el conjunto de todas las combinaciones podría codificar solo
4x4 = 16 aminoácidos.
c. Con tres nucleótidos, el conjunto de todas las combinaciones puede codificar
4x4x4 = 64 aminoácidos (es decir, 64 combinaciones diferentes de cuatro nucleótidos tomadas tres a la vez).
2. Los resultados de combinaciones de mutaciones de desplazamiento de marco muestran que el código está en tripletes. Las mutaciones que alteran la longitud que agregan o eliminan uno o dos nucleótidos tienen fenotipo defectuoso grave (cambian el marco de lectura, por lo que se altera toda la secuencia de aminoácidos después de la mutación). Pero los que agregan o eliminan tres nucleótidos tienen poco o ningún efecto. En este último caso, se mantiene el marco de lectura, con una inserción o deleción de un aminoácido en un sitio. Las combinaciones de tres deleciones (o inserciones) de un solo nucleótido diferentes, cada una de las cuales tiene un fenotipo de pérdida de función individualmente, pueden restaurar la función sustancial de un gen. El marco de lectura de tipo silvestre se restaura después de la 3ra deleción (o inserción).
B. Experimentos para descifrar el código
1. Se han desarrollado varios sistemas libres de células diferentes que catalizan la síntesis de proteínas. Esta capacidad para llevar a cabo la traducción in vitro fue uno de los avances técnicos necesarios para permitir a los investigadores determinar el código genético.
a. Reticulocitos de mamíferos (conejos): ribosomas que producen activamente gran cantidad de globina.
b. Extractos de germen de trigo
c. Extractos bacterianos
2. La capacidad de sintetizar polinucleótidos aleatorios fue otro desarrollo clave para permitir que los experimentos descifraran el código. S. Ochoa aisló la enzima polinucleótido fosforilasa, y demostró que era capaz de unir nucleósidos di fosfatos (NDP) en polímeros de NMP (ARN) en una reacción reversible.
NnDP n+ nPi
La función fisiológica de la polinucleótido fosforilasa es catalizar la reacción inversa, la cual se utiliza en la degradación del ARN. Sin embargo, en un sistema libre de células, la reacción directa es muy útil para hacer polímeros de ARN aleatorios.
3. Los homopolímeros programan la síntesis de homopolipéptidos especficos (Nirenberg y Matthei, 1961).
a. si proporciona únicamente UDP como sustrato para la polinucleótido fosforilasa, el producto será un homopolímero poli (U).
b. La adición de poli (U) a un sistema de traducción in vitro (por ejemplo, lisados de E. coli), da como resultado un polipéptido recién sintetizado que es un polímero de polifenilalanina.
c. Así, UUU codifica Phe.
d. Asimismo, poli (A) síntesis programada de poli-Lys; AAA codifica Lys.
Síntesis programada Poly (C) de Poly‑Pro; CCC codifica Pro.
Síntesis programada Poli (G) de Poly‑Gly; GGG codifica Gly.
4. Uso de copolímeros mixtos
a. Si se mezclan dos NDP en una proporción conocida, la polinucleótido-fosforilasa producirá un copolímero mixto en el que el nucleótido se incorpora a una frecuencia proporcional a su presencia en la mezcla original.
b. Por ejemplo, considere una mezcla 5:1 de A:C. La enzima utilizará ADP 5/6 del tiempo, y CDP 1/6 del tiempo. Un ejemplo de un posible producto es:
AACAAAAACAACAAAAAAAACAAAACAAAACAAAC...
|
Composición |
Número |
Probabilidad |
Frecuencia relativa |
|---|---|---|---|
|
3 A |
1 |
0.578 |
1.0 |
|
2 A, 1 C |
3 |
3 x 0.116 |
3 x 0.20 |
|
1 A, 2 C |
3 |
3 x 0.023 |
3 x 0.04 |
|
3 C |
1 |
0.005 |
0.01 |
c. Entonces, la frecuencia con la que ocurrirá AAA en el co‑polímero es
(5/6) (5/6) (5/6) = 0.578.
Este será el codón que ocurre con mayor frecuencia, y puede normalizarse a 1.0 (0.578/0.578 = 1.0)
d. La frecuencia con la que ocurrirá un codón con 2 A y 1 C es
(5/6) (5/6) (1/6) = 0.116.
Hay tres formas de tener 2 A y 1 C, es decir, AAC, ACA y CAA. Entonces la frecuencia de ocurrencia de todos los codones A2C es de 3 x 0.116.
Normalizando a AAA teniendo una frecuencia relativa de 1.0, la frecuencia de codones A2C es 3 x (0.116/0.578) = 3 x 0.2.
e. Lógica similar muestra que la frecuencia esperada de codones AC2 es 3 x 0.04, y la fequencia esperada de CCC es 0.01.
|
Radioactivo |
Cpm precipitable |
Observado |
Teórico |
|||||
|---|---|---|---|---|---|---|---|---|
|
aminoácido |
- plantilla |
+ plantilla |
incorporación |
incorporación |
||||
|
Lisina |
60 |
4615 |
100.0 |
100 |
||||
|
Treonina |
44 |
1250 |
26.5 |
24 |
||||
|
Asparagina |
47 |
1146 |
24.2 |
20 |
||||
|
Glutamina |
39 |
1117 |
23.7 |
20 |
||||
|
Prolina |
14 |
342 |
7.2 |
4.8 |
||||
|
Histidina |
282 |
576 |
6.5 |
4 |
||||
Estos datos son de Speyer et al. (1963) Cold Spring Harbor Symposium in Quantitative Biology, 28:559. La incorporación teórica es el valor esperado dado el código genético tal como se determinó posteriormente.
f. Cuando esta mezcla de copolímeros mixtos se utiliza para programar la traducción in vitro, la Lys se incorpora con mayor frecuencia, la cual puede expresarse como 100. Esto confirma que AAA codifica a Lys.
g. En relación con la incorporación de Lys como 100, Thr, Asn y Gln se incorporan con valores de 24 a 26, muy cerca de la expectativa de aminoácidos codificados por uno de los codones A2C. Sin embargo, estos datos no muestran cuál de los codones A2C codifica cada aminoácido específico. Ahora sabemos que ACA codifica Thr, AAC codifica Asn y CAA codifica Gln.
h. Pro e His se incorporan con valores de 6 y 7, lo que se aproxima al 4 esperado para los aminoácidos codificados por los codones AC2. Por ejemplo, CCA codifica Pro, CAC codifica His. ACC codifica Thr, pero esta incorporación se ve eclipsada por las “26.5” unidades de incorporación en ACA. O, más exactamente, “26.5” @ 20 (ACA) + 4 (ACC) para Thr.
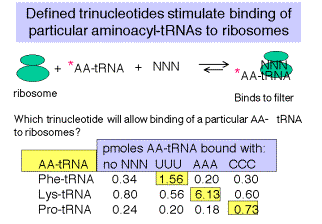
5. Los codones de trinucleótidos definidos estimulan la unión de aminoacil-ARNt a los ribosomas
a. A altas concentraciones de cationes Mg 2+, el mecanismo de initación normal, que requiere f‑Met‑ARNt, puede anularse, y los trinucleótidos definidos pueden usarse para dirigir la unión de aminoacil-ARNt específicos marcados a los ribosomas.
b. Por ejemplo, si los ribosomas se mezclan con UUU y Phe‑Trnaphe radiomarcado, bajo estas condiciones, se formará un complejo ternario que se adherirá a la nitrocelulosa (“ensayo Millipore” que lleva el nombre del fabricante de la nitrocelulosa).
c. Entonces se pueden probar todas las combinaciones posibles de nucleótidos tripletes.
Figura 3.4.2. Datos de Nirenberg y Leder (1964) Ciencia 145:1399.
6. Polinucleótidos sintéticos de secuencia repetitiva (Khorana)
a. Copolímeros alternos: e.g. (UC) n programa la incorporación de Ser y Leu. Entonces UCU y CUC codifican Ser y Leu, pero no pueden decir cuál es cuál. Pero en combinación con otros datos, por ejemplo los copolímeros mixtos aleatorios en la sección 4 anterior, se pueden hacer algunas determinaciones definitivas. Dicho trabajo posterior mostró que UCU codifica Ser y CUC codifica Leu.
b. los programas de poli (AUG) incorporación de Poly‑Met y poli‑ASP a altas concentraciones de Mg. AUG codifica Met, UGA es una parada, por lo que GUA debe codificar Asp.
C. El código genético
Al compilar observaciones de experimentos como los señalados en la sección anterior, se determinó la capacidad de codificación de cada grupo de 3 nucleótidos. Esto se conoce como el código genético. Se resume en el Cuadro 3.4.4. Esto nos dice cómo la célula se traduce del “lenguaje” de los ácidos nucleicos (polímeros de nucleótidos) al de las proteínas (polímeros de aminoácidos).
Cuadro 3.4.4. El Código Genético
Posición en Codón
|
1r |
2do. |
3er |
||||||||||||||
|
U. |
C. |
A. |
G. |
|||||||||||||
|
U |
UUU |
Phe |
UCU |
Ser |
UAU |
Tyr |
UGU |
Cys |
U |
|||||||
|
UUC |
Phe |
UCC |
Ser |
UAC |
Tyr |
UGC |
Cys |
C |
||||||||
|
UUA |
Leu |
UCA |
Ser |
UAA |
Término |
UGA |
Término |
A |
||||||||
|
UUG |
Leu |
UCG |
Ser |
UAG |
Término |
UGG |
Trp |
G |
||||||||
|
C |
CUU |
Leu |
CCU |
Pro |
CAU |
Su |
CGU |
Arg |
U |
|||||||
|
CUC |
Leu |
CCC |
Pro |
CAC |
Su |
CGC |
Arg |
C |
||||||||
|
CUA |
Leu |
CCA |
Pro |
CAA |
Gln |
CGA |
Arg |
A |
||||||||
|
CUG |
Leu |
CCG |
Pro |
CAG |
Gln |
CGG |
Arg |
G |
||||||||
|
A |
AUU |
Ile |
ACU |
Thr |
AAU |
Asn |
AGU |
Ser |
U |
|||||||
|
AUC |
Ile |
ACC |
Thr |
AAC |
Asn |
AGC |
Ser |
C |
||||||||
|
AUA |
Ile |
ACA |
Thr |
AAA |
Lys |
AGA |
Arg |
A |
||||||||
|
AGO* |
Met |
ACG |
Thr |
AAG |
Lys |
AGG |
Arg |
G |
||||||||
|
G |
GUU |
Val |
GCU |
Ala |
GAU |
Asp |
GGU |
Gly |
U |
|||||||
|
GUC |
Val |
GCC |
Ala |
GAC |
Asp |
GGC |
Gly |
C |
||||||||
|
GUA |
Val |
GCA |
Ala |
GAA |
Glu |
GGA |
Gly |
A |
||||||||
|
GUG* |
Val |
GCG |
Ala |
MORDAZA |
Glu |
GGG |
Gly |
G |
||||||||
* A veces se utilizan como codones iniciadores.
2. Del total de 64 codones, 61 codifican aminoácidos y 3 especifican la terminación de la traducción.
3. Degeneración
La degeneración del código genético se refiere al hecho de que la mayoría de los aminoácidos están especificados por más de un codón. Las excepciones son metionina (AUG) y triptófano (UGG). La degeneración se encuentra principalmente en la tercera posición. En consecuencia, las sustituciones de un solo nucleótido en la tercera posición pueden no conducir a un cambio en el aminoácido codificado. Estas se denominan sustituciones de nucleótidos silenciosas o sinónimos y no alteran la proteína codificada. Esto se discute con más detalle a continuación.
El patrón de degeneración permite organizar los codones en "familias" y "parejas”. En 9 grupos de codones, los nucleótidos en las dos primeras posiciones son suficientes para especificar un aminoácido único, y cualquier nucleótido (abreviado N) en la tercera posición codifica ese mismo aminoácido. Estos comprenden 9 “familias” de codones. Un ejemplo es ACN que codifica treonina.
Existen 13 “pares” de codones, en los que los nucleótidos en las dos primeras posiciones son suficientes para especificar dos aminoácidos. Un nucleótido de purina (R) en la tercera posición especifica un aminoácido, mientras que un nucleótido de pirimidina (Y) en la tercera posición especifica el otro aminoácido.
Estos ejemplos se suman a más de 20 (el número de aminoácidos) porque la leucina (codificada por UUR y CUN), la serina (codificada por UCN y AGY) y la arginina (codificada por CGN y AGR) están codificadas tanto por una familia de codones como por un par de codones. Los codones UAR que especificaban la terminación de la traducción se contabilizaron como un par de codones. Los tres codones que codifican isoleucina (AUU, AUC y AUA) están a medio camino entre una familia de codones y un par de codones.
4. Los aminoácidos químicamente similares a menudo tienen codones similares.
5. El codón principal que especifica el inicio de la traducción es AUG
Las bacterias también pueden usar GUG o UUG, y muy raramente AUU y posiblemente CUG. Utilizando datos de los 4288 genes identificados por la secuencia completa del genoma de E. coli, se determinó la siguiente frecuencia de uso de codones en la iniciación:
- AUG se utiliza para 3542 genes.
- GUG se utiliza para 612 genes.
- UUG se utiliza para 130 genes.
- AUU se utiliza para 1 gen.
- Se puede usar CUG para 1 gen.
Independientemente del codón que se use para la iniciación, el primer aminoácido incorporado durante la traducción es f-Met en bacterias.
6. Tres codones especifican terminación de traducción: UAA, UAG, UGA.
De estos tres codones, la UAA se usa con mayor frecuencia en E. coli, seguida de UGA. UAG se usa con mucha menos frecuencia.
- La UAA se utiliza para 2705 genes.
- UGA se utiliza para 1257 genes.
- La UAG se utiliza para 326 genes.
7. El código genético es casi universal
En las raras excepciones a esta regla, las diferencias con respecto al código genético son bastante pequeñas. Por ejemplo, una excepción es el ARN del ADN mitocondrial, donde tanto UGG como UGA codifican Trp.
D. Uso diferencial de codones
1. Varias especies tienen diferentes patrones de uso de codones: Por ejemplo, se puede usar 5' UUA para codificar Leu el 90% del tiempo (determinado por secuencias de nucleótidos de muchos genes). Puede que nunca use CUR, y la combinación de UUG más CUY puede representar el 10% de los codones.
2. La abundancia de ARNt se correlaciona con el uso de codones en ARNm naturales: En este ejemplo, el ARNt con AAU 3' en el anticodón será el más abundante.
3. El patrón de uso de codones puede ser un predictor del nivel de expresión del gen: En general, los genes más altamente expresados tienden a usar codones que se usan frecuentemente en genes en el resto del genoma. Esto ha sido cuantificado como un “índice de adaptación de codones”. Así, al analizar genomas completos, un gen previamente desconocido cuyo perfil de uso de codones coincide con el uso de codones preferido para el organismo puntuaría alto en el índice de adaptación de codones, y se propondría que se trata de un gen altamente expresado. Asimismo, uno con una puntuación baja en el índice puede codificar una proteína de baja abundancia.
La observación de un gen con un patrón de uso de codones que difiere sustancialmente del resto del genoma indica que este gen puede haber ingresado al genoma por transferencia horizontal desde una especie diferente.
4. El uso de codones preferido es una consideración útil en la “genética inversa”: Si conoces incluso una secuencia parcial de aminoácidos para una proteína y quieres aislar el gen para ella, la familia de secuencias de ARNm que pueden codificar esta secuencia de aminoácidos se puede determinar fácilmente. Debido a la degeneración en el código, esta familia de secuencias puede ser muy grande. Dado que probablemente se usarán estas secuencias como sondas de hibridación o como cebadores de PCR, cuanto mayor sea la familia de secuencias posibles, más probable es que se pueda obtener hibridación con una secuencia diana que difiera de la deseada. Así se quiere limitar el número de secuencias posibles, y al hacer referencia a una tabla de preferencias de codones (suponiendo que sean conocidas por el organismo de interés), entonces se pueden usar los codones preferidos en lugar de todos los codones posibles. Esto limita el número de secuencias que uno necesita hacer como sondas o cebadores de hibridación.
E. Oscilación en el anticodón
Esta flexibilidad en la posición de “bamboleo” permite que algunos ARNt se emparejen con dos o tres codones, reduciendo así el número de ARNt requeridos para la traducción. Las siguientes reglas de “bamboleo” significan que los 61 codones (para 20 aminoácidos) pueden ser leídos por tan solo 31 anticodones (o 31 ARNt).
Además de los pares de bases habituales, uno puede tener pares G‑U y I en la primera posición anticodón puede emparejarse con U, C o A (reglas de oscilación).
5' base del anticodón = 3' base del codón =
primera posición en el ARNt tercera posición en el ARNm
C G
A U
U A o G
G C o U
I U, C o A

F. Tipos de mutaciones
Sustituciones de bases
Esto ya se ha cubierto en la Segunda Parte, Reparación del ADN. Solo como recordatorio, hay dos tipos de sustituciones de bases.
- Transiciones: Una purina sustituye a una purina o una pirimidina sustituye a otra pirimidina. La misma clase de nucleótidos permanece. Los ejemplos son A sustituyendo G o C sustituyendo T.
- Transversiones: Una purina sustituye a una pirimidina o una pirimidina sustituye a una purina. Una clase diferente de nucleótido se coloca en el ADN, y la hélice se distorsionará (especialmente con un par de bases purina-purina). Los ejemplos son A sustituyendo T o C, o C sustituyendo A o G.
A lo largo del tiempo evolutivo, la tasa de acumulación de transiciones excede la tasa de acumulación de transversiones.
Efecto de las mutaciones en el ARNm
- Las mutaciones de sentido erróneo provocan la sustitución de un aminoácido. Dependiendo del reemplazo particular, puede tener o no una consecuencia fenotípica detectable. Algunos reemplazos, por ejemplo, una valina para una leucina en una posición que es importante para mantener una hélice a, pueden no causar un cambio detectable en la estructura o función de la proteína. Otros reemplazos, como valina por un glutamato en un sitio que hace que la hemoglobina se polimerice en estado desoxigenado, provocan patología significativa (anemia falciforme en este ejemplo).
- Las mutaciones sin sentido provocan la terminación prematura de la traducción. Se producen cuando una sustitución, inserción o deleción genera un codón de terminación en el ARNm dentro de la región que codifica el polipéptido en el ARNm de tipo silvestre. Casi siempre tienen graves consecuencias fenotípicas.
- Las mutaciones de desplazamiento de marco son inserciones o deleciones que cambian el marco de lectura del ARNm. Casi siempre tienen graves consecuencias fenotípicas.
No todas las subsituciones de bases alteran los aminoácidos codificados
- La sustitución de bases puede conducir a una alteración en la secuencia polipeptídica codificada, en cuyo caso la sustitución se denomina no sinónima o no silenciosa.
- Si la sustitución de bases ocurre en un sitio degenerado en el codón, de manera que el aminoácido codificado no se altera, se denomina sustitución sinónima o silenciosa.
Ejemplo:
A C U ‑> A A U es una sustitución no sinónima que da como resultado Thr → Asn
mientras que,
AC U ‑> AC C es una sustitución sinónima que da como resultado Thr → Thr
- El examen de los patrones de degeneración en el código genético muestra que las sustituciones no sinónimas ocurren principalmente en la primera y segunda posición del codón, mientras que las sustituciones sinónimas ocurren principalmente en la tercera posición. No obstante, existen varias excepciones a esta regla.
- En general, la tasa de fijación de sustituciones sinónimas en una población es significativamente mayor que la tasa de fijación de sustituciones no sinónimas. Este es uno de los argumentos de apoyo más fuertes a favor del modelo de evolución neutra, o deriva evolutiva, como causa principal de las sustituciones observadas en poblaciones naturales.


