19: Regulación transcripcional en eucariotas
- Page ID
- 58752
Promotores
1. Los genes eucariotas difieren en su estado de expresión
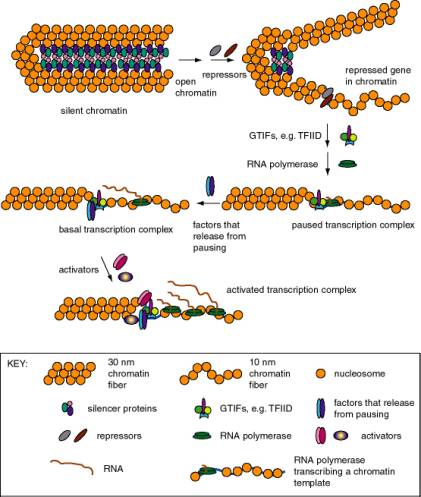
a. Transcripción basal
- Frecuentemente se estudia mediante transcripción in vitro, utilizando moldes definidos y extractos de núcleos o componentes purificados.
- Requiere ARN polimerasa con factores de transcripción generales (por ejemplo, TFIID, TFIIA, TFIIB, TFIIE, TFIIF y TFIIH para la ARN polimerasa II), como se cubrió anteriormente en la Parte Tres.
b. Transcripción activada
- Ocurre a través de activadores transcripcionales que interactúan directa o indirectamente con el complejo de transcripción general para aumentar la eficiencia de la iniciación.
- Los activadores transcripcionales pueden unirse a secuencias de ADN específicas en los elementos promotores aguas arriba, o pueden unirse a potenciadores (véase la Sección B a continuación).
- La idea básica es aumentar la concentración local de los factores de transcripción generales para que el complejo de iniciación pueda ensamblarse más fácilmente. El hecho de que los activadores estén unidos al ADN que está cerca de la diana (o se acerque por el lazo del ADN) significa que la concentración local de esa proteína es alta, y por lo tanto puede impulsar la concentración local de los factores de transcripción generales que interactúan.
3. Polimerasas estancadas
La ARN polimerasa transcribirá aproximadamente de 20 a 40 nucleótidos al inicio de algunos genes y luego se detendrá en un sitio de pausa. El ejemplo clásico son los genes de choque térmico en Drosophila, pero también se conocen otros casos. Estos genes son activados por la liberación de polimerasas estancadas para alargarse. En el caso de los genes de choque térmico, esto requiere factor de transcripción de choque térmico (HSTF). El mecanismo aún está en estudio; algunas ideas interesantes son:
- La fosforilación del CTD de la subunidad grande de la ARN polimerasa II provoca liberación a elongación (“aclaramiento promotor”). Un candidato (pero no el único) para la quinasa CTD es TFIIH.
- Adición de un factor de procesividad (análogo a E. Colinus A?) , tal vez TFIIS.
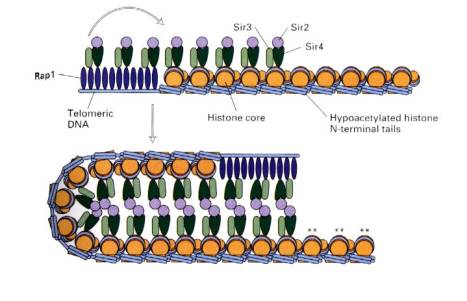
B. Silenciadores
C. Potenciadores
- Los potenciadores son secuencias reguladoras de acción cis que aumentan el nivel de expresión de un gen, pero operan independientemente de la posición y orientación. Estos dos últimos criterios operativos distinguen a los potenciadores de los promotores.
- Ejemplos
a. Región de control SV40
- El SV40 (virus de simios 40) infecta las células de riñón de mono, y también provocará la transformación de las células de roedores. Tiene un genoma de ADN bicatenario de aproximadamente 5 kb. Por su implicación en la tumorigénesis, ha sido un tema favorito de los virólogos moleculares. La región temprana codifica t umor a nti g ens (T-Ag y t-Ag) con muchas funciones, entre ellas estimular la replicación del ADN de SV40 y bloquear la acción de supresores tumorales endógenos como p53 (la “Molécula del Año” de 1993). La región tardía codifica tres proteínas de la cápside llamadas VP1, VP2 y VP3 (v iral p roteína n). Una región entre los genes temprano y tardío controla tanto la replicación como la transcripción de ambas clases de genes.
- La región control tiene un origen de replicación con sitios de unión para T-Ag.
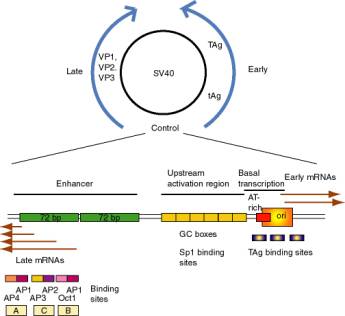
- El SV40 de tipo silvestre expresa T-Ag tras la infección de células de mono y lisa las células infectadas. Sin embargo, una cepa viral que carece de las repeticiones de 72 pb muestra un nivel muy reducido de T-Ag y rara vez lisa las células infectadas.
- Si las repeticiones de 72 pb se vuelven a agregar al genoma mutante de SV40, excepto que se colocan entre los extremos de los genes temprano y tardío (180° desde su posición de tipo salvaje), T-Ag se expresa en un nivel alto y se obtienen infecciones productivas.
- Si se invierte la orientación de las repeticiones de 72 pb, todavía se obtiene un alto nivel de expresión de genes virales e infección productiva. De hecho, es necesario para la expresión de los genes tardíos en el tipo salvaje, los cuales se transcriben en dirección opuesta a los genes tempranos.
- Se concluye que el potenciador es necesario para la transcripción eficiente de los promotores diana, pero puede actuar en cualquier orientación y en una variedad de diferentes posiciones y distancias de las dianas.
- Los trabajos realizados virtualmente concurrentemente con lo descrito anteriormente mostraron que las repeticiones de 72 pb funcionan en otros genes “heterólogos”, de manera que, por ejemplo, los genes de b-globina podrían expresarse en células no eritroides. De hecho esta fue una de las observaciones clave en el descubrimiento del potenciador.
- Una copia de la región de 72 pb funcionará como potenciador, pero dos copias funcionan mejor.
b. Genes de inmunoglobulina
- Este fue el primer potenciador de un gen celular descubierto. Los investigadores señalaron que una región del intrón estaba excepcionalmente bien conservada entre las secuencias de humanos, conejos y ratones, y los experimentos de deleción posteriores mostraron que el potenciador intrónico era necesario para la expresión.
- Después del reordenamiento del gen de inmunoglobulina para fusionar regiones VDJ, uno se deja con un intrón grande entre este gen combinado de región variable y la región constante. Se encuentra un potenciador en ese intrón, y otro potenciador se encuentra 3' del sitio de adición de poliA.
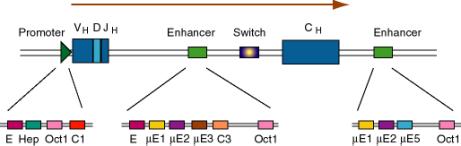
(3) Los potenciadores tienen múltiples sitios de unión para proteínas reguladoras transcripcionales
(a) Varios de estos sitios llevan el nombre del potenciador en el que fueron descubiertos. Por ejemplo, mE1, mE2, etc. son sitios de unión para las proteínas e nhancer identificadas en el gen para la cadena pesada de inmunoglobulina m (mu).
La proteína YY1 (ying yang 1) se une al sitio mE1 (CCAT es el núcleo del consenso) y dobla el ADN allí.
El sitio octámero (ATTTGCAT) está unido por dos proteínas relacionadas. Oct1 se encuentra en todos los tejidos examinados, mientras que Oct2 es específico linfoide, el primer ejemplo de un factor de transcripción específico de tejido. Los activadores transcripcionales que no tienen su propia secuencia de unión al ADN, como VP16 del virus Herpes, se unirán a las proteínas Oct, que se unen al ADN, y el complejo puede activar la transcripción.
(b) Algunas proteínas se unirán a sitios tanto en el promotor como en el potenciador, por ejemplo, proteínas Oct. Recuerda que Oct1 también actúa en el potenciador SV40.
c. Resumen
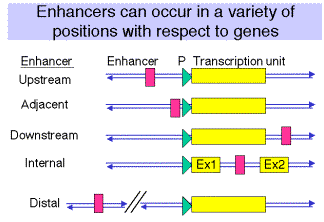
- La posición del potenciador puede estar prácticamente en cualquier lugar con respecto al gen, pero el promotor siempre está en el extremo 5'.
- Se conocen ejemplos de potenciadores 5' al gen (aguas arriba), adyacentes al promotor (como en SV40), aguas abajo del gen (algunos genes de globina), dentro del gen (inmunoglobulinas) o muy aguas arriba dentro de una región de control de locus (genes de globina, ver Capítulo 20.)
3. Múltiples sitios de unión para activadores transcripcionales
a. Todos los potenciadores caracterizados hasta ahora tienen múltiples sitios de unión para proteínas activadoras.
b. Se necesitan múltiplos de sitios de unión para la función del potenciador.
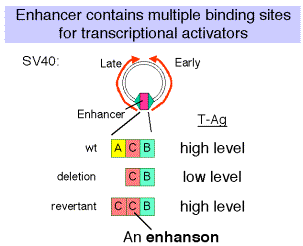
- En experimentos con el potenciador SV40, se observó que algunas mutaciones que disminuyeron la infectividad del virus causaron una mutación de uno de los dominios del potenciador, por ejemplo el dominio A. Cuando estos mutantes se seleccionaron entonces para pseudorevertantes a tipo silvestre, con infectividad restaurada en gran parte, se encontró que los pseudo-revertantes habían duplicado uno de los dominios restantes. Posteriormente, se demostró que los multímeros de los diversos sitios de unión a proteínas eran activos, pero los monómeros tuvieron poca actividad.
- El dominio (por ejemplo, A, C y B en el potenciador de SV40) con al menos dos sitios de unión se denomina enhanson. Múltiples potenciadores conforman un potenciador.
El método de cribado de dos híbridos es una forma rápida y sensible de probar a un gran grupo de proteínas para determinar su capacidad de interactuar in vivo con una proteína en particular. Por ejemplo, se puede caracterizar un componente de un complejo regulador y un ADNc disponible. Este ADNc para la proteína “cebo” se fusiona a segmentos de ADN que codifican un dominio de unión a ADN bien conocido, como el de LexA, que se une a lex o. Cuando se introduce en células de levadura con el gen lacZ (que codifica beta-galactosidasa) bajo el control de lex o, el gen lacZ no se expresa porque la proteína cebo híbrida no tiene dominio de activación. Una biblioteca de ADNc a ensayar se fusiona con el ADN que codifica el dominio de activación de GAL2. Cuando éstas se transforman en células de levadura que portan el cebo híbrido Lexa_DBD-bait y el reportero lex o - lacZ, solo las proteínas híbridas que interactúan con el cebo estimularán la expresión de lacZ. Las células transformadas que son positivas en este ensayo son portadoras de un plásmido con un gen híbrido con el ADNc que codifica una proteína (la “trampa”) que interactúa con la proteína de interés (cebo).
D. Dominios de unión a ADN
Las vistas tridimensionales asistidas por computadora de varios factores de transcripción, que ilustran muchos de los dominios descritos aquí, se pueden ver como tutoriales de Chime en
- www.bmb.psu.edu/pugh/514/mdls
- www.Clunet.edu/biodev/omm/cro/cromast.htm
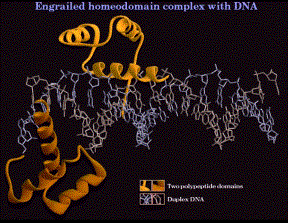
1. Hélice-giro-hélice, homeodominio
- La secuencia del “homeodominio” forma tres hélices separadas por giros apretados.
- La hélice tres ocupa el surco mayor en el sitio de unión en el ADN. Es la hélice de reconocimiento, formando interacciones específicas (enlaces H e interacciones hidrófobas) con los bordes de los pares de bases en el surco mayor.
- Las hélices uno y dos son perpendiculares y superiores a la hélice tres, proporcionando alineación con la cadena principal del fosfodiéster. La cola N-terminal de la hélice interactúa con el surco menor del ADN en la cara opuesta del ADN.
- Helix two + helix three es comparable al motivo hélice-giro-hélice identificado por primera vez en el sistema l Cro y represor.
(5) Ejemplos
a) Los genes homeóticos y sus familiares.
Todos estos están involucrados en la regulación de eventos tempranos del desarrollo en Drosophila. Son factores de transcripción (que regulan los genes que determinan el próximo destino del desarrollo), y tienen este mismo motivo proteico para sus dominios de unión al ADN.
Algunos ejemplos específicos son los productos de estos genes:
- el gen de la regla de pares eve = incluso omitido
- el gen de polaridad del segmento es = engrailed
- el gen homeótico Antp = antennapedia

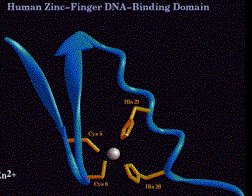
(d) En una proteína con 3 dedos Zn adyacentes, por ejemplo Sp1 (recuerde esta proteína del promotor temprano de SV40), cada dedo se une en el surco mayor para entrar en contacto con tres pares de bases adyacentes. Para el sitio de unión de alta afinidad, un dedo entra en contacto con GGG, el siguiente dedo entra en contacto con GCG y el dedo restante entra en contacto con GGG. Así que los tres dedos se curvan para entrar en contacto con la ranura mayor durante la mayor parte de una vuelta de la hélice.
(e) Los miembros de esta clase de proteínas de dedo Zn tienen múltiples dedos, generalmente en una matriz en tándem. Los ejemplos incluyen TFIIIA (el motivo fue descubierto en esta proteína) con 9 dedos, una proteína de unión a CAC (relacionada en cierta medida con Sp1) con 3 dedos y Drosophila ADR1 con 2 dedos.
(2) Cys2Cys2
a) Secuencia de consenso:
Cys-X2-Cys-X1-3-Cys-X2-Cys
(b) Forma una estructura claramente diferente a la de los dedos Cys2His2 Zn.
- Obsérvese que el número de aminoácidos entre las 2 “mitades” del dedo (1 a 3 en este caso) es mucho menor que los 12 que separan las dos mitades de un dedo Cys2His2 Zn.
- Los dedos Cys2Cys2 no son intercambiables con los dedos de Zn Cys2His2 en experimentos de intercambio de dominios.
- Las proteínas no tienen repeticiones extensas del motivo, en contraste con las proteínas con dedos Cys2His2 Zn.
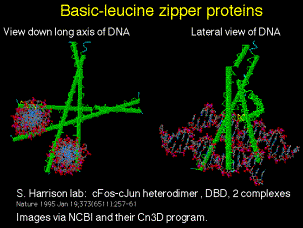
(3) Los ejemplos incluyen heterodímeros que pueden intercambiar parejas
(a) MyoD es una proteína clave en el compromiso de los tejidos mesodérmicos a la diferenciación muscular. Otros parientes, como la miogenina y myf5, son igualmente importantes y proporcionan funciones redundantes. Todos son específicos del músculo y tienen un dominio de unión similar. MyoD es activo cuando tiene E12 o E47 como su compañero heterodimérico; cuando está activo estimulará la transcripción de genes específicos de músculo como el que codifica la creatina quinasa. E12 y E47 se descubrieron inicialmente como proteínas que se unían a potenciadores de genes de inmunoglobulinas, pero que se encuentran en prácticamente todos los tipos de células. Otra proteína, llamada Id, también puede unirse a E12 o E47 por su dominio HLH. Sin embargo, Id carece de un dominio básico, por lo que los heterodímeros con Id no están activos. Por lo que la actividad de las proteínas bHLH puede regularse mediante el intercambio de parejas.
(b) Un tema en desarrollo es que uno de los socios de un heterodímero bHLH es ubicuo (por ejemplo, E12, E47 en mamíferos, da = sin hija en Drosophila) y el otro es específico de tejido (MyoD o AC-S = achaete-scute, un regulador de la neurogénesis en Drosophila). Los componentes ubicuos pueden estar involucrados en la regulación de una variedad de otras proteínas específicas de tejido con dominios bHLH.
(c) Myc, uno de los muchos reguladores del ciclo celular, es una proteína bHLH. Forma socios con Max, y es posible que esto sea importante en la regulación del ciclo celular.
E. Dominios de activación transcripcional
1. Ácidos
Se ha postulado que este dominio es una “mancha ácida” o una hélice anfipática con residuos ácidos en una cara. Estudios físico-químicos recientes de GAL4 han mostrado estructura de lámina b. En este punto no se ha establecido una sola estructura. Ejemplos:
Proteína GAL4, VP16, GCN4, receptor de hormona glucocorticoide, AP1 y el represor l (activación de PRM).
2. Rico en GLN
Este dominio es rico en glutamina, como su nombre lo indica. Ejemplos de proteínas que contienen el dominio son Sp1, Antp, Oct1 y Oct2
3. Pro-ricos
Nuevamente, el dominio es rico en prolina. Los ejemplos incluyen CTF/NF1 (implicado en la regulación de la replicación como n uclear f actor 1, y propuesto como una de las muchas proteínas que se unen a motivos CCAAT).
4. El trabajo hasta el momento no ha establecido estructuras secundarias o terciarias bien definidas para estos dominios.
Una posibilidad es que los dominios de activación asuman su estructura apropiada después de unirse a su diana, es decir, un modelo de ajuste inducido.
Cuadro 4.5.1. Factores de transcripción eucaritoicos seleccionados y sus propiedades
| Nombre | Sistema | Sitio de unión (hebra superior) | Estructura cuaternaria | Dominio de unión a ADN | Dominio de activación | Otros comentarios |
| Engrillado | desarrollo temprano | homeodominio | ||||
| Sp1 | SV40, genes de limpieza celular | GGGGCGGGG | monómero | 3 dedos de Zn Cys2His2 | Rico en GLN | fosfoproteína |
| AP1 | SV40, potenciadores celulares | TGASTCA | heterodímero, Jun-Fos, Jun2, otros | región básica + Cremallera Leu | ácido | regulado por fosforilación |
| Oct1 | linfoide y otros genes | ATTTGCAT | monómero, pero puede unirse a VP16 | POU dominio + homeodominio (HTH) | Rico en GLN, también se une a VP16 | Oct1 es ubicuo, Oct2 es específico linfoide |
| GAL4 | levadura galactosa regulón | CGGASGACWGTCSTCCG | homodímero | Zn2Cys6, cúmulo binuclear | ácido | |
| Receptor de glucocorticoides | genes sensibles a glucocorticoides | TGGTACAAATGTTCT | citoplasma: con proteínas de “choque térmico”; núcleo: homodímero | 2 dedos de Zn, Cys2Cys2 | cerca de dedo Zn | la unión del ligando hormonal cambia la conformación, pasar al núcleo y activar genes |
| MyoD | determinación de la miogénesis | CAGCTG | heterodímero con E12/E47: activo; heterodímero con ID: inactivo | hélice-básica-bucle-hélice | cambiar socios para activar o desactivar | |
| HMG (I) Y | gen del interferón y otros | ranura menor | monómero (?) | dobla el ADN para proporcionar interacciones favorables de otras proteínas | ||
| VP16 | Virus del herpes simple | no se unen firmemente al ADN | se une a proteínas como Oct1 | dominio de activación ácida; muy potente | se une a otras proteínas que por sí mismas se unen específicamente al ADN |
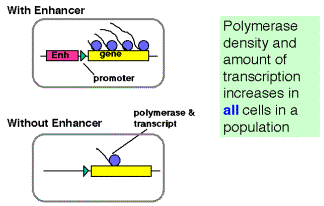
a. En los modelos de bucle, los activadores unidos al potenciador se acercan muy cerca de sus dianas en el promotor mediante la formación de bucles en el ADN.
- Los activadores pueden hacer contacto directo con su diana (quizás el complejo previo a la iniciación), o pueden operar a través de un intermediario llamado coactivador o mediador.
- Si se forma un bucle, en principio no importa cuán grande sea el bucle o si el sitio de unión del activador es 5' o 3' con respecto a la diana. Esto podría explicar la capacidad de los potenciadores para operar independientemente de la posición.
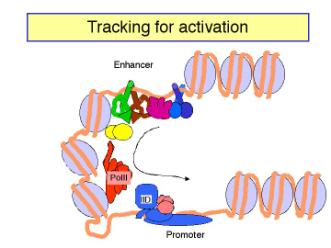
c. El modelo de looping es favorecido en este momento. Sin embargo, ha sido difícil diseñar experimentos que definitivamente descarten el seguimiento. Varias observaciones muestran que el ADN puede formar bucles in vitro, permitiendo el contacto entre las proteínas en el potenciador y las del promotor. Por ejemplo:
- Mediante microscopía electrónica, se pueden visualizar bucles de ADN mantenidos unidos por interacciones entre las proteínas activadoras unidas al potenciador y las proteínas unidas al promotor.
- Los enfoques bioquímicos muestran que los dominios de activación de los factores de transcripción pueden unirse a componentes del complejo previo a la iniciación, como el TFIID (ver Sección H).
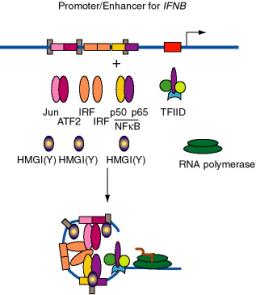
b. Por ejemplo, el potenciador para el gen interferón-b, que se encuentra justo aguas arriba del promotor, tiene sitios de unión para tres factores de transcripción diméricos “convencionales”: NFKB (p50 + p65), IRF y un heterodímero de ATF2 + Jun (un pariente de AP1). Además, existen tres sitios de unión específicos para HMGI (Y).
- HMGI (Y) es un miembro del “grupo de alta movilidad” de proteínas cromosómicas no histonas. La mayoría de las proteínas HMG son abundantes en el núcleo, aunque no tan abundantes como las histonas.
- HMGI (Y) se une en el surco menor del ADN y dobla el ADN.
- También hace contactos específicos proteína-proteína con IRF, ATF2 y NFkB, incluso en ausencia de ADN.
- Al doblar el ADN en posiciones precisas en una cantidad definida, y al facilitar la unión de otras proteínas, HMGI (Y) parece desempeñar un papel crítico en el ensamblaje del complejo potenciador en yuxtaposición con el promotor.
- En general, las proteínas que doblan el ADN pueden ser los agentes que hacen que el bucle acerque las proteínas de unión al potenciador a sus dianas.
c. Otras proteínas que doblan el ADN
Camp-cap (recuerda esto de la represión catabolita en E. coli), IHF = factor huésped de integración (requerido para la integración de l ADN para formar un profago, a través de un gran complejo llamado intasoma), y YY1 (ying yang 1) que tiene efectos negativos o positivos sobre una gran variedad de genes en mamíferos.


